博文
弥合多组学差距
||
弥合多组学差距
就像读取映射工具改变了基因组序列分析一样,将新数据集映射到已建立的参考数据的能力为单细胞基因组学领域提供了一个令人兴奋的机会。作为完全无监督聚类的替代方案,监督映射方法利用大量精心策划的引用来解释和注释查询配置文件。这一策略是通过管理和公开发布参考数据集以及开发新的计算工具实现的,包括统计学习和基于深度学习的方法,这些方法已成功应用于这一目标。
现有方法的局限性是主要关注单细胞RNA测序(scRNA-seq)数据。单细胞转录组学非常适合参考数据集的组装和注释,特别是因为差异表达(DE)基因标记通常可以被解释以帮助注释细胞簇。这导致了高质量、精心策划和专业注释的参考数据的发展,特别是来自包括人类细胞图谱、人类生物分子图谱项目(HuBMAP)和Chan Zuckerberg Biohub联盟在内的参考数据集。映射到这些参考数据集有助于数据的协调、细胞本体和命名方案的标准化以及scRNA-seq数据集在实验条件和疾病状态下的比较。
一个关键的挑战是将参考图谱扩展到其他分子模式,包括染色质可及性的单细胞测量(例如,带测序的转座酶可及染色质的单细胞测定(scATAC-seq))、DNA甲基化(单细胞亚硫酸氢盐测序)、组蛋白修饰(靶向和标记下的单细胞切割(scCUT&Tag))和蛋白质水平(飞行时间细胞仪(CyTOF18)),每一个都测量与scRNA-seq不同的一组特征。缺乏转录组范围的测量为无监督注释带来了挑战。理想情况下,来自不同模式的数据集可以映射到scRNA-seq参考数据上,确保已建立的细胞标签和本体得到保留。前期研究者们已经绘制了模式数据集的方法,例如,将ATAC-seq信号的基因体总和(或DNA甲基化水平的倒数)作为转录输出的代表。这些做出了严格的生物学假设(例如,可访问的染色质与活性转录有关),但这些假设可能并不总是正确的,特别是在分析细胞转变或发育轨迹时。
在这里,Hao等人介绍了“桥接整合”,它通过利用单独的数据集来整合测量不同模态的单细胞数据集,其中两种模态同时作为分子“桥接”进行测量。多元桥数据集可以由一组不同的技术生成,有助于在不同的测量之间转换信息,从而在不需要任何限制性生物学假设的情况下实现稳健的集成。作者们说明了方法的广泛适用性,证明了其在五种不同分子模式下的性能(图1a)。此外,引入了“原子草图集成”,它结合了字典学习和数据集草图,以提高大规模单细胞分析的计算效率,并能够快速集成跨越数百万个细胞的数十个数据集。桥梁集成如图1b所示。首先,该方法不对模态之间的关系进行假设,因为这些是从多组数据集自动学习的。其次,提出的关键进展是向项目数据集的转换,该数据集描述了由共享原子集表示的不同模态。一旦转化,最终比对步骤与多种单细胞整合技术兼容,包括Harmony、mnnCorrect、Seurat、Scanorama或scVI。在本文,作者们用mnnCorrect算法的实现来执行这一步骤。
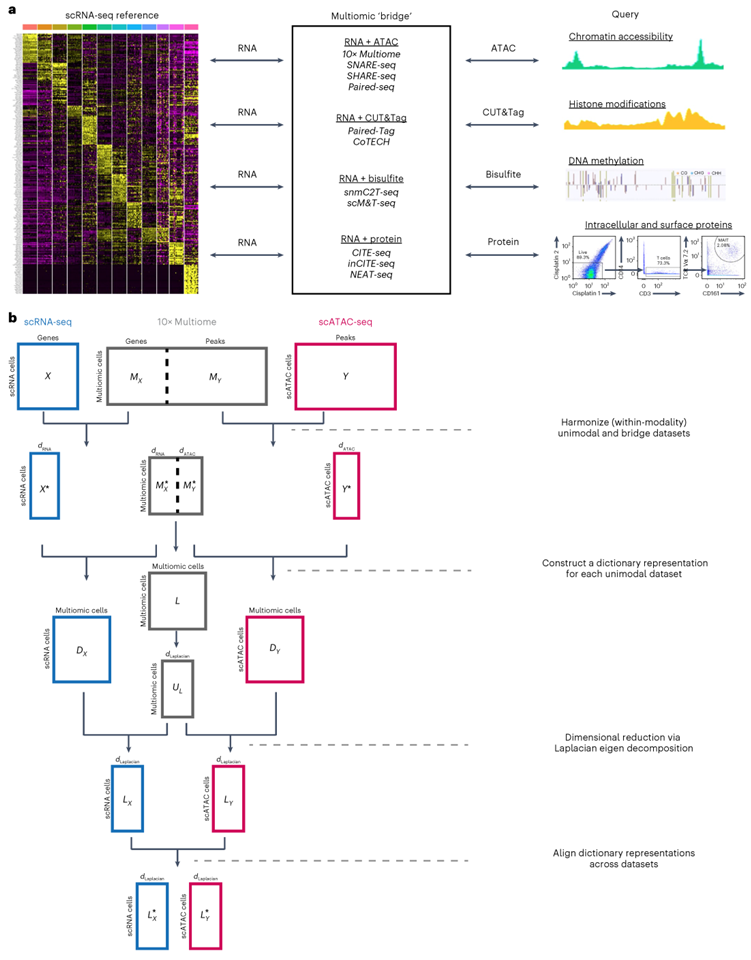
图1 跨模态与分子桥的整合。a、 桥梁集成工作流程的大致示意图。测量不同模态的两个数据集(例如,scRNA-seq和scATAC-seq)可以通过同时测量两种模态的第三个数据集进行协调(例如,10x多组)。使用各种可用作桥梁的多组技术来证明桥梁整合,包括10x多组、配对标签、snmC2T和CITE-seq,每种技术都有助于与不同分子模式的整合。中间框列出了可用于生成桥接数据集的替代多组技术。b、 桥梁整合过程中每个步骤的数学示意图
他们的方法基于字典学习,多组学桥接数据集中的每个细胞构成“字典”中的一个“原子”,用于重建单模态数据集,以产生一组共享的特征,使它们能够相互映射。这种方法的一个重要优点是,它减少了对可能并不总是成立的生物学假设的依赖,例如染色质状态和转录总是相关的。作者们证明了他们的方法在整合转录组数据与染色质可及性、DNA甲基化、组蛋白修饰和蛋白质丰度的独立单细胞测量方面的适用性。例如,将scATAC-seq数据映射到scRNA-seq参考数据上,揭示了罕见的免疫细胞亚群,并鉴定了其表达“滞后”于染色质开放的基因。此外,作者们通过对包括数百个个体和数百万个细胞的scRNA-seq数据集进行大规模整合,证明了他们方法的可扩展性。
总之,这项工作提供了一种强大的方法,通过使用多组学分子桥来整合不同模式的多个单细胞数据集。将整个组学谱中的单细胞图谱结合起来的能力将继续完善我们对健康和疾病中细胞生物学的理解。该方法在Seurat工具包的第5版中实现(http://www.satijalab.org/seurat),旨在拓宽单细胞参考数据集的实用性,并促进不同分子模式之间的比较。
参考文献
[1] Attwaters M. Bridging the multi-omics gap. Nat Rev Genet. 2023 Aug;24(8):488. doi: 10.1038/s41576-023-00632-7.
[2] Hao Y, Stuart T, Kowalski MH, Choudhary S, Hoffman P, Hartman A, Srivastava A, Molla G, Madad S, Fernandez-Granda C, Satija R. Dictionary learning for integrative, multimodal and scalable single-cell analysis. Nat Biotechnol. 2023 May 25. doi: 10.1038/s41587-023-01767-y.
以往推荐如下:
5. EMT标记物数据库:EMTome
8. RNA与疾病关系数据库:RNADisease v4.0
9. RNA修饰关联的读出、擦除、写入蛋白靶标数据库:RM2Target
13. 利用药物转录组图谱探索中药药理活性成分平台:ITCM
19. 基因组、药物基因组和免疫基因组水平基因集癌症分析平台:GSCA
22. 研究资源识别门户:RRID

https://blog.sciencenet.cn/blog-571917-1397817.html
上一篇:单细胞多组学时代的基因调控网络推理
下一篇:TRUST4:从批量和单细胞转录组数据中重建免疫系统