博文
功能宏基因组是什么鬼?干什么用?
|
功能宏基因组是什么鬼?干什么用?
吴崇明 中国医学科学院药用植物研究所 chomingwu@163.com
(综合了4个健康人的功能宏基因组文库终于构建成功了,下一步将着手开展肠道菌群功能基因与人体健康和药物作用的机理性研究工作。整个课题的规模设想很大,欢迎有志于肠道菌群研究并且在微生物培养等方面具有条件和实力的科研同仁加入进来,一起开展一系列有趣的研究,互帮互助,资源和权益共享!)
环境和人体中存在的微生物蕴含着海量的功能基因,可能产生大量结构新颖、功能独特的活性分子。但是这些微生物,特别是海洋和人体微生物,绝大部分尚不能单独培养,因此难以用传统的培养和测序方法对每种细菌的功能、活性和代谢产物进行详细地解析和研究。为了解决这个问题,功能宏基因组(Functional metagenomics)方法应用而生。
所谓功能宏基因组,就是将样品中的全体微生物的DNA提取、剪切和插入到合适的载体上(如Fosmid质粒);然后转化到易于培养的模式细菌(例如E. Coli等)中,构建包含样品中全部微生物DNA信息的宏基因组文库;下一步,对文库菌进行培养和扩大,并利用药理、生化、和分析化学技术对文库菌进行活性筛选,获得阳性克隆菌;通过提取阳性克隆菌的载体并测序,得知赋予阳性克隆菌功能的插入DNA序列;最后通过对插入DNA序列进行解析,与其表现出的功能进行关联,从而获得细菌DNA序列的相应功能,并进行验证。
1. 功能宏基因组学研究历史
2008年,威斯康星大学大学Jo Handelsman教授首次用功能宏基因组学方法鉴定了土壤细菌中能够抵抗β-内酰胺抗生素的基因——β-内酰胺酶,相关结果发表在ISME J上(Allen HK, et al. Functional metagenomics reveals diverse beta-lactamases in a remote Alaskan soil.ISME J. 2009;3(2):243-51.)。同时,Handelsman教授对舞毒蛾幼虫肠道的细菌抗-抗生素谱也进行了分析,这也是将功能宏基因组应用到肠道菌群的第一个实例(Allen HK, et al. Resident microbiota of the gypsy moth midgut harbors antibiotic resistance determinants. DNA Cell Biol. 2009;28(3):109-17.)。随后,多个课题组利用该方法对不同来源的细菌样本进行了抗生素耐受研究,发现了不同种类的对抗不同抗生素的细菌基因。后续的研究中,人们将抗-抗生素基因研究也转向了应对其他胁迫的研究,比如高盐(Culligan EP, et al. Functional metagenomics reveals novel salt tolerance loci from the human gut microbiome.ISME J. 2012 Oct;6(10):1916-25.)(Note: 胁迫有多种,相同的胁迫可以由不同的方法或化合物实现,剖析不同条件下细菌基因的耐胁迫基因,对于应对不同的人体健康危机具有重要参考价值)
除了研究抗生素耐受性基因之外,功能宏基因组学方法也广泛应用于细菌代谢基因的研究。在Handelsman教授发表第一篇功能宏基因组学研究论文一年后,比利时列日大学的Moreno Galleni于2009年9月在ISME J发表论文,报道了利用功能宏基因组学在北极土壤样品中发现能够水解羧甲基纤维素的细菌基因(Berlemont R. et al., Insights into bacterial cellulose biosynthesis by functional metagenomics on Antarctic soil samples. ISME J. 2009;3(9):1070-81.)。2010年,法国Université de Toulouse的Gabrielle Potocki-Veronese将功能宏基因组应用于人体肠道微生物组,并发现了73个能够降解碳水化合物的细菌酶,从而首次对人体肠道微生物组的碳水化合物水解酶信息进行了全景扫描(Tasse L, et al. Functional metagenomics to mine the human gut microbiome for dietary fiber catabolic enzymes.Genome Res. 2010;20(11):1605-12.)。
2010年,法国INRA的Hervé M. Blottière做了一个有趣而大胆的尝试。他们直接用宏基因组文库菌与含有NF-kB报告基因的人细胞进行孵育,观察不同细菌对NF-kB报告基因的影响,从而利用细胞实验筛选宏基因组的功能基因(Lakhdari O, et al.Functional metagenomics: a high throughput screening method to decipher microbiota-driven NF-κB modulation in the human gut. PLoS One. 2010;5(9). pii: e13092.)。这是一个工作量非常巨大的方法,而且直接用细菌孵育细胞存在诸多问题。然而,却给人们提了一个醒——原来功能宏基因组可以这么玩儿!(Note: 相比直接用菌与细胞共孵育,我更推荐用细菌裂解物或者细菌培养外液孵育细胞,以减少菌本身对细胞的危害)
2. 功能宏基因组学的改进
功能宏基因组学提出的前3年(2009-2012)受到了热烈的追捧,也取得了诸多令人振奋的成果。但是很快就陷入了平庸,原因是这个方法非常依赖于宏基因组文库的构建和基因的异源表达,而文库构建和保持过程中不可避免出现基因的丢失,宿主菌也不可能正确表达所有细菌来源的基因片段,所以该方法的产出率并不高。同时,在活性筛选方面大多采用抗生素、环境胁迫和底物代谢的方法进行广泛筛选,这些方法只能反映细菌的很少一部分功能,而大部分与疾病相关的功能则不能体现,因此该方法必须经过大幅改良后,才能继续发挥强大的功效。大量科学家为此付出了艰辛的劳动。
2.1 多宿主表达
2010年,美国洛克菲勒大学的合成生物学顶级科学家Sean F. Brady教授,首次应用功能宏基因组学方法发现新型细菌代谢产物。由于功能宏基因组必须依赖基因的异源表达,而异源表达可能导致许多基因并不能表达或者不能正确表达,导致所得的有用信息受到很大限制。为此,Brady课题组将宏基因组学在6种不同的细菌中进行表达,然后通过显色或抗生素筛选方法对阳性克隆进行筛选,并分离独特的代谢产物(Craig JW, et al. Expanding small-molecule functional metagenomics through parallel screening of broad-host-range cosmid environmental DNA libraries in diverse proteobacteria. Appl Environ Microbiol. 2010;76(5):1633-41.)。这是Brady教授第一次使用功能宏基因组学方法,他后续对此方法进行了改进(Iqbal HA, et al. Natural Product Discovery through Improved Functional Metagenomics in Streptomyces. J Am Chem Soc. 2016 Aug 3;138(30):9341-4.)。
2.2 稳定同位素标记技术(DNA stable-isotope probing (DNA-SIP))
2000年,美国University of Warwick的J. Colin Murrell利用稳定同位素标记技术+16S DNA测序方法鉴定了环境微生物中能够代谢甲醇的细菌(Radajewski S, et al. Stable-isotope probing as a tool in microbial ecology. Nature. 2000 Feb 10;403(6770):646-9.)。2014年,加拿大University of Waterloo的 Charles进一步使用5种底物(glucose, cellobiose, xylose, arabinose, and cellulose)的同位素标记方法分析了土壤中能够代谢这些糖的细菌分类情况(Verastegui Y, et al. Multisubstrate isotope labeling and metagenomic analysis of active soil bacterial communities. MBio. 2014;5(4):e01157-14.)。(Note:这种方法虽然能够快速鉴定能够代谢某些特定底物的细菌,但是要求所用底物必须能够成为DNA的构成原料,而且必须是被细菌大量消耗的底物,对于用量微小的药物或者不容易进入DNA合成的底物,则难以使用这个方法鉴定其代谢菌。)
2.3 微流控芯片技术
针对传统功能筛选方法不能广泛体现细菌宏基因组的生物活性,而直接的活性筛选需要巨大的细胞培养工作,美国University of Cambridge的Florian Hollfelder尝试使用微流控芯片技术解决上述问题(Colin PY, et al. Ultrahigh-throughput discovery of promiscuous enzymes by picodroplet functional metagenomics. Nat Commun. 2015 Dec 7;6:10008.)。
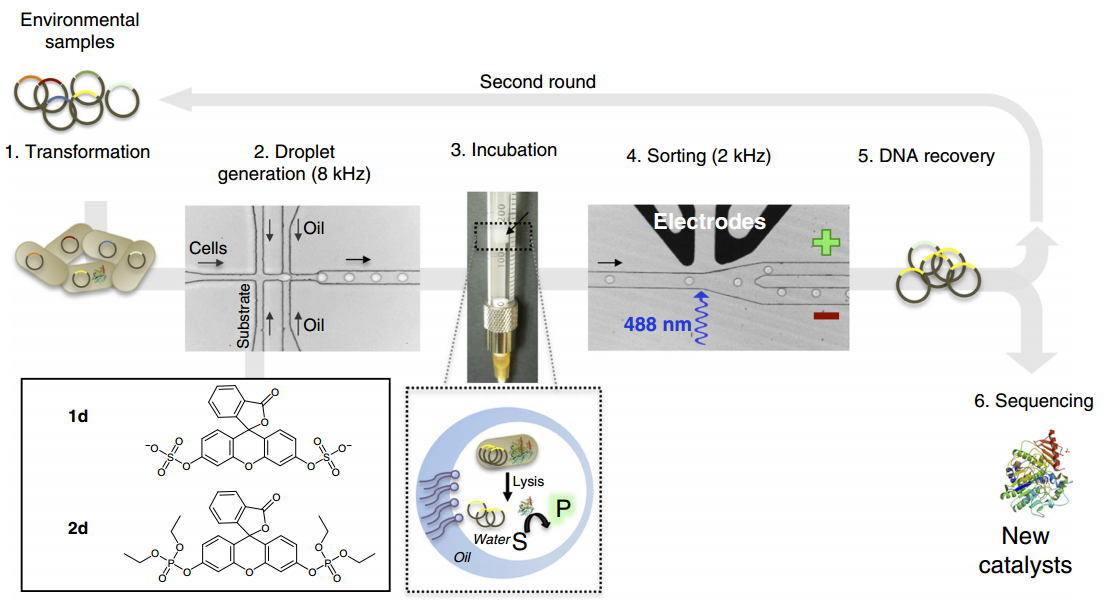
(1)宏基因组文库在E. coli中表达,并将单个细菌包封在一个2 pl的脂滴中;(2)细菌与裂解剂和荧光底物共包封;(3)在合适的培养条件下孵育细菌2天,通过基因活性产生可检测的反应产物;(4)将乳剂在此注射到分选芯片中,对每个脂滴进行分析,利用亮度或荧光强度对脂滴进行分选;(5)将阳性细菌脱乳化,并回收DNA;(6)将DNA转化 E coli进行扩增;(7)在经过第二轮筛选后,对DNA进行测序分析。(Note:这种方法很高效,但是需要专门的设备,技术壁垒很高,需要与有条件的人合作。)
2.4 细胞功能合并化合物分离鉴定
在Blottière的工作基础上,Brady教授利用文库菌的培养液对含有NF-kB报告基因的人细胞进行孵育,在75000个克隆中寻找到了26个潜在可影响NF-kB表达的基因。进一步,他们从阳性菌中获得了能够发挥药理功能的化合物——N-acyl-3-hydroxy palmitoyl glycine (commendamide),其是GPCR的激动剂。(Cohen LJ, et al. Functional metagenomic discovery of bacterial effectors in the human microbiome and isolation of commendamide, a GPCR G2A/132 agonist. Proc Natl Acad Sci U S A. 2015 Sep 1;112(35):E4825-34.)
2.5 功能宏基因组+生物信息学方法
功能宏基因组可用的活性检测方法有限,特别是对于那些目的性明确的研究。为此,将功能基因组学方法和生物信息学方法相结合,可以部分解决这个问题。2015年,德国Lars I. Leichert课题组首次将两种方法结合起来,发现了具有脂酶活性的细菌基因(Masuch T, et al. A combined bioinformatics and functional metagenomics approach to discovering lipolytic biocatalysts. Front Microbiol. 2015 Oct 13;6:1110.)。虽然他们的发现并没有特别重大的医学意义,但是开启了一个研究的新模式。2017年,Brady课题组进一步利用该方法,从人肠道菌群中发现了GPCR调节剂,从而证明了该方法在医学应用中的重大价值(Cohen LJ, et al. Commensal bacteria make GPCR ligands that mimic human signalling molecules. Nature. 2017 Sep 7;549(7670):48-53.)。
3. 功能宏基因组的一般步骤
(1) 建立表达文库;
(2) 对文库菌的表达产物进行分析,发现与对照菌相比有明显新产物的克隆;
(3) 分离该单克隆,并对表达载体进行测序,发现与新产物相关的功能基因;
(4) 同时分离鉴定该未知新产物的结构;
(5) 如果该化合物为新化合物或者生物合成过程未知,则进一步通过生物信息学和分子生物学方法,确定该化合物的生物合成过程;
(6) 与(3)和(4)同时进行菌提取物药理活性研究,若发现该菌提取物有较好活性,则可以快速确定其中的活性化合物。
这样,综合(1)-(6)的结果,将有助于快速发现活性天然产物,确定其生物合成通路,同时实现活性天然产物的大规模绿色生产。从而建立一个海洋药物发现和研究的综合体系。
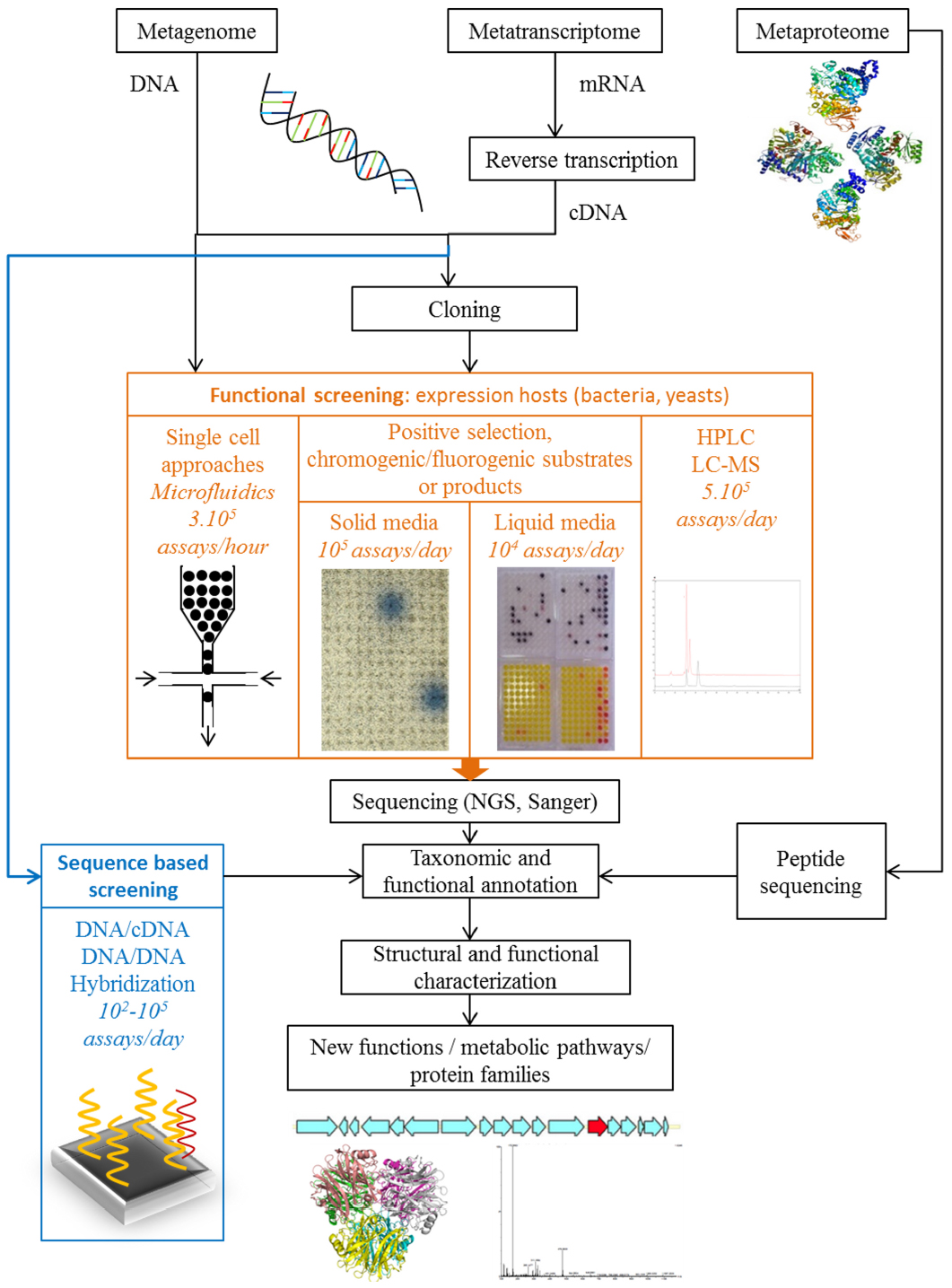
图2. 功能宏基因组操作流程(步骤见前文叙述;图片摘自Ufarté L, et al. Discovery of new protein families and functions: new challenges in functional metagenomics for biotechnologies and microbial ecology. Front Microbiol. 2015 Jun 5;6:563.)
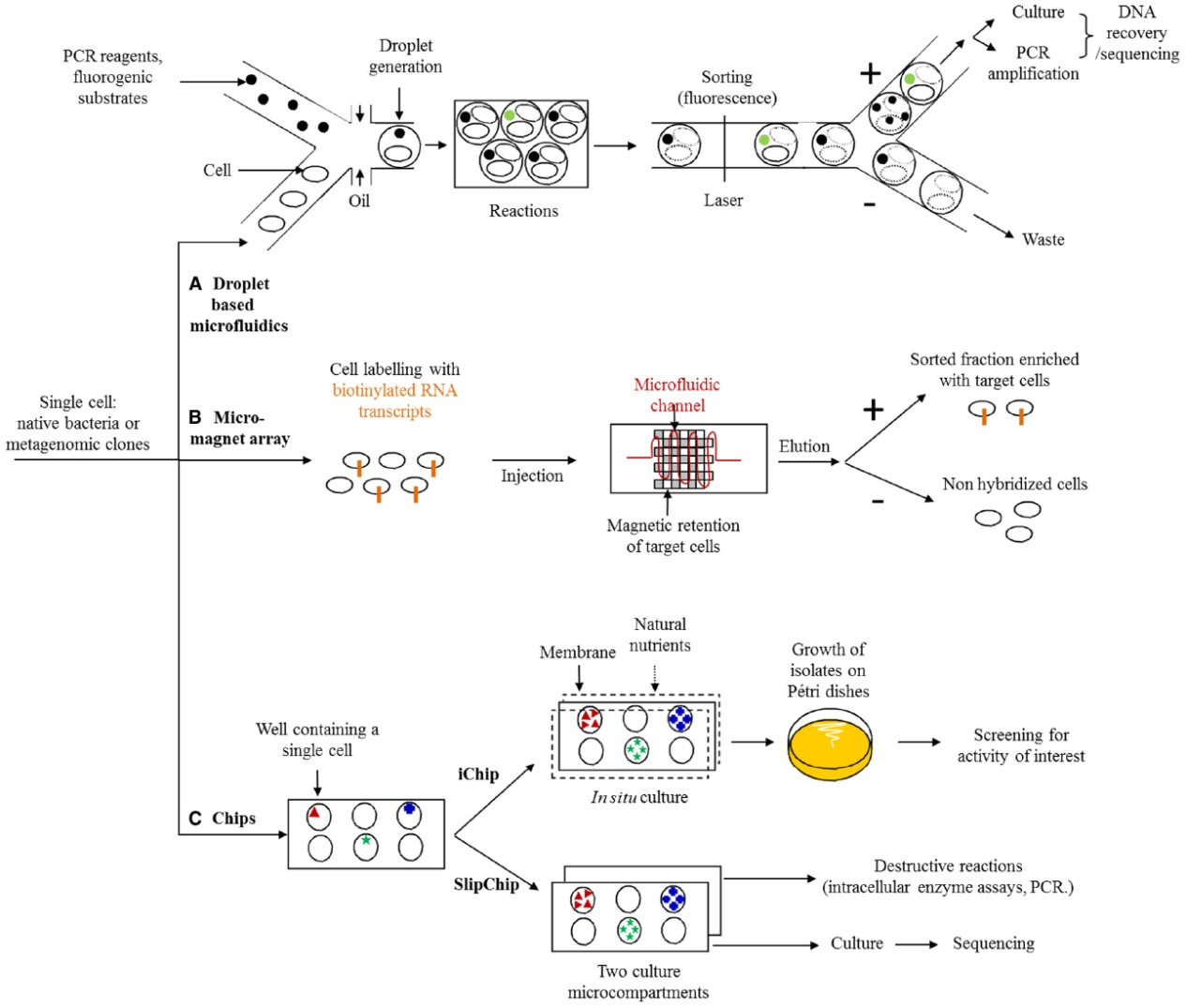
图3. 基于微流控芯片的功能宏基因组研究(步骤见前文叙述;图片摘自Ufarté L, et al. 2015. Front Microbiol)
4. 功能宏基因组应用于新化合物发现和合成生物学研究
传统的通过克隆和异源表达潜在功能基因来获得新型天然产物的方法,受到表达宿主菌不能识别独特的启动子以及对表达产物(转录本和蛋白质)进行正确的剪接和修饰,导致遭遇失败的概率极大,使得整个前期研究工作的努力付之东流。为了深入挖掘海洋微生物资源,我们需要改变研究思路,从“推测生物合成途径——克隆潜在功能基因——在宿主中异源表达——验证活性产物”的传统研究规程变成“建立基因文库——发现产生新产物的克隆——鉴定新型产物和相应功能基因——解析生物合成途径”的功能宏基因组研究策略。
这个策略与传统思路相比的优势在于,我们先行鉴定了有新产物生成,然后解析生物合成通路,也就是先有了最终的结果,从而确保整个研究课题必定是成功的,只不过我们不能预先确定该新产物是否是新型结构分子而已。如果运气好,所得产物是新结构分子的话,那么就可以确保一项重大发现,如果运气不好,所得产物非新结构物质,但通过仔细分析和对比功能基因的来源与结构,仍可望获得有意义的发现,特别是当该产物是目前难以大规模制备的物质时,我们就可能为该化合物的规模化制备建立一个新体系,从而具有了较大的经济价值。
https://blog.sciencenet.cn/blog-3384137-1120334.html
上一篇:原来我们的肠道菌群也可以自己产生药物!问题是怎么发现呢?
下一篇:女性不容易得心脏病——肠道菌群很重要