博文
在线绘制染色体叠加密度和标记图
|
导读:生物信息学数据分析的本质在于解决实际的生物学、医学问题。将诸如基因密度、标记基因等特征在染色体上进行可视化展示,能够帮助我们更直观地将分析结果与生物学意义联系起来。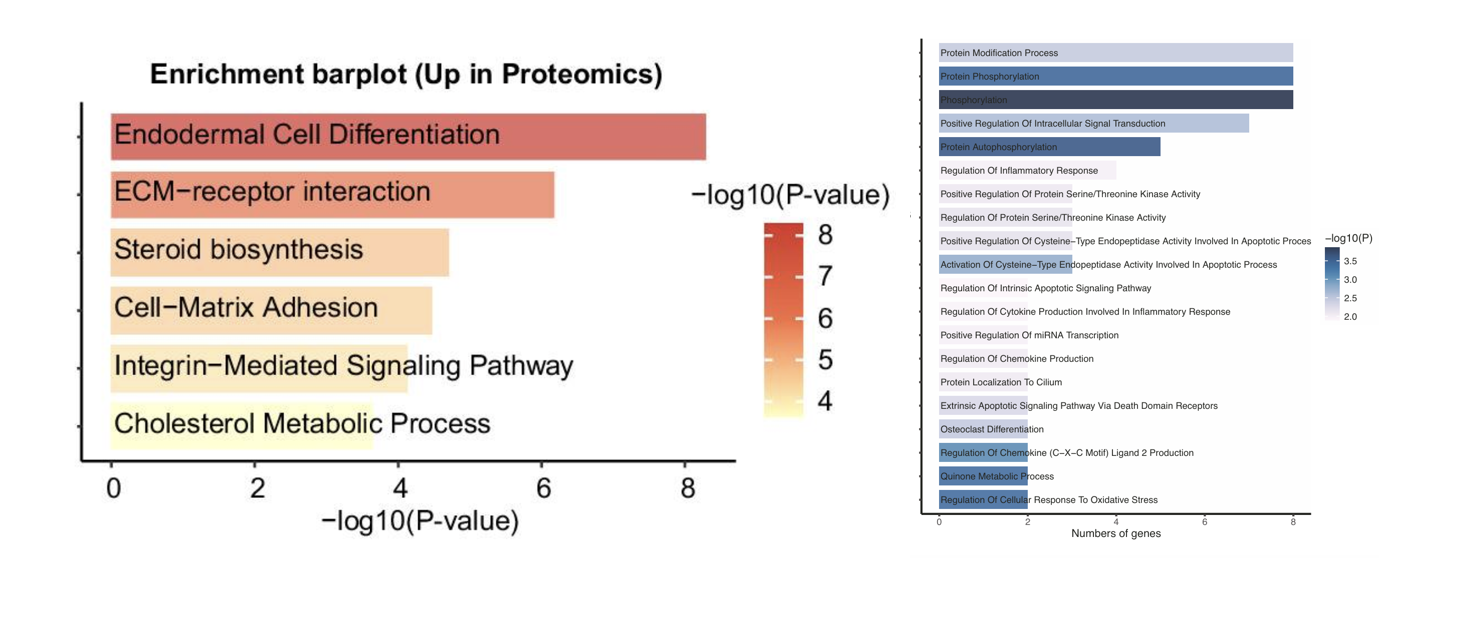 图1.染色体密度图
图1.染色体密度图
《Frontiers in Genetics》文章Optical genome mapping uncovers clinically relevant structural variants in congenital heart disease with heterotaxy. Fig 2D的染色体图展示了77758 个结构变异(Structural Variation,SV)在染色体上的位置分布情况(密度热图)。染色体右侧的正方形代表SV类型,分别表示缺失(deletion),插入(insertion),重复(duplication), 倒位(inversion)和反向重(duplication_inverted)。
染色体顶部对应起始位置,底部对应终止位置,中间的凹陷处表示着丝粒(centromere)。该图常用于ChIP-seq,MeRIP-seq,基因组测序等组学分析中,一般在文章中可以写一小段话进行描述:例如先描述染色体内部:颜色深的地方鉴定到的Peak更多,再描述染色体间:某某染色体鉴定到的Peak比某某染色体更多,再具体描述:某某类型的peak具有什么位置特征。与将数据映射到地图上有异曲同工之妙。
注:Y轴的着丝粒很短,所以图上基本看不到。
1,打开作图URL
https://www.bioinformatics.com.cn/plot_basic_ideogram_heatmap_marker_plot_287
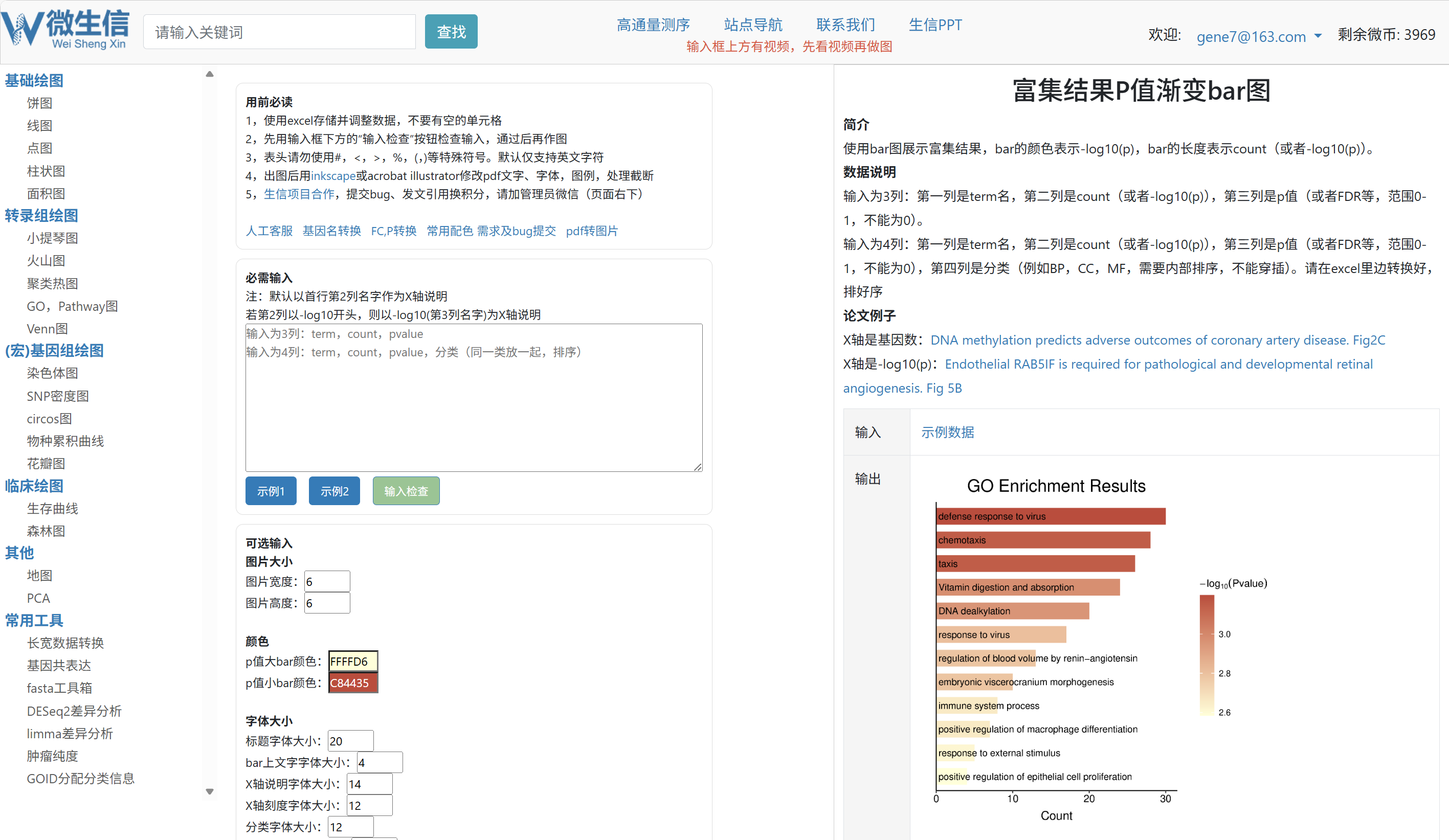
图2.绘图页面
2,示例数据
点击图片上方的示例数据,下载,并使用excel打开。
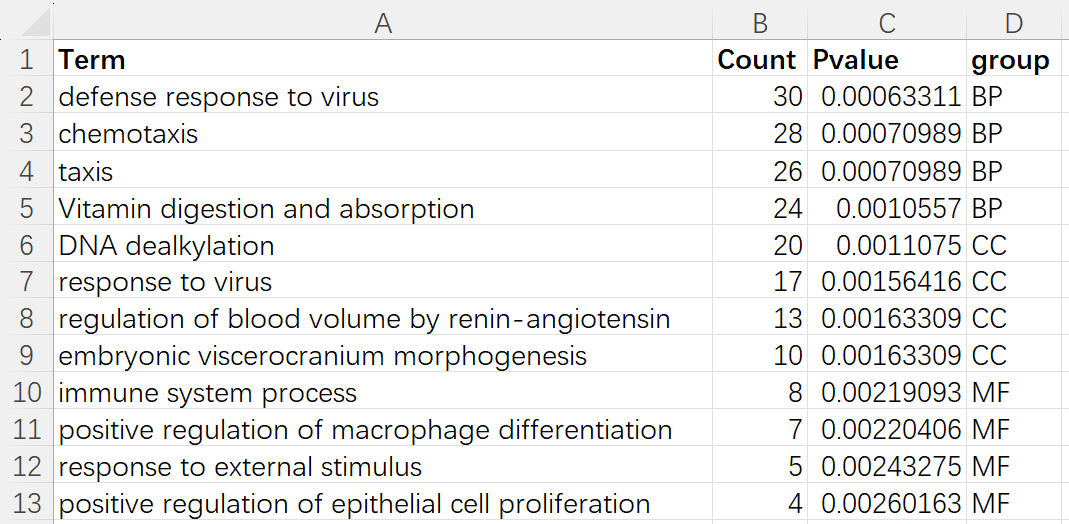
图3. 示例数据
示例数据包括密度数据(染色体密度,必需)和Marker数据(右侧标记,可选)
密度数据(必需)包括四列:第一列是染色体(1-22,X,Y,不带chr),第二列是起始坐标,第三列是终止坐标,第四列是Value值(例如该区域中包含多少基因,Peak的score等)。
Marker数据(可选)也包括四列:第一列是染色体(1-22,X,Y,不带chr),第二列是起始坐标,第三列是终止坐标,第四列是类别(例如不同类型的RNA,不同的调控类型等)。
注:这里仅支持hg38的基因组
3,输入检查
示例数据:点击输入框下面的“示例”按钮,将载入密度数据。
真实数据:数据放在excel中,调整好后,Ctrl+A选中数据,Ctrl+C拷贝,Ctrl+V将数据粘贴到输入框中。
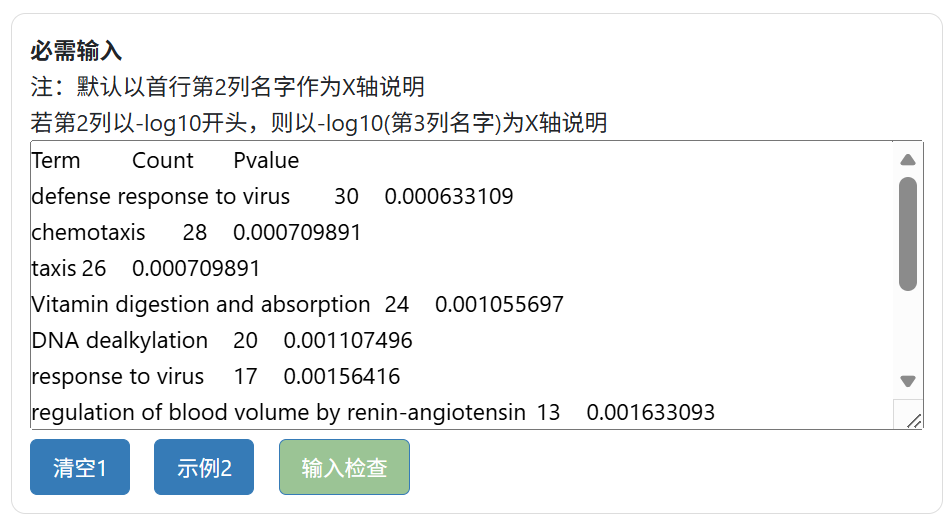
图4. 载入数据,输入检查
然后使用输入框下面的“输入检查”按钮先对输入数据进行检查。若检查不通过,请根据检查提示重复【修改-输入检查】步骤,直到检查通过(如下图所示),然后可以继续选择其他参数。
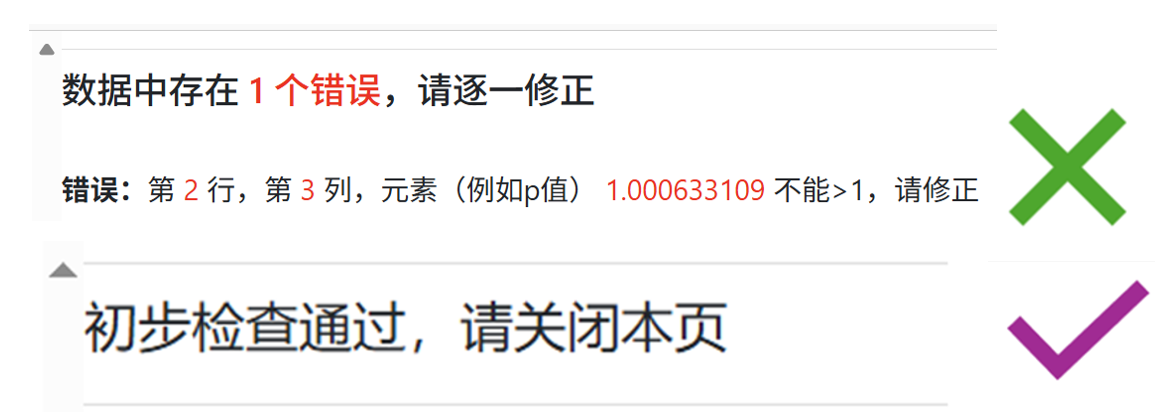
图5.输入检查结果
注:输入检查是新添加的功能,它会根据不同模块的输入要求,逐行逐列检查输入数据,并给出提示,以确保数据符合模块输入要求。
4,选择参数
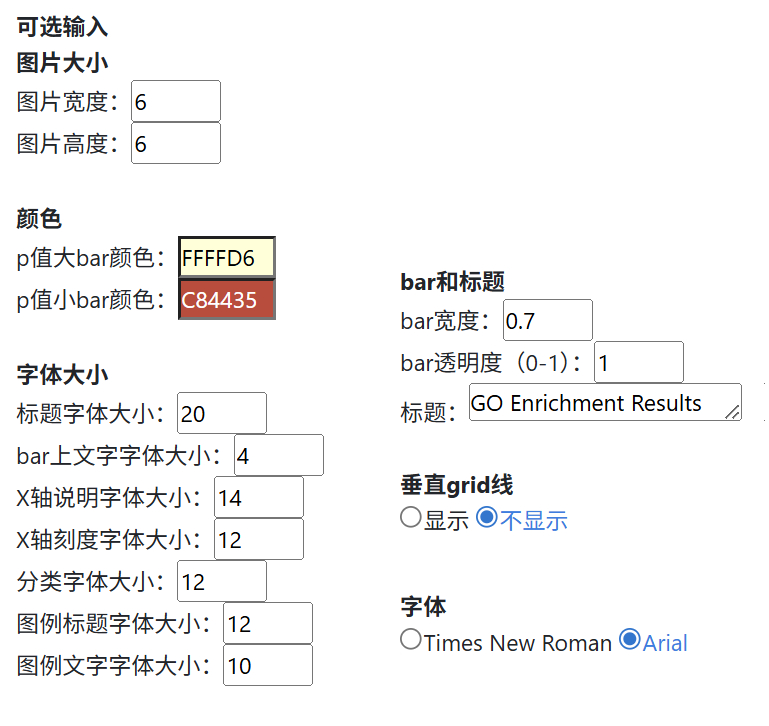
图6.绘图参数
Marker数据
包括四列,可以直接从excel里边拷贝粘贴过来。若不填,则仅绘制密度热图
热图颜色
密度热图的低,中,高颜色,分别对应colorbar的Low,(Middle)和High
Marker形状和颜色
按照marker在marker数据中从上到下出现的顺序,分别设置了Marker的形状和颜色。最多支持6类。
5,提交出图
检查通过,并且参数选好后,点击“提交”按钮,约2s后,会在页面上显示染色体密度图。我们提供了pdf、svg两种矢量图,png、tiff两种标量图供大家下载使用。
图7.预览图
由于该图四周空白较多,请使用acrobat illustrator等软件编辑矢量图(pdf或svg),进行组图,调整文字位置,字体,添加说明等操作,以满足个性化需求。
图8.带marker
代码版:
library(RIdeogram)
data(human_karyotype, package="RIdeogram")
data(Random_RNAs_500, package="RIdeogram")
ideogram(karyotype=human_karyotype, overlaid =gene_density, label=Random_RNAs_500 , label_type="marker", output = "chromosome.svg")
微生信助力高分文章,用户340000+,谷歌学术8500+
https://blog.sciencenet.cn/blog-707141-1517191.html
上一篇:在线绘制Nature Communication同款p值渐变的富集分析结果条形图
下一篇:在线绘制带分组的圆圈热图以展示数值变化