博文
biolord:解码单细胞数据
||
biolord:解码单细胞数据
细胞的基因表达谱同时编码多种属性信息,如细胞类型、组织起源和分化阶段(图1a)。单细胞技术可以在单细胞分辨率下为细胞群体提供有关这种表达谱的信息。然而,解码测量到的基因表达,解开彼此之间的纠缠过程仍然是一个主要的挑战。一个解纠缠的表示可以揭示不同生物过程的存在和特征,允许重建细胞身份的多个属性,如对扰动和感染进展的反应。早期的研究建议使用因子分析或非负矩阵分解来识别与不同属性相关的程序。最近,专门针对特定任务的解纠缠的计算方法被提出,解决的任务包括解耦扰动响应,解耦群体特定属性或单细胞数据的分布外采样。然而,这些方法要么是特定任务的,不能解决一般的解纠缠问题;依赖于线性和独立性假设,不能整合单细胞测量之外的多种类型的信息,或者不能提供通用的重建程序。
在机器学习中,解纠缠方法将世界视为由未知的前向过程生成的,该过程将生成因素(属性)映射到可观察数据中。例如,汽车的图像是由模型和姿态等几个属性生成的。解纠缠的目标是颠倒这个过程,例如,将汽车图像映射到代表其模型和姿态的变量中。然后,解纠缠的表示可以用于数据操作,生成看不见的模型和姿态组合。类似地,在生物环境中,给定标记的单细胞数据,例如,细胞类型和年龄注释(已知属性),解纠缠表示将从未知属性中解耦已知属性,细胞类型和年龄。未知属性对应于细胞特异性特征,例如,与批效应、生物噪声或未分类的生物过程有关。解纠缠表示可用于数据生成、操作和获得生物学洞察力(例如,预测未观察到的细胞类型和年龄组合的测量特征,或识别某些细胞类型或状态的驱动基因)。
利用计算机视觉领域解除纠缠的最新进展,Piran等人提出了biolord(生物表示解除纠缠),这是一个用于学习单细胞数据中解除纠缠表示的深度生成框架。为了将单细胞数据分解为其潜在属性,假设一个由单细胞测量组成的训练集,每个测量都对有限的已知属性集进行部分监督。例如,已知属性可以是细胞类型标签、测量时间或扰动值,属性可以是分类的(离散的;例如,细胞类型)或有序(连续;例如,年龄)。给定部分监督,biolord找到一个解纠缠的潜在空间,由每个已知属性的嵌入和数据中剩余未知属性的嵌入组成(图1b)。在这些之上,biolord学习了一个生成器,它将已知和未知属性的表示映射到可观察的单细胞数据中。反过来,它可以利用解纠缠的潜在空间来预测不同内部或外部条件下不同细胞状态的单细胞测量。通过引入信息约束获得成功的解纠缠,模型的损失函数试图最大化重建的准确性(强制完整性),同时最小化未知属性中编码的信息(限制其容量)。biolord修改了原来的框架,专门用于图像分析,以说明通过架构和设计选择单细胞数据的特征(图1b)。此外,提出了该框架的扩展,生物标记分类,它可以应用于具有部分标记属性的数据集,并为缺失的标签提供分类(图1)。
该框架的通用性使其能够应用于不同的生物环境,可以通过一系列丰富的下游分析任务进行研究(图1c)。利用模型的生成方面,可以进行反事实预测,预测未见的细胞状态并执行数据操作。应用于预测对看不见的药物或基因扰动的反应。分解后的潜空间表示可以独立研究不同的属性及其内部结构。例如,人类胎儿染色质图谱的这种表现揭示了组织之间的关系,样本估计后概念年龄和细胞类型属性。此外,biolord可以将测量的特征与细胞状态联系起来。最后,biolord可以应用于部分标记的数据集,并用于获得整个数据集的标记(属性分类)。biolord应用于时空疟原虫感染图谱,以完成对不同状态的缺失分类(最初仅提供最新时间点),从而使我们能够研究感染状态的瞬态轨迹。作者们使用scvi-tools库实现了biolord(https://github.com/nitzanlab/biolord)。
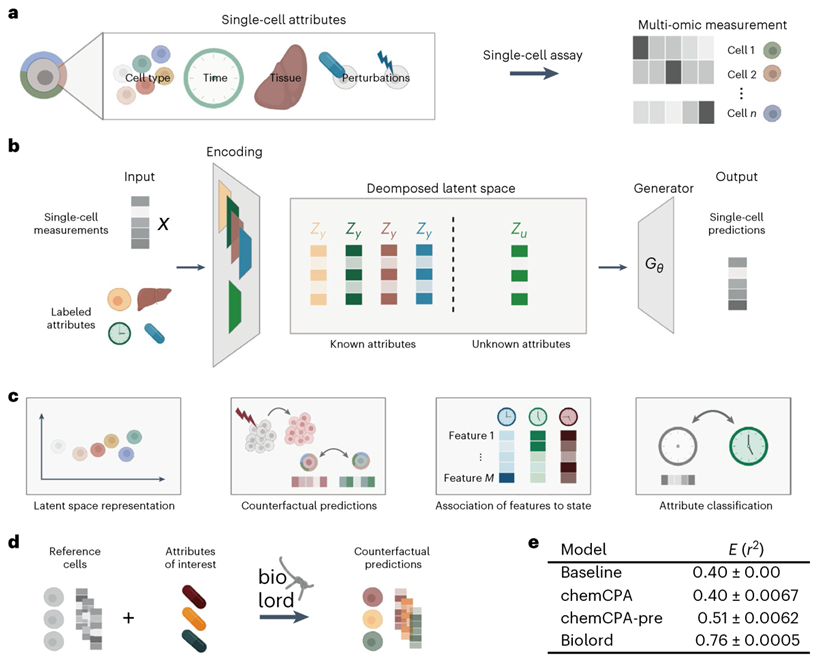
图1 解开已知和未知属性纠缠的生物框架。a、单细胞数据编码细胞身份的多个属性。b、生物模型示意图概述。给定观察属性的单细胞测量值和标签,biolord分别对每个属性进行编码,并对未知属性进行单一编码。这些定义了一个分解的潜在空间,作为提供测量预测的生成模块的输入。c、biolord可用于多个下游任务。从左到右潜空间表示:分解后的潜空间可以用来洞察单个属性的底层结构。 反事实预测:给定一个控制细胞和看不见的(目标)标签作为输入,biolord可以预测看不见的细胞状态的基因表达,并研究基因表达的变化,这些变化与细胞属性的操纵相对应。特征与状态的关联:通过操纵已知属性,biolord可以识别与不同可能状态相关的测量特征,例如,通过操纵控制细胞进入感染状态并识别与感染相关的基因。属性分类:使用半监督生物世界架构,可以用缺失的属性标记细胞。d、获得反事实预测的概要概述。将一组参考细胞的测量值作为输入,这些细胞对进行预测的属性具有不同的赋值。例如,将对照细胞和多种药物作为输入,这些药物可以用于产生反事实预测,即这些细胞的基因表达谱如何在每种药物的作用下发生变化。e、在sci-Plex 3数据集上对biolord在未知药物预测方面的表现进行评估,该数据集包括来自暴露于188种化合物的三种癌细胞系的约65万个单细胞转录组。报告了10μM剂量的结果,这被认为是最严格的设置,因为测量结果与控制状态的偏差最大,因此很难预测。均值和方差报告了每个模型的十种不同的随机种子初始化。图面板a-d是用BioRender.com创建的。
参考文献
[1] Piran Z, Cohen N, Hoshen Y, Nitzan M. Disentanglement of single-cell data with biolord. Nat Biotechnol. 2024 Jan 15. doi: 10.1038/s41587-023-02079-x.
以往推荐如下:
5. EMT标记物数据库:EMTome
8. RNA与疾病关系数据库:RNADisease v4.0
9. RNA修饰关联的读出、擦除、写入蛋白靶标数据库:RM2Target
13. 利用药物转录组图谱探索中药药理活性成分平台:ITCM
19. 基因组、药物基因组和免疫基因组水平基因集癌症分析平台:GSCA
22. 研究资源识别门户:RRID
24. HMDD 4.0:miRNA-疾病实验验证关系数据库
25. LncRNADisease v3.0:lncRNA-疾病关系数据库更新版
26. ncRNADrug:与耐药和药物靶向相关的实验验证和预测ncRNA
28. RMBase v3.0:RNA修饰的景观、机制和功能
29. CancerProteome:破译癌症中蛋白质组景观资源
30. CROST:空间转录组综合数据库

https://blog.sciencenet.cn/blog-571917-1423655.html
上一篇:scPerturb:协调单细胞扰动数据
下一篇:2023年单细胞RNA测序技术展望