博文
Hi-C Insulation score计算原理解析
||
基于insulation score识别TAD边界的算法来自文献[1]:
1) 先将染色体分为10kb的bin,再计算每个bin里的平均互作数,因为每个bin的长度都相等,这里说的平均互作数强调的是ICE校正之后每个bin的互作数。那么后面的分析最小单位就是这里10kb的bin,即分辨率为10kb,具体课题需按基因组大小调整分辨率
2) 以50个bins为一个square,一个square的边长为50*10kb=500kb,将square沿着互作矩阵的主对角线进行滑动,滑动一次为一个bin的长度,并计算square内的互作信号总和。
注意:这里说的沿着主对角线滑动是方框的一个角沿着主对角线滑动,如下图。如果之前有了解Hi-C辅助组装软件3D-DNA的mis-assembly识别算法,就不会对这种处理感到陌生。
这样就算出每个bin的insulation score,然而不同染色体的比对情况和互作情况会有偏差,因此需要做一次normalization
3) Normalization
采用的方法是每个bin的insulation score除以整条染色体上所有bins的insulation score的平均值,再将上述比值取log2对数
4) 染色体上每个bin的normalizated insulation score得到一个向量,其中波谷(或最小值)处表示互作减少,可以认为是TAD的边界。
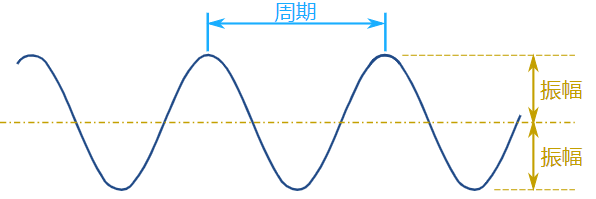
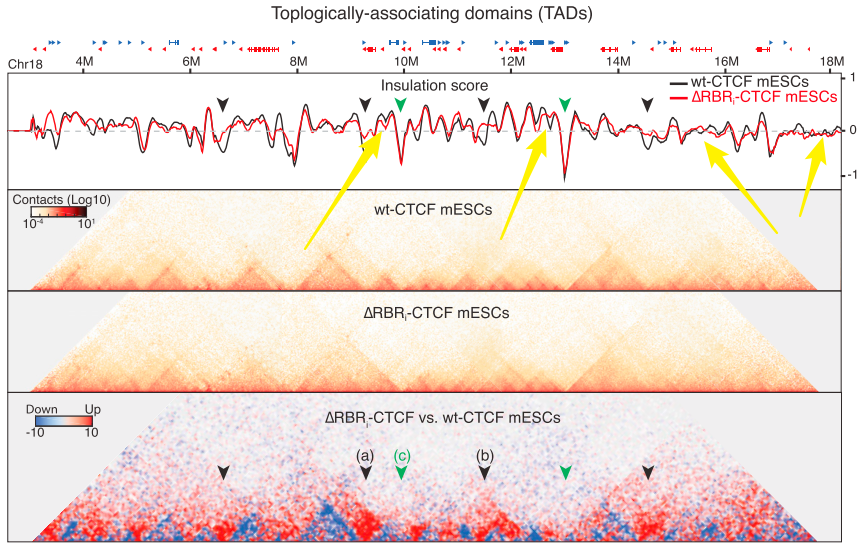
因为insulation score曲线的波动非常大,即使在波峰附近也会出现小区域下凹的情况,如下图黄色箭头标记的位点,如何既考虑凹下去的区域长度,又考虑到凹下去的程度(即如上图中既考虑到周期又考虑到振幅)?仅通过找极低值点,再一刀切(例如小于每于-0.5的波谷判定为boundary),肯定是不行的,以波谷下降程度来算也不行,作者就提出一套称为"delta vector"的方法来做:
"a delta vector was calculated to approximate the slope of the normalized insulation vector."
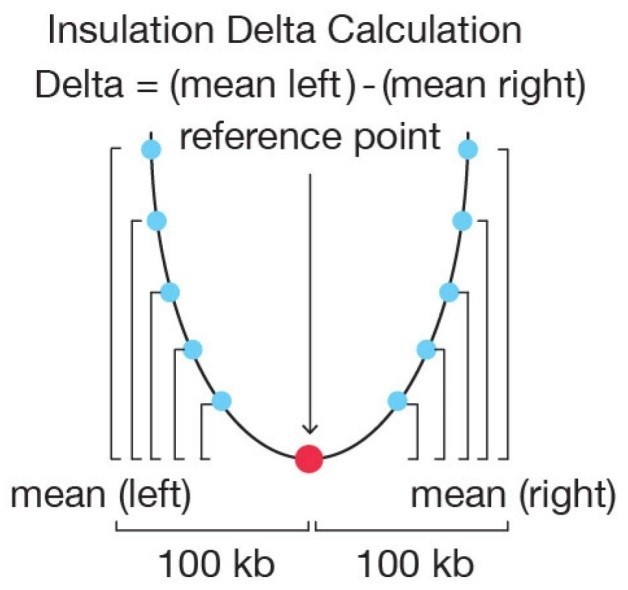
图片修改自文章[2]
5) dalta向量方法
以每个boundary bin(如下图中红点)左侧10个bin(长度100kb)的平均normalized insulation score减去右侧10个bin(长度100kb)的平均normalized insulation score,所得的值即为当前bin(即central bin)的delta值。具体如下图
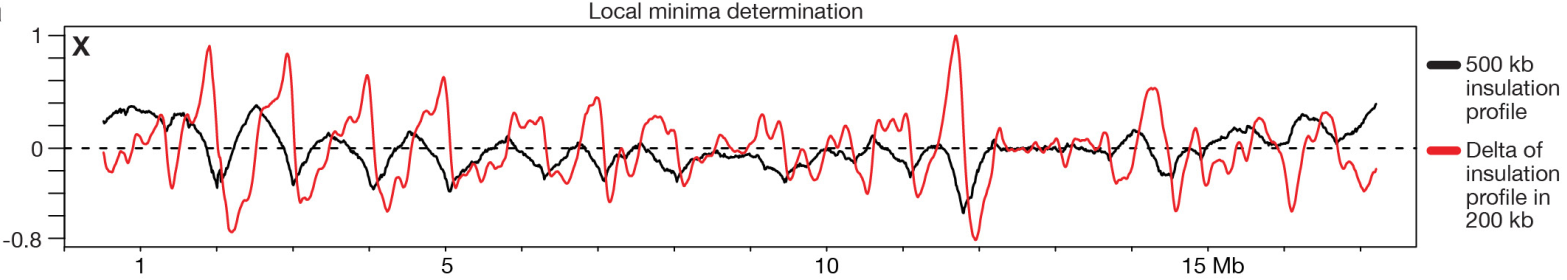
如下图黑色线为insulation score curve,红色的线为delta insulation score curve。很明显dalta为0的bin对应波峰或波谷,因此需去掉波峰,再将波谷对应的位点进行boundary strength过滤。
6) 边界强度
边界强度定义为在delta向量中,boundary bins的从左侧100kb到右侧100kb的局部最大值减最小值 ("The boundary strength was defined as the difference in the delta vector between the local maximum to the left and local minimum to the right of the boundary bin."),白话说就是boundary bin前后100kb范围内,波的变化程度。
去掉boundary strength<0.1的bins,因为这些bins前后100kb变化不大,也即波形不够尖锐。去掉的这些bins附近,要么太平缓,要么波周期太短。
7) 精确度
精确度定义为多个生物学重复识别到的TAD boundary一致性。因为不同生物学重复识别到的TAD boundary不可能在bin分辨率上一致(即可能当中会错开几个bin,比方说生物学重复一的第86-89个bins是boundary,但是生物学重复二的92-94个bins是boundary)。因此在检测准确度时是将多个生物学重复混在一起找最低点的bin,再在该bin的上下游30kb内查找(上游30kb+最低点bin10kb+下游30kb,合计70kb),据作者统计大于80%的生物学重复doundary在这个范围内有overlap。
参考文献:
[1] Emily Crane, Qian Bian, Rachel Patton McCord, et al. Condensin-driven remodelling of X chromosome topology during dosage compensation. 2015. Nature
[2] Anders S. Hansen, Tsung-Han S. Hsieh, Claudia Cattoglio, et al. Distinct Classes of Chromatin Loops Revealed by Deletion of an RNA-Binding Region in CTCF. 2019. Molecular Cell
https://blog.sciencenet.cn/blog-2970729-1375812.html
上一篇:rpy2的安装
下一篇:R boxplot显著性绘图包ggpubr的使用注意点(一)