博文
神经元结构的无偏倚发现——全脑与单细胞比较分析识别调控神经功能的神经元
|
神经元结构的无偏倚发现——全脑与单细胞比较分析识别调控神经功能的神经元
神经元结构由分布在中枢神经系统内、通过突触连接的神经元构成,其协同活动调控着从呼吸、运动到认知等各类神经功能。解析这些神经元结构及其在疾病中的异常变化,是神经科学领域的核心目标。历史上,该领域的研究主要采用还原论框架:基于先验假设,将研究问题转化为对特定神经元亚群的探究。如今,高通量技术(包括啮齿动物模型的全中枢神经系统成像、单细胞转录组检测等)的出现,使得观察和表征整个中枢神经系统中的神经元亚群成为可能。研究规模的扩大进一步推动了比较实验设计的发展,而这类设计可通过计算框架实现分析。这些技术进步预示着一个新时代的到来:在中枢神经系统复杂性掩盖下、与多种神经功能相关的神经元结构,如今可通过无偏倚的方式被发现,并借助基因引导的实验操作进行深入研究。
在过去二十年中,技术方法的进步使得操控和记录哺乳动物特定神经元亚群成为可能[1]。光遗传学、化学遗传学、钙成像以及重组酶系统等技术方法,已将还原论框架确立为神经科学的核心范式。然而,还原论的局限性在于:中枢神经系统的巨大复杂性,使得那些难以捉摸却可能至关重要的神经元结构始终处于隐藏状态。从历史研究来看,无脊椎动物(如黑腹果蝇,*Drosophila melanogaster*)或低等脊椎动物(如斑马鱼,*Danio rerio*)的复杂神经元结构研究更易开展;但在过去五年中,技术进步大幅提升了在哺乳动物(如小家鼠,*Mus musculus*)中发现和解析复杂神经元结构的研究规模与分辨率。其中最令人瞩目的技术进展包括全神经系统成像和大规模单细胞生物学技术的兴起。
神经功能源于分布在整个神经系统中的神经元结构的协同活动。因此,确定这些结构的解剖位置,是理解神经功能背后神经元机制的第一步,这一过程需要借助能够对“与所研究神经功能相关的神经元”进行全中枢神经系统可视化的技术方法。早期在哺乳动物中绘制这类神经元结构的研究,采用的是低通量方法:通过组织切片、标记神经元活动标志物(如cFos蛋白)、逐段成像等步骤,重建神经元及其轴突投射的近似图像。为解决这一局限性,组织透明化技术应运而生——该技术可使啮齿动物的脊髓和脑组织实现光学透明,同时保持整个中枢神经系统的三维细胞完整性[2,3]。这些技术进一步推动了光片显微镜的优化,使其能够对透明化组织样本中嵌套的复杂三维神经元结构进行高分辨率可视化和重建。
与此同时,针对这些大规模数据集的计算分析技术也在同步发展,如今已能实现真正的全脑扰动生物学研究。在标准化行为任务中标记“活动依赖性基因表达”的脑组织库,或通过荧光蛋白标记以揭示神经元连接的脑组织库,均可通过自动分割处理,进而配准到通用坐标框架(如艾伦脑图谱,Allen Brain Atlas)[4]。在这些统一的坐标系统中,可通过体素水平的统计分析确定脑和脊髓中受扰动最显著的区域,从而明确这些区域在调控所研究神经功能中的作用[4,5]。这一研究框架已带来诸多意外发现:例如,近期构建的小鼠全脑功能图谱发现,丘脑剑突核(xiphoid nucleus)是冷暴露条件下调控寻食行为的关键区域——尽管此前该结构从未被认为与进食相关[6];另一项研究构建了小鼠不完全脊髓损伤后步行功能恢复过程中“活跃神经元”的全脑图谱,该图谱指出:外侧下丘脑(通常被认为与动机行为和寻食行为相关,而非步行功能)是解释步行恢复的关键脑区[5]。这一发现进一步推动了外侧下丘脑深部脑刺激疗法的研发,该疗法已被证实可促进不完全脊髓损伤患者的步行功能恢复。
尽管全脑图谱为确定“与特定神经功能相关的神经元亚群”的解剖位置提供了高分辨率工具,但目前这些技术仍无法解析这些亚群的分子身份——因为mRNA转录本的多重标记技术仅能可靠检测有限数量的标志基因。这种解剖学研究在分子分辨率上的局限性,与“神经系统任意给定区域内的神经元亚群均表现出显著转录多样性”这一认知的不断深化形成了鲜明对比[7]。
如今,单细胞和单核转录组学技术已能检测单个神经元所表达的完整转录程序;空间转录组学技术则进一步实现了这些转录程序在神经组织细胞结构中的定位。因此,这些技术提供了必要的分子分辨率,可用于研究“全脑解剖图谱和功能图谱所重点关注的中枢神经系统区域内”神经元亚群的多样性。早期在神经系统中应用这些技术的研究,主要聚焦于两个方向:一是建立神经系统特定区域内神经元亚群的分类体系,二是将转录组特征与预先定义的形态学或电生理学分类进行关联[8,9]。随着单细胞和空间转录组学技术规模的扩大,如今已能够在多种实验条件或疾病模型中,对单个神经元亚群的转录组特征进行大规模检测[10]。由于在所有实验条件下都会同时检测完整的神经元亚群集合,因此无需预先选择特定的神经元亚群。
要从比较单细胞转录组学实验产生的数据中得出生物学推论,需要借助能够解决实验核心生物学问题的分析方法。早期为识别“所研究神经功能背后的神经元亚群”而开展的研究,采用的分析范式主要依赖两种方式:一是将即时早期基因(immediate early genes)的表达作为神经元活动的替代指标;二是通过量化各神经元亚群在不同实验条件下的差异表达基因总数,作为这些神经元转录反应强度的替代指标。然而,人们很快发现,这类量化方法很容易产生假阳性和假阴性结果[11]。这一统计分析挑战推动了更具针对性的计算方法的发展:其中部分方法旨在识别神经元亚群或其他细胞类型相对比例的变化[12];另一种用于识别“与疾病或生物学扰动相关的神经元亚群”的框架,则基于这样一种假设——在单细胞检测的多维空间中,对扰动有反应的神经元亚群比无反应的亚群更易区分。相应地,这种可区分程度可通过“基于全转录组基因表达对特定神经元亚群进行分类的准确率”来量化。这些理念被整合到一个名为“细胞类型优先排序(cell-type prioritization)”的机器学习框架中[9,13]。在小鼠模型中应用该方法的研究发现:一种神经元亚群是瘫痪后步行功能恢复的必要且充分条件[9,14];同时还发现,衰老大脑中下丘脑和脑室周围神经元微环境的神经元存在扰动[15]。尽管该方法功能强大,但其有效性依赖于合理的实验设计——因为某些类型的批次效应(batch effects)和小样本量可能会干扰有效的推论得出。
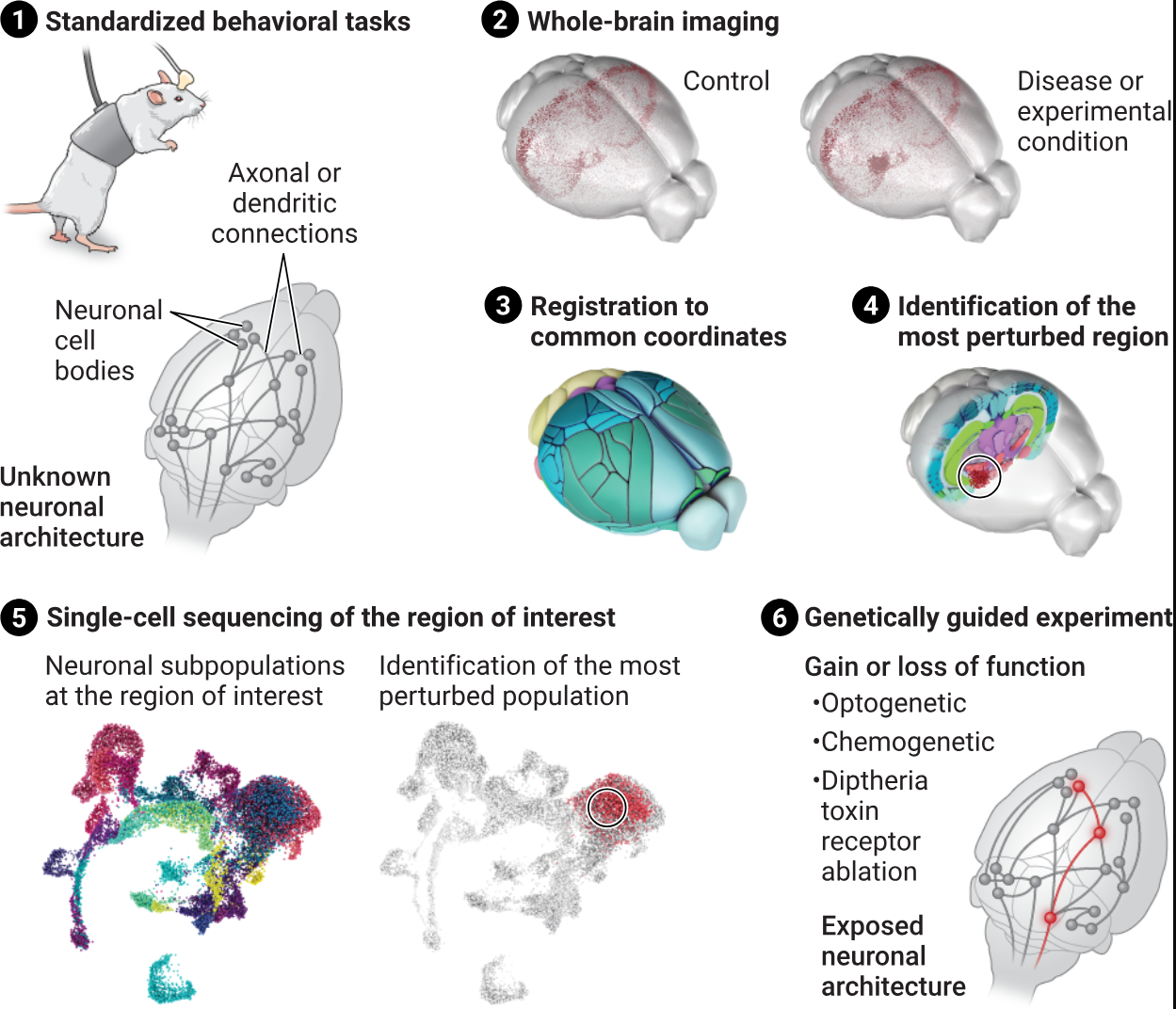
神经元结构识别框架
一套用于无偏倚识别“与行为或疾病相关的神经元结构”的框架,其研究流程始于动物模型中的标准化行为任务(1);通过组织透明化和光片显微镜技术,对全脑中“该任务期间活跃的神经元”的活动标志物进行可视化,从而确定这些神经元的位置(2);将这些数据配准到通用坐标系统,以明确活跃神经元的位置(3、4);在后续实验中,通过单细胞测序表征目标区域内的神经元亚群,并识别受该行为任务扰动最显著的亚群(5);最后,通过针对该亚群的基因修饰,确定其在行为调控中的因果作用,并进一步研究已识别神经元的连接方式和计算特征(6)。
如今,在啮齿动物中开展全脑和单细胞扰动生物学研究的技术方法已日趋成熟,随之而来的问题是:如何发现此前未被识别的、与神经功能相关的神经元结构?应用这些技术方法的首要前提,是建立能够重现“所研究疾病或神经功能”的啮齿动物模型和标准化行为任务。尽管随着转基因模型复杂程度的不断提升,神经疾病或行为相关的动物模型数量已大幅增加,但仍有部分神经疾病和行为难以找到合适的动物模型,或相关模型的适用性存在争议。在建立合适的动物模型和一种(或多种)相关行为任务后,下一步研究需在多个生物学重复和实验条件下,对全脑或脊髓中“被活动依赖性标志物(如cFos)标记的神经元”进行可视化。随后,可将这些脑和脊髓样本库配准到通用坐标系统(如艾伦脑图谱)中,并通过统计和机器学习方法对数据集进行分析,从而确定在特定神经功能或行为调控中起关键作用的解剖区域[4,5]。
接下来的研究步骤包括:在不同实验条件下,通过单细胞和空间转录组学技术对上述关键区域进行分析,结合适当的统计或机器学习方法,揭示“脑或脊髓关键区域内与生物学反应相关的神经元亚群”的身份。之后,可通过基因引导的沉默和激活操作、连接组可视化(包括神经元亚群特异性电子显微镜成像)、神经元亚群特异性钙瞬变或单细胞记录等技术,对这些关键神经元亚群进行研究,以明确“嵌套在神经元结构中的各亚群”的因果作用和计算逻辑。这一方法框架有望揭示啮齿动物模型中复杂神经功能背后的神经元结构(见图)。尽管部分研究步骤在技术上具有挑战性、实验成本高昂,或两者兼具,但组织透明化和成像技术的持续创新,以及单细胞实验成本的不断降低,正为将这一研究策略从啮齿动物推广到更大体型的哺乳动物创造更多可能。
用于加速“与复杂神经疾病和功能相关的神经元结构”发现的技术,目前正快速发展。未来的研究前沿可能在于:最终将全脑成像技术与单细胞及空间转录组学技术整合为单一方法——该方法不仅能在稳态条件下,还能在扰动条件下,检测所有神经元亚型的完整转录组,从而在整个完整的中枢神经系统中发现神经元结构。
https://blog.sciencenet.cn/blog-41174-1509521.html
上一篇:首个慢性疲劳综合征血液检测方法问世
下一篇:氢气和NAC协同抗氧化效应 缓解药物肝损伤