博文
qPCR检测的表观遗传生物钟---相关基因和甲基化位点
||
目前主流表观遗传时钟(epigenetic clock)多采用生物芯片(Infinium MethylationEPIC等)或二代/三代测序技术,在筛选数万至数十万甲基化位点后,精选数百至上千个与衰老及慢性病强相关的CpG位点,建立数学模型预测生物年龄及其他指标。此类方法需要昂贵仪器、专用试剂盒及生物信息学专业团队支持,操作复杂、成本较高,难以普及至普通人群。
相对而言,基于实时荧光定量PCR(qPCR)的简化表观遗传时钟(minimal clocks)只需检测少数(3–10余个)已验证与衰老高度相关的CpG位点,操作简便、成本低廉、结果周期短(通常1–2天),适合临床体检、高通量筛查及大规模人群应用。尽管其精度可能略低于全表观组芯片/测序方法,但大量研究显示其预测误差可控制在可接受范围内(多数模型MAE 3–6年),足以提供有价值的生物年龄参考,并指导抗衰老干预。
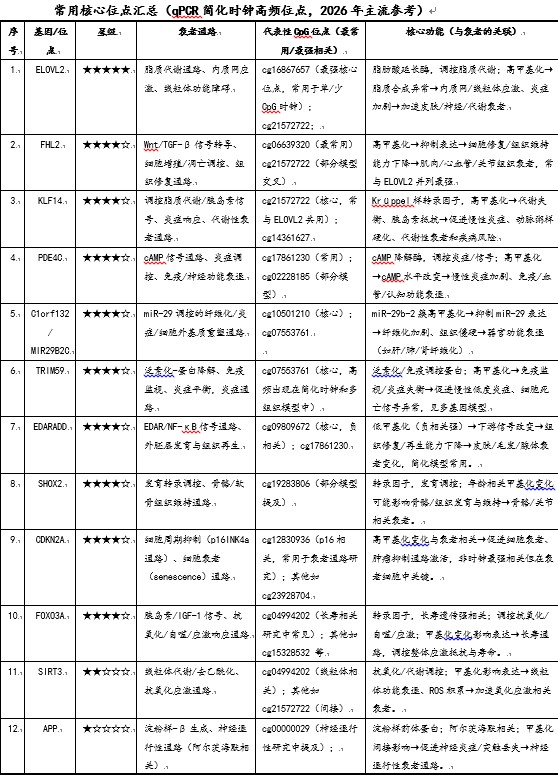
主流qPCR甲基化检测方法
qPCR检测甲基化表观时钟的主要具体方法
( Bisulfite conversion + Methylation-Specific qPCR (MS-qPCR) 或 MethylLight(亚硫酸氢盐转化 + 甲基化特异性定量 PCR(MS-qPCR)或甲基化荧光定量 PCR(MethylLight)
这里简单介绍一下:亚硫酸氢盐转化 + 甲基化特异性定量 PCR(MS-qPCR)或甲基化荧光定量 PCR(MethylLight) 亚硫酸氢盐转化 + 甲基化特异性定量 PCR(MS-qPCR) 是一种经典的、灵敏度较高的DNA甲基化检测方法,常用于检测特定CpG位点的甲基化水平,尤其适合表观遗传时钟中少量核心位点的快速定量(如ELOVL2、FHL2、KLF14等)。
核心原理: 利用亚硫酸氢盐(bisulfite)处理DNA后,未甲基化的胞嘧啶(C)转化为尿嘧啶(U,在PCR中读作T),而甲基化的胞嘧啶(5mC)保持不变,从而产生序列差异。然后设计甲基化特异性引物和探针,通过实时荧光定量PCR(qPCR)选择性地扩增并定量甲基化序列。
主要实验流程
1. DNA提取,提取高质量基因组DNA(通常需要≥50–200 ng)。
2. 亚硫酸氢盐转化(Bisulfite conversion),使用亚硫酸氢钠(或商业试剂盒如EpiTect、EZ DNA Methylation等)处理DNA。化学反应:未甲基化C → 脱氨基 → U(后续PCR读作T);甲基化C(5mC)→ 不发生变化,仍为C;这一步是整个方法的基础,转化效率通常需>98%(否则易假阳性/假阴性);转化后DNA单链化、纯化、回收。
3. 引物与探针设计: 针对目标CpG位点(如ELOVL2的cg16867657),设计两套引物:甲基化特异性引物(M primers):序列完全匹配转化后仍为C的甲基化序列;非甲基化特异性引物(U primers):序列匹配转化后C→T的非甲基化序列。常结合TaqMan探针(荧光标记)进一步提高特异性(称为MethyLight或qMSP)。
4. 实时荧光定量PCR(qPCR):使用M引物+探针对转化后的DNA进行扩增;只有真正甲基化的模板才能被有效扩增(因为U引物序列不匹配);通过Ct值(循环阈值)定量甲基化水平;
常用计算方式:甲基化百分比 = 2^(-ΔCt)×100%(ΔCt = Ct_目标 - Ct_内参);或与已知甲基化标准品比较,得出绝对甲基化比例;
5. 数据分析
以百分比(% methylation)或相对定量形式报告每个CpG位点的甲基化水平。多个位点组合可代入年龄预测模型,计算生物年龄。
主要优点:灵敏度高;可检测低至1–5%的甲基化水平;特异性好:能区分单CpG位点的甲基化状态;通量适中、成本较低:适合检测5–20个核心位点的简化表观遗传时钟;快速:从DNA到结果通常1–2天
局限性: 依赖亚硫酸氢盐转化效率(转化不完全会导致假阳性);引物设计要求高(需避开非特异性扩增);一次只能检测少数几个位点(不适合全基因组分析);对DNA质量和输入量较敏感;
常见应用场景(与表观遗传时钟相关)
简化生物年龄评估(3–10个CpG位点组合);
法医学年龄预测(ELOVL2、FHL2、TRIM59等);
肿瘤标志物检测(启动子区高甲基化基因);
临床研究中验证核心衰老相关CpG位点;
这是目前最成熟的qPCR-based甲基化定量方法之一,许多发表的简化时钟论文都使用过这种技术(或其变体MethyLight)。
https://blog.sciencenet.cn/blog-3302154-1520720.html
上一篇:第四代表观遗传时钟--因果时钟(causal clocks)
下一篇:为什么有些医院要自己开发专属的“病历版ChatGPT”?