博文
出线粒体基因组中对健康和疾病最为关键的位点  精选
精选
|
限制因素揭示出线粒体基因组中对健康和疾病最为关键的位点
线粒体DNA(mtDNA)是存在于线粒体(细胞的“动力工厂”)中的那部分基因组。一项对近6万人的线粒体基因组的分析显示了自然选择是如何从人群中去除有害的基因变异的,进而揭示出哪些线粒体DNA位点对人类健康和疾病最为重要。
推特 脸书 电子邮件
这是对以下文献的总结:莱克(Lake), N. J. 等人所著《量化人类线粒体基因组中的限制因素》,《自然》杂志第635卷,第390 - 397页(2024年)。
问题
通过研究人类基因组,我们能够识别出在健康和疾病中起作用的基因变异。对数千个体基因组进行测序的相关工作已经运用数据驱动的方法来分析所获得的海量数据集。其中一种方法就是识别“受限”的基因组区域[1,2],这些区域是因负向进化选择而基因变异较少的基因组部分,因此最有可能包含与疾病相关的变异。人们会使用一种突变模型来预测在无选择情况下某个基因或区域预期的变异水平,然后将该预测结果与实际观测到的变异情况进行对比,以找出对健康和疾病至关重要的受限位点。
限制因素模型在识别细胞核基因组中作为疾病基础的有害变异方面极为有用。然而,细胞核的限制因素模型无法应用于线粒体中的基因组部分,线粒体是产生化学能并调节诸如新陈代谢和细胞死亡等过程的细胞器。
解决方案
我们旨在通过开发一种限制因素模型来分析线粒体DNA中的变异,以此解决上述问题。第一步是构建一个线粒体突变模型。这颇具挑战性,因为线粒体基因组规模较小,缺乏中性的非编码变异,且其突变机制独特,所有这些因素意味着用于细胞核基因组的突变模型并不适用。因此,我们对先前用于模拟癌细胞突变的复合似然模型进行了调整[3],并将其应用于一组新出现(即非遗传获得)的突变,以此来模拟突变的可能性。
我们进行限制因素分析时面临的另一个难题是,人类细胞包含许多份线粒体基因组拷贝。这使得异质性(即携带某一变异的线粒体DNA拷贝所占的比例)成为衡量选择作用的一个关键指标。因此,我们开发了一种限制因素模型,该模型利用来自gnomAD(一个包含56,434个人的线粒体DNA变异信息的大型数据集[4])的异质性数据来计算观测到的和预期的变异情况。
我们运用自己开发的限制因素模型对gnomAD中的线粒体DNA变异情况进行评估。我们观测到的不影响蛋白质编码序列的变异(即同义变异)数量与预期的大致相同,但已知具有致病性的变异则明显减少,这从而证实了该模型的有效性。将该模型应用于每个基因后发现,许多基因都受到限制,在蛋白质编码基因的非同义变异以及RNA分子的变异方面也观察到了类似的模式。每个基因中受限程度最高的区域富含与疾病相关的变异,而且它们往往会在三维结构中聚集在一起,凸显出对功能重要且与疾病相关的位点。
为每个位点制定一个局部限制分数,这揭示出了整个人类线粒体DNA中受限程度最高的位点(图1)。通过将这些限制指标应用于线粒体疾病患者个体的变异以及英国生物银行数据集[5]中的变异,我们展示了这些指标如何有助于找出作为罕见病和常见病基础的线粒体DNA变异。
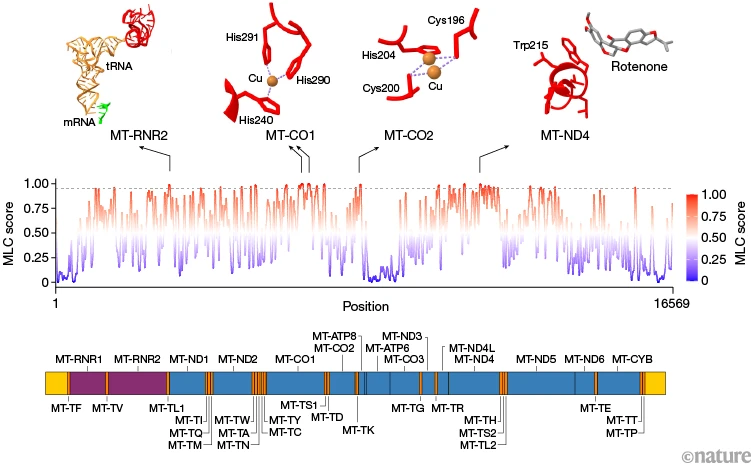
图1 | 人类线粒体基因组中受限程度最高的位点。线粒体DNA局部限制(MLC)分数是一种衡量指标,用于量化线粒体基因组中每个位点的限制程度。上图展示了受限程度最高的位点示例,其线粒体DNA局部限制分数超过0.95(虚线所示):位于线粒体核糖体中转运RNA(tRNA,橙色)和信使RNA(mRNA,绿色)界面处的MT - RNR2,核糖体RNA(rRNA)以红色显示;MT - CO1和MT - CO2中的铜(Cu)结合位点(氨基酸残基以红色显示);以及MT - ND4中的复合物I抑制剂(鱼藤酮)结合位点。下图中,非编码区域以黄色显示,蛋白质编码区域以蓝色显示,核糖体RNA以紫色显示,转运RNA以橙色显示。Cys:半胱氨酸;His:组氨酸;Trp:色氨酸。
未来方向
我们的研究强调了线粒体基因组对人类健康和疾病的重要性。我们发现预期的变异明显减少,这意味着许多有害的线粒体DNA变异仍未被检测到。我们为线粒体基因组中的每个基因和区域制定了限制指标。这些指标可用于识别对人类表型起因果作用的基因变异。
然而,我们的人类线粒体DNA限制因素模型存在一些局限性。例如,该模型主要检测针对异质性位点变异的选择作用,而无法检测针对同质性位点变异的较弱选择作用,而后者也可能与疾病相关。此外,我们仅提供了群体层面的限制因素量化情况,未能涵盖具有不同遗传背景的人群中可能存在的选择差异。
我们计划利用规模更大、更多样化的群体数据集以及改进后的限制因素模型来克服这些局限性。未来的研究还可以对具体功能尚不明确的受限位点进行探究,以巩固我们对线粒体基因组关键位点的理解。——妮科尔·J·莱克(Nicole J. Lake)和蒙科尔·莱克(Monkol Lek)就职于美国康涅狄格州纽黑文市的耶鲁大学医学院。
专家意见
莱克等人利用大型基因组数据库的数据,对人类线粒体DNA的突变限制和选择限制进行了详细的量化分析。这些分析基于核苷酸特异性突变的似然模型的预期值以及来自相应数据库的线粒体DNA变异的观测值,开发了几种量化限制因素的方法。该论文对人类线粒体DNA的选择限制进行了全面分析,对未来与疾病相关的研究分析有着明确的启示意义。——大卫·兰德(David Rand)就职于美国罗得岛州普罗维登斯市的布朗大学。
论文背后的故事
细胞核基因组的限制因素模型和指标在人类遗传学研究中被广泛应用。开展这项工作的一个主要动机源于我们在研究细胞核和线粒体基因组变异时的经验,当时我们意识到限制因素模型对线粒体基因组也会大有帮助。尽管我们对分析线粒体DNA中的限制因素满怀热情,但这并非易事,因为有多个技术障碍需要克服。不过,gnomAD线粒体DNA数据库的出现让解决这一挑战恰逢其时。得益于偶然的交流,以及一支优秀的同事和合作者团队,我们成功建立并验证了一个线粒体DNA限制因素模型。当看到突变模型与gnomAD中观测到的中性变异相关联时,那是一个“灵光乍现”的时刻,因为这意味着我们找到了前进的方向。我们很高兴能为人类遗传学工具包增添一个线粒体基因组限制因素模型及相关指标。——N.J.L.
来自编辑的话
限制因素模型有助于识别那些发生破坏会产生有害影响的基因或基因区域,从而为衡量功能重要性提供一种方法。此类模型已被广泛用于研究细胞核基因和基因组区域,但一直缺少线粒体基因组的限制因素模型。此处所描述的工作填补了这一空白,将有助于更好地理解线粒体基因在人类健康和疾病中所起的作用。——米歇尔·特伦克曼(Michelle Trenkmann),《自然》杂志资深编辑
https://blog.sciencenet.cn/blog-41174-1464806.html
上一篇:如何确保只有死亡的细胞才会破裂
下一篇:皮肤拥有独立的免疫系统