博文
aBIOTECH | 谢卡斌团队揭示ADAR过高表达可在水稻原生质体中造成大量RNA脱靶编辑
||
aBIOTECH | 谢卡斌团队揭示ADAR过高表达可在水稻原生质体中造成大量RNA脱靶编辑

RNA作为遗传信息传递的核心分子,其功能实现离不开RNA结合蛋白(RBP)的调控。RBP是调控基因表达的关键因子之一,RBP通过动态地结合不同基因的转录本参与多种RNA的加工、定位和降解等过程,进而调控植物的各项生命活动。对植物RBP-RNA互作网络的研究能够为解析基因表达调控的分子逻辑、阐明植物生长发育(如器官建成、开花时序)和逆境适应(如抗旱耐盐、抗病防虫)的核心机制提供关键线索。利用RNA靶向编辑技术研究蛋白-RNA互作网络的TRIBE (targeted RNA identification by editing) 方法在动物和植物中得到大量应用。该技术通过将RBP与腺苷脱氨酶ADAR的脱氨酶结构域(ADARdd)融合,利用ADAR能够对RNA进行A-to-I(测序中显示为 A-to-G)编辑的方式标记RBP的RNA靶标。同时ADAR也是靶向RNA编辑的核心元件之一。华中农业大学谢卡斌团队前期借助TRIBE技术,成功鉴定出数百个与水稻双链RNA结合蛋白OsDRB1相互作用的RNA靶标,构建了OsDRB1-RNA的互作网络。TRIBE方法在水稻中表现出良好的实用性和高效性,然而TRIBE技术基于ADAR对RNA的靶向编辑能力,其编辑效率、序列偏好及脱靶风险在植物中尚未有明确报道。
近日,华中农业大学谢卡斌团队在aBIOTECH 发表了题为“High levels of ADAR overexpression induce abundant and stochastic off-target RNA editing in rice protoplasts”的研究论文,该研究针对上述问题提出了指导性建议。
该研究发现在水稻原生质体中过表达ADARdd时会产生大量的脱靶编辑,但在稳定转化ADARdd过表达的水稻叶片中并没有检测到明显的脱靶现象。为了深入探究造成这种现象的原因,研究通过构建不同的ADARdd表达载体并在水稻原生质体中表达,系统探究了ADARdd表达水平、原生质体生理状态、RNA结合结构域等因素对ADARdd脱靶编辑的影响。结果证实,ADARdd脱靶编辑的发生与原生质体的生理状态无关,且引入RNA结合结构域或核定位信号(NLS)也无法降低ADARdd的脱靶活性。真正的关键调控因素是ADARdd的表达水平,研究发现ADARdd表达突破临界阈值(log2[TPM]>13)时,会引发大量随机脱靶编辑。
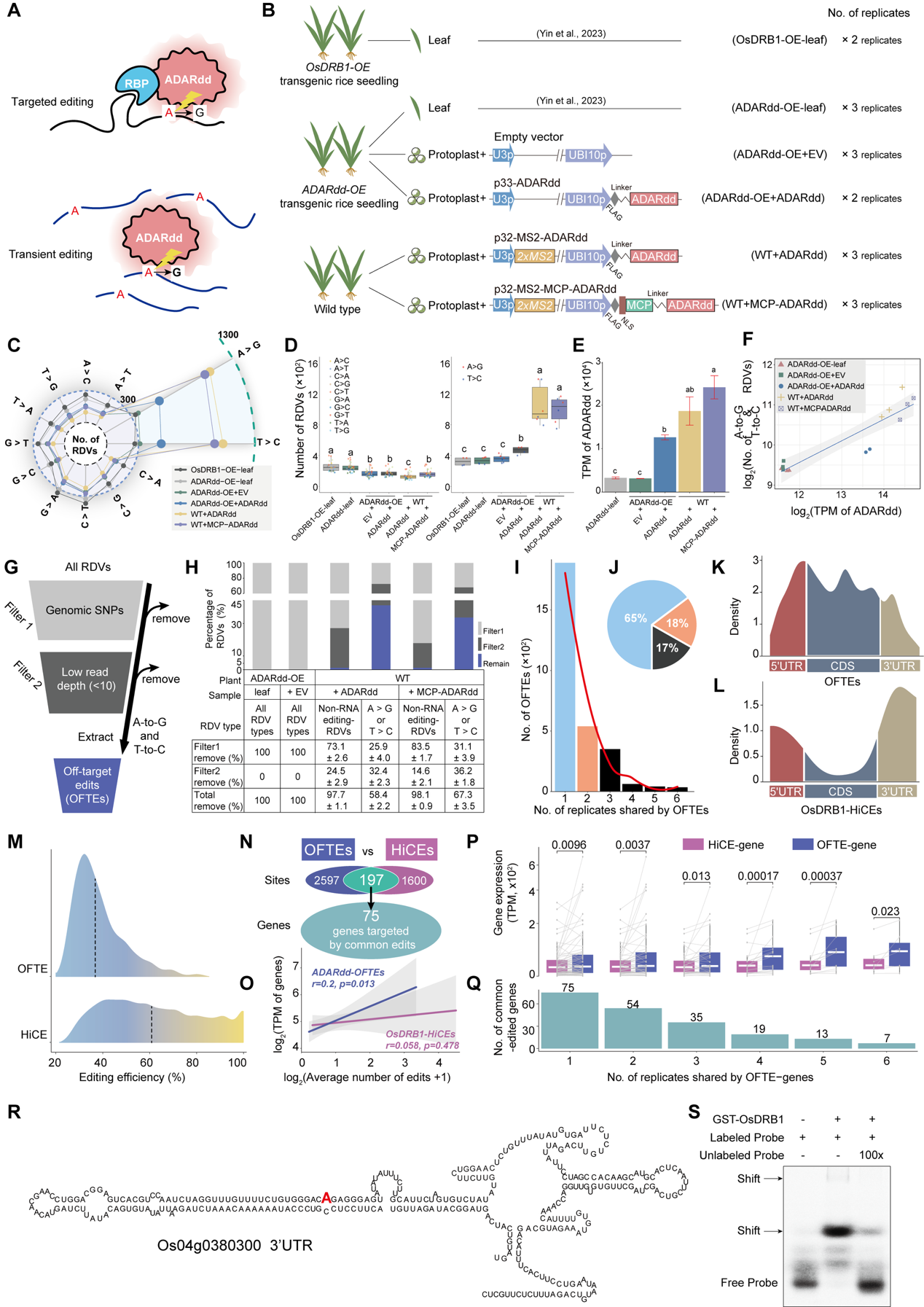
图1. 水稻原生质体中过量表达ADARdd诱导的脱靶RNA编辑 进一步对脱靶编辑的位点进行分析显示,ADARdd介导的脱靶编辑具有明显的“随机性”和“低效率”特征。与团队之前的研究中利用ADARdd在稳定遗传的水稻中对OsDRB1的RNA靶标进行编辑而产生的高度置信的编辑位点(High-confidence editis, HiCEs)相比,ADARdd在原生质体中产生的脱靶编辑位点(Off-target edits, OFTEs)均匀的分布在mRNA上,且编辑效率低于40%;而HiCEs则明显富集于mRNA的非翻译区(UTRs),编辑效率高达40%-100%。此外,对原生质体的转录组数据进一步分析揭示,不同启动子驱动ADARdd表达时,脱靶风险存在显著差异:使用水稻UBIQUITIN10启动子驱动时,稳定转基因植株中ADARdd的表达水平低于临界阈值,脱靶效应极小;而胁迫诱导型启动子(如调控OsSalT、OCPI2基因的启动子)在逆境条件下会到达该阈值,如用于驱动ADARdd也会存在脱靶风险,需在实验设计中谨慎选用(图1)。
本研究得到科技创新2030重大项目、国家自然科学基金等项目的资助。华中农业大学已毕业博士殷帅为第一作者,谢卡斌教授为通讯作者。
引用本文:Yin S, Li S, Yuan Y, Xie K. High levels of ADAR overexpression induce abundant and stochastic off-target RNA editing in rice protoplasts. aBIOTECH 2026;7(1):100007.
https://doi.org/10.1016/j.abiote.2025.100007
https://blog.sciencenet.cn/blog-3458049-1519458.html
上一篇:aBIOTECH直播预告 | 于笑寒—AI for Agriculture:Ultra-Fine-Grained Vis
下一篇:aBIOTECH | 玉米基因编辑技术新突破!赵久然团队创新建立了玉米商业化骨干亲本系统化、工程化国产基因编辑生物育种技
