博文
aBIOTECH | 夏兰琴团队利用水稻烷基嘌呤DNA糖基化酶研发单碱基编辑器rAKBEs并在水稻中实现腺嘌呤单碱基转换
||
aBIOTECH | 夏兰琴团队利用水稻烷基嘌呤DNA糖基化酶研发单碱基编辑器rAKBEs并在水稻中实现腺嘌呤单碱基转换和颠换 
近日,中国农业科学院作物科学研究所作物精准育种技术创新团队在aBIOTECH上发表了题为“Fusion of a rice endogenous N-methylpurine DNA glycosylase to a plant adenine base transition editor ABE8e enables A-to-K base editing in rice plants”的研究论文。
类似于人和小鼠来源的MPG,在水稻中同样存在着MPG同源蛋白,作者分离鉴定了水稻来源的MPG(OsMPG),其与人源MPG核苷酸序列相似性为43.19%,氨基酸序列相似性为27.36%,同源性较低。通过将OsMPG或其突变体mOsMPG与TadA8e-nCas9(D10A)融合,在植物中分别建立第一代和第二代A-to-K单碱基编辑系统rABKE01和rABKE02。在此基础上,进一步将rAKBE01和rAKBE02与反式激活因子VP64融合,从而获得了第三代和第四代A-to-K单碱基编辑系统rAKBE03和rAKBE04(图1)。
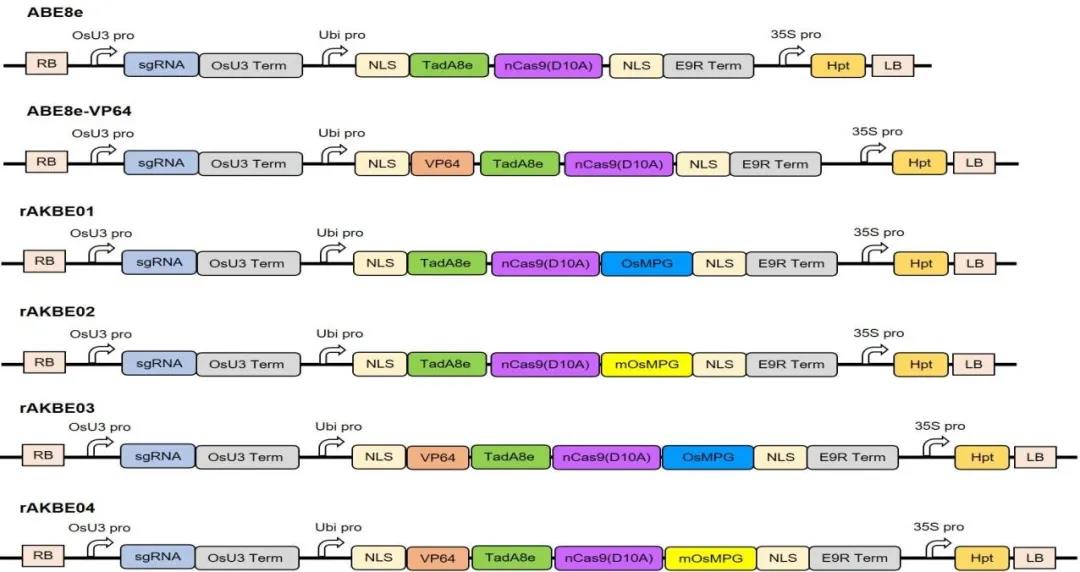
图1 植物rAKBEs载体结构示意图
水稻原生质体中rAKBEs编辑系统介导的碱基编辑活性测定结果表明,rAKBE01编辑效率最低,仅能在OsNRT1.1B位点实现A-to-C/T 的碱基编辑;rAKBE04编辑效率最高,且与rAKBE02和 rAKBE03相比,rAKBE04将A-to-C/T碱基编辑效率分别提高了6.57倍和1.75倍(图2)。此外,由于OsMPG参与碱基切除修复(BER)途径,rAKBE03和rAKBE04在靶位点位置产生了随机indel,效率约为0.39%-1.09%(图2)。
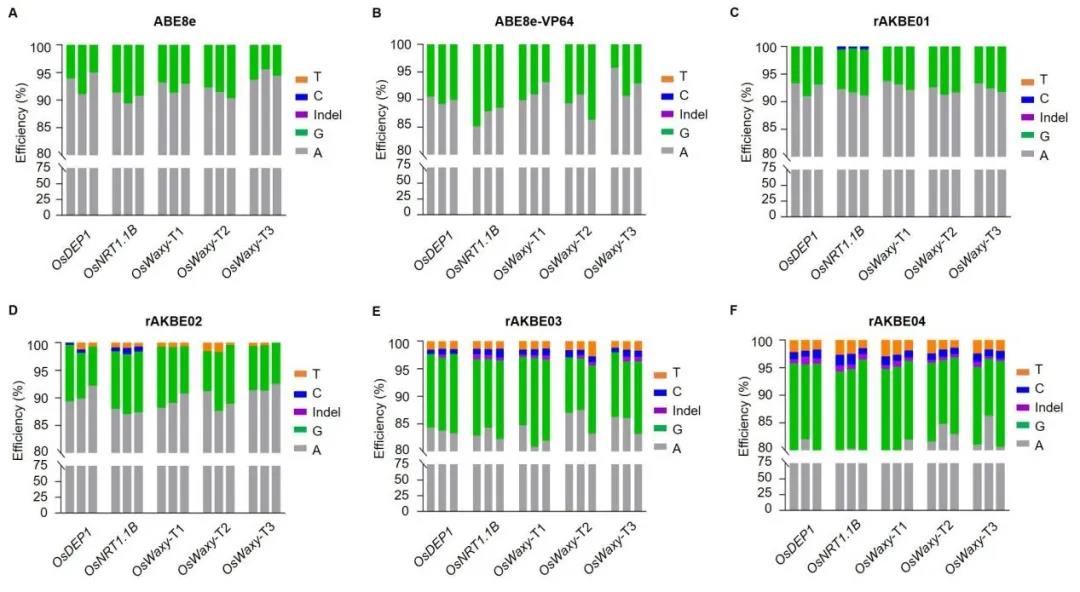
图2 水稻原生质体rAKBE介导的单碱基编辑及indel效率统计
水稻稳定转化结果表明,rAKBEs可在水稻内源基因位点介导A-to-K单碱基编辑,其中rAKBE04碱基编辑器介导腺嘌呤转换与颠换效率最高,其在水稻5个内源基因位点介导的A-to-G、A-to-T和indels效率分别为70.97%- 92.31% 、1.67%-4.84%和1.30%-5.13%(表1)。此外,与本实验室前期报道的基于hMPG的植物A-to-K碱基编辑器pAKBEs相比,rAKBE04不仅扩展了碱基编辑窗口,而且可在编辑窗口内实现更高效的A-to-G碱基转换,同时伴随着更少的A-to-T碱基颠换和indel。因此,开发具有不同编辑效果的碱基编辑器,对于在不同应用背景下,产生特定碱基替换的等位基因,进一步通过定向进化创制新种质具有重要意义。同时,该系统的研发将进一步推动水稻等其他作物的精准分子育种。
表1 水稻稳定转化株系rAKBEs编辑效率统计

本研究得到农作物基因资源和育种全国重点实验室和国家自然科学基金基础科学中心项目、海南崖洲湾种子实验室揭榜挂帅项目、作物分子育种国家工程研究中心等资助。中国农业科学院作物科学研究所博士研究生李玉才和副研究员李少雅为本文共同第一作者,夏兰琴研究员为本文通讯作者。水稻遗传转化得到作科所转基因中心吴传银研究员创新研究组的大力支持。
引用本文:
Li, Y., Li, S., Li, C. et al. Fusion of a rice endogenous N-methylpurine DNA glycosylase to a plant adenine base transition editor ABE8e enables A-to-K base editing in rice plants. aBIOTECH (2024). https://doi.org/10.1007/s42994-024-00138-8 作者简介

夏兰琴,中国农科院作物科学研究所研究员,博士生导师,农业农村部“神农英才”领军人才和中国农业科学院领军人才。现任中国农科院作物精准育种技术创新团队首席,农业农村部基因编辑创新利用重点实验室(海南)常务副主任。近年来主要从事农作物基因编辑新技术和新种质创制相关研究。率先构建了小麦多基因敲除、水稻胞嘧啶单碱基编辑、A-K单碱基编辑器、高效双碱基编辑器、单基因和多基因引导编辑及基因替换等技术体系,创制了高抗性淀粉水稻和小麦、氮高效水稻和小麦、抗除草剂水稻等系列基因编辑新种质。团队拥有农业农村部神农英才计划领军人才1人、青年拔尖人才1人、中国农业科学院青年创新专项支持计划1人。以第一或通讯作者在Nature Biotechnology、Molecular Plant等期刊发表SCI论文70余篇,获得授权发明专利30余项,其中美国授权发明专利1项。
相关阅读:
aBIOTECH | 中山大学李剑峰团队开发富集多基因CRISPR突变体和剔除Cas9转基因的双功能筛选系统
aBIOTECH | 张韬-机器学习方法在作物基因编辑中的应用
aBIOTECH | 浙江大学刘建祥团队通过多靶点基因编辑创制抗百草枯水稻
aBIOTECH | 福建农林大学关跃峰课题组报道南方大豆易炸荚的“基因疗法”
aBIOTECH | 河北农业大学赵建军/李君综述基因编辑技术在芸薹属蔬菜作物中的应用及展望
aBIOTECH | 日本冈山大学马建锋评述:基因编辑主效耐盐碱基因AT1提高作物耐盐碱性
https://blog.sciencenet.cn/blog-3458049-1426907.html
上一篇:aBIOTECH | 中国农科院作科所建立基因编辑大豆可视化识别技术体系
下一篇:特约综述 | 陈艳梅-蛋白质翻译后修饰之间的互作关系及其协同调控机理