博文
古菌利用氢气产生能量的秘密《细胞》
|
一个国际科学小组通过展示古生菌如何利用氢气,重新定义了我们对古生菌的理解。古生菌是20亿年前人类的微生物祖先。
这篇论文由莫纳什大学生物医学发现研究所的科学家们领导,包括Chris Greening教授、Jill Banfield教授和Bob Leung博士,他们重写了基础生物学的教科书。
Bob Leung博士说,这一关于地球上最古老的存在形式之一的发现也可能支持人类的存在,包括设计新的方法来利用氢来实现未来的绿色经济。
“人类直到最近才开始考虑使用氢作为能源,但古细菌已经这样做了10亿年。生物技术专家现在有机会从这些古细菌中获得灵感,用于工业生产氢气。”
在生命金字塔的最顶端,有三个生命 "领域":真核生物(动物、植物和真菌都属于真核生物)、细菌和古细菌。古生菌是一种单细胞生物,可以生活在地球上最极端的环境中。最被广泛接受的科学理论还表明,真核生物,如人类,是由一个非常古老的古生菌谱系通过交换氢气与细菌细胞融合而进化而来的。
“我们的发现使我们更接近于理解这个关键过程是如何产生包括人类在内的所有真核生物的。”Leung说。
该团队分析了数千种古细菌的基因组,寻找产氢酶,然后在实验室中生产这些酶,以研究它们的特性。他们发现一些古生菌使用一种不寻常的酶,叫做[FeFe]氢化酶([FeFe]-hydrogenases.)。
在地球上许多最具挑战性的环境中都发现了制造这些氢酶的古细菌,包括温泉、油藏和海底深处。
这些氢化酶被认为仅限于生命的两个“领域”:真核生物和细菌。在这里,研究小组首次证明了它们存在于古细菌中,并且它们的形式和功能非常多样化。
古细菌不仅具有最小的利用氢的酶,而且具有最复杂的利用氢的酶。
这篇论文表明,一些古生菌的产氢酶是地球上所有生命形式中最小的。这可以为工业环境中的生物制氢提供简化的解决方案。
Greening教授说,这些关于古细菌如何利用氢的发现对向绿色经济过渡有潜在的应用价值。
“工业目前使用珍贵的化学催化剂来使用氢。然而,我们从大自然中知道,生物催化剂的功能可以是高效和有弹性的。我们能不能用这些来改进我们使用氢的方式?”
古生菌具有古老的起源和在生物技术方面的潜在应用,继续吸引着研究人员,并为进一步发现和转化提供了有希望的途径。
古菌利用氢气产生能量的秘密《细胞》
摘要
微生物氢气(H2)循环支撑了多样厌氧生态系统的多样性和功能性。在负责此过程的三个进化上独特的氢化酶超家族中,[FeFe]氢化酶曾被认为仅限于细菌和真核生物。在这里,我们通过结合现有和新基因组的分析以及广泛的生化实验,展示了厌氧古菌编码多样、活跃且古老的[FeFe]氢化酶谱系。[FeFe]氢化酶由九个古菌门的基因组编码,并由产氢的阿斯加德古菌培养物表达。我们报告了一种在DPANN古菌中发现的超微型氢化酶,它结合了催化H-簇并产生H2。此外,我们在其他十个古菌目中识别并表征了通过[FeFe]和[NiFe]氢化酶融合形成的显著混合复合体。系统发育分析和结构建模表明混合型氢化酶具有深远的进化历史。这些发现揭示了古菌新的代谢适应机制,为生物技术发展提供了简化的H2催化剂,并且意外地揭示了两种主要的H2代谢酶之间交织在一起的进化历史。
Minimal and hybrid hydrogenases are active from archaea: Cell
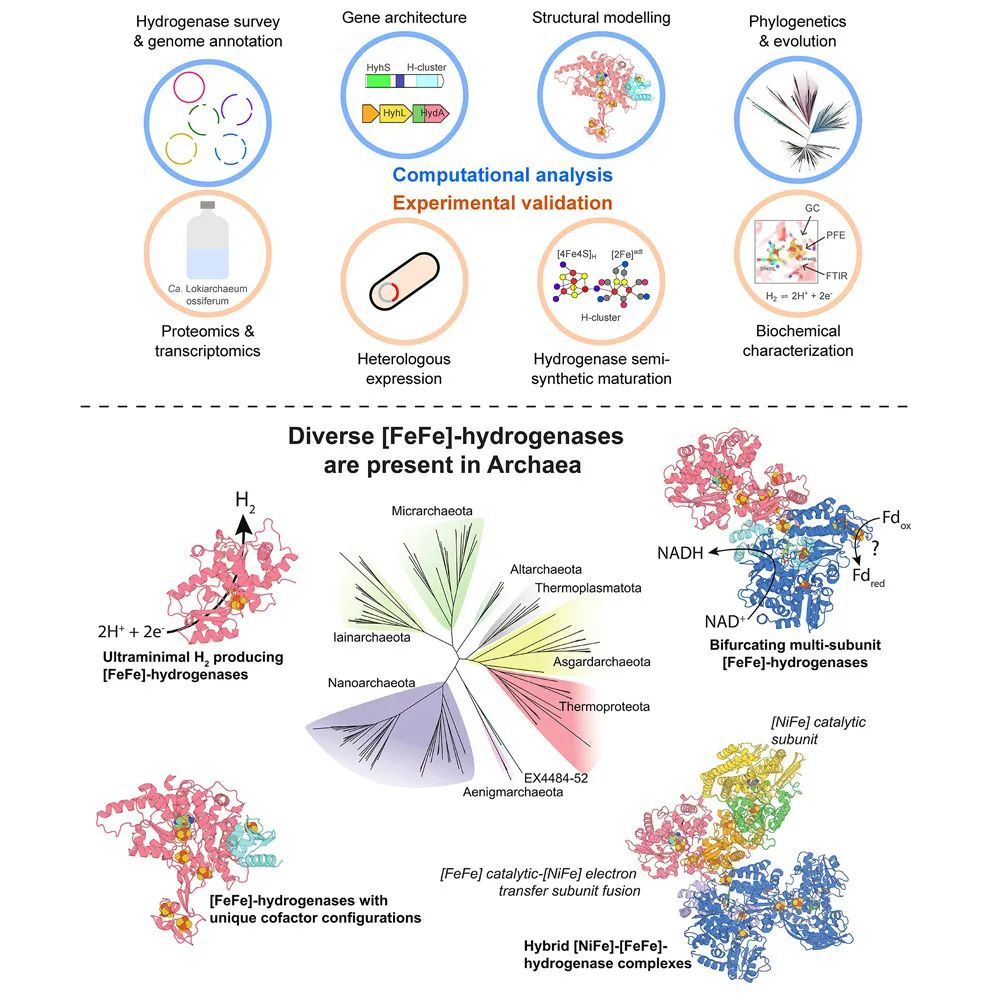
研究亮点
来自九个不同门的古菌编码结构多样的[FeFe]氢化酶
未培养的DPANN古菌产生活性超微型[FeFe]氢化酶
多样化的古菌编码古老的混合型[FeFe]和[NiFe]氢化酶
产氢的阿斯加德古菌培养物表达[FeFe]氢化酶
氢分子(H2)被誉为未来绿色能源的载体。在生物学背景下,这种富含能量的气体已经在生物能量学中发挥了核心作用,而进化已经推动了复杂的微生物H2经济体系的发展。许多细菌、古菌和微生物真核生物利用称为氢化酶的金属酶消费和产生H2气体(H2 ⇌2H+ + 2e−)。三种氢化酶在微生物中独立进化,即[FeFe]、[NiFe]和[Fe]氢化酶,它们在金属辅助因子和催化机制上有所不同。
H2在微生物生理学中扮演多重角色。跨越所有三个领域的微生物在发酵过程中产生H2以处理电子。许多细菌和古菌还利用来自H2氧化的电子进行呼吸和碳固定。最近,发现了对严格厌氧菌的能量保存至关重要的电子分岔氢化酶复合体。氢化酶现在被认为在环境上无处不在,分类学上广泛分布,编码在大多数细菌和古菌门以及许多单细胞真核生物的基因组中。人们越来越认识到,微生物H2代谢塑造了全球生物地球化学循环,支持了多样化生态系统的生物多样性,并影响了健康与疾病。此外,这些高效酶在发展中的H2能经济中有着日益增长的工业应用,并为合成催化剂的设计提供了灵感。H2可能是原始的电子供体,但继续在微生物学中发挥核心作用,既作为理想的能源,也作为可扩散的电子汇。此外,细菌和古菌之间的H2交换是真核生物起源的基础,正如各种共生假说所描述的那样。
三类氢化酶在其生理作用和分类分布上有所不同。[FeFe]氢化酶通常作用迅速但对氧气敏感,最知名的作用是在严格厌氧菌中。这些酶目前包括四个系统发育上不同的组(A-D组),可以通过基于结构域架构和遗传组织的两种不同方案进一步细分。它们包括将铁氧还蛋白氧化耦合到发酵性H2生产(A1组)的单体酶、可逆地从H2向烟酰胺腺嘌呤二核苷酸(NAD+)和铁氧还蛋白分流电子的三聚体酶(A3组)、具有甲酸脱氢酶的丝状复合体,催化依赖H2的CO2转化为甲酸(A4组)、催化性氢化酶结构域与PAS结构域融合的假定感觉氢化酶(C组)以及几个功能未定义的组(例如,B和D组)。尽管存在这种多样性,[FeFe]氢化酶都被预测依赖于相同的有机金属辅助因子进行催化,即“H-簇”。迄今为止,这些酶仅在厌氧细菌和真核生物中被特征化,似乎在培养的古菌中不存在。[NiFe]氢化酶是极其结构和功能多样的酶,由跨越所有生态系统的细菌和古菌编码。它们目前被细分为四个主要组(1-4组)和亚组,每个组在系统发育、遗传组织和生理作用上都有所不同。[NiFe]氢化酶的催化(大)亚基和电子传递铁硫(小)亚基与其他亚基结合,取决于亚组;形成的不同复合体可以介导呼吸、发酵、能量转换、电子分岔、碳固定和H2感应过程。相比之下,[Fe]氢化酶是一个更狭窄的谱系,有助于古菌的甲烷生成。三类氢化酶在系统发育上不相关,尽管它们有一些相似的结构特征,并且被认为在遗传或结构上不会关联。[NiFe]氢化酶被预测存在于最后的普遍共同祖先(LUCA)中,而[FeFe]氢化酶被认为后来在发酵性细菌中进化。
在对氢化酶分布进行全面调查的8年里,由于基因组解析宏基因组学(即从混合群落样本中恢复微生物基因组)的应用,已知的微生物多样性有了巨大的扩展。这种扩张在古菌领域尤为显著。值得注意的发展包括发现了Asgard超门,预测真核生物是从其中进化而来的;DPANN超门迅速扩张,其中包括必需的共生体;以及发现能够进行厌氧烷烃代谢的新谱系。这些古菌大多数未经培养,需要采用不依赖培养的方法来表征它们。许多新的谱系似乎能够进行H2代谢。例如,最近培养的Asgard古菌“Candidatus Prometheoarchaeum syntrophicum”通过一种电子分岔/共聚[NiFe]氢化酶的活动发酵产生H2,这被其共生的产甲烷伴侣所消耗。在这些新发现的古菌中报道的H2代谢酶属于一系列已建立和新颖的[NiFe]氢化酶亚组。几项基因组研究还暗示,未培养的DPANN古菌可能编码[FeFe]氢化酶。然而,这一点仍有争议,因为古菌似乎缺乏合成生物学上独特的催化性H-簇所需的三种成熟酶(HydEFG),并且到目前为止,没有证据表明古菌[FeFe]氢化酶的活性。
在这里,我们通过搜索所有公开可用的物种代表性分离株基因组和宏基因组组装基因组(MAGs),以及为本研究生成的多个MAGs,系统地分析了古菌域中[FeFe]氢化酶的多样性。我们依靠两项创新来验证这些发现。首先,我们使用基于AlphaFold2的结构建模来测试保守的基因簇是否编码发散的氢化酶复合体。关键的是,然后我们将异源酶生产与人工成熟相结合,以确认古菌氢化酶是否具有催化活性并显示H-簇光谱信号。通过这种综合方法,我们证明了古菌拥有活跃的超微型[FeFe]氢化酶,并识别出与之前描述的酶相比具有不同序列、结构和可能功能的意外复合体,包括与[NiFe]氢化酶的杂交复合体。这些发现改变了我们对微生物H2代谢分布和进化的理解,并具有广泛的生物学、化学和生物技术意义。
结果与讨论
结构上和遗传上多样化的[FeFe]氢化酶由9个古菌门编码
我们在Genome Taxonomy Database (GTDB)的2,339个古菌物种集群和我们的未发表古菌MAGs库中搜索了编码[FeFe]氢化酶催化亚基(HydA)的基因。总共有130个古菌基因组(90个以前报道过,40个是新的)编码了[FeFe]氢化酶,跨越9个门和17个类(图1;表S1)。除了一些Asgard古菌外,每个基因组中只存在一个[FeFe]氢化酶。这些酶通过分析结构域(表S2;图S1)、遗传组织(表S2;图S2)、成熟酶(表S3;图1)和主要系统发育(下文详细描述)进行了验证和分类。这些酶分为六个不同的组,即规范的A1组(n = 26)、A3组(n = 44)和B组(n = 12)以及在本工作中定义的E组(n = 30)、F组(n = 21)和G组(n = 3)(图1;表S2)。古菌A1组和E组[FeFe]氢化酶是由三个DPANN门(Iainarchaeota、Micrarchaeota和Nanoarchaeota)编码的推定发酵酶。这些酶的平均序列分别为363和286个残基(在排除任何截短序列后),比先前表征的最小氢化酶(Chlamydomonas reinhardtii HydA1; 457个残基)要小得多(表S2)。然而,最广泛分布的氢化酶是电子分岔/共聚A3组[FeFe]氢化酶。这种氢化酶及其伴侣二磷酸酶(HydB)和硫氧还蛋白(HydC)亚基至少由六个DPANN门、Thermoplasmatota(E2类)和一些Asgard古菌(Lokiarchaeia类)编码(图1)。

图1 系统发育和代谢多样化的古菌编码[FeFe]氢化酶
在培养的古菌中,只有Asgard古菌富集培养物编码[FeFe]氢化酶。我们在“Ca. P. syntrophicum”(图1;表S1)和我们最近报道的培养物“Candidatus Lokiarchaeum ossiferum”(表S4)的基因组中检测到了B组[FeFe]氢化酶。尽管之前推测“Ca. P. syntrophicum”通过发酵产生H2,但这种活性被认为是由其3C组[NiFe]氢化酶引起的,而其[FeFe]氢化酶被错误地注释为F420H2依赖性脱氢酶亚基;基于之前报道的转录组,[FeFe]氢化酶(每千碱基百万读数309 [RPKM])与[NiFe]氢化酶(333 RPKM)的表达水平相似,这表明它可能对观察到的H2生产有贡献。我们还进行了蛋白质组学和转录组学分析,以深入了解“Ca. L. ossiferum”的代谢能力(表S4);这种古菌也高水平合成[FeFe]氢化酶(18.03标记自由定量[LFQ]强度[log2]),但其[NiFe]氢化酶(24.00 LFQ强度[log2])是细胞中最丰富的复合体之一,因此可能主导H2生产(表S4)。尽管如此,鉴于这些酶通常比它们的[NiFe]对应物具有更高的活性,并且所有生化特征化的3c组[NiFe]氢化酶在细胞条件下氧化H2,[FeFe]氢化酶可能对H2生产有贡献。考虑到这两种培养物的极端缓慢生长速率和产量,我们目前无法对这些酶在体内的不同作用进行更深入的分析。几种其他Lokiarchaeia和Heimdallarchaeia的MAGs也编码多样化的[FeFe]氢化酶(图1;表S1)。
为了更好地理解推定的古菌[FeFe]氢化酶的结构和功能,我们使用AlphaFold2进行了结构建模。古菌A1组和E组[FeFe]氢化酶是单聚体酶(仅含HydA),预测会折叠成具有溶剂暴露催化H-簇的H-簇域(图2A和2B;图S3A和S3B)。然而,与细菌[FeFe]氢化酶相比,这些酶的环结构更加紧密,缺少额外的铁硫簇(图S4)。每个建模的酶都包含连接H-簇所需的半胱氨酸残基,尽管它们的质子转移残基不同(图S4A–S4C)。因此,尽管这些酶体积小,但理论上能够进行H2催化。来自“Ca. P. syntrophicum”(表示为Ps)的B组[FeFe]氢化酶建模为HydA和HydC亚基之间的异二聚体,这两个亚基由基因簇编码;它包含两个2×[4Fe-4S]铁氧还蛋白样域,一个作为从H-簇到蛋白主体另一未知功能的电子中继(图2C)。结构建模还支持了这样一个观点:古菌A3组[FeFe]氢化酶形成了类似于最近在发酵和产乙酸细菌中结构特征化的三聚体电子分岔复合体(HydABC)(图2D;图S3D)。
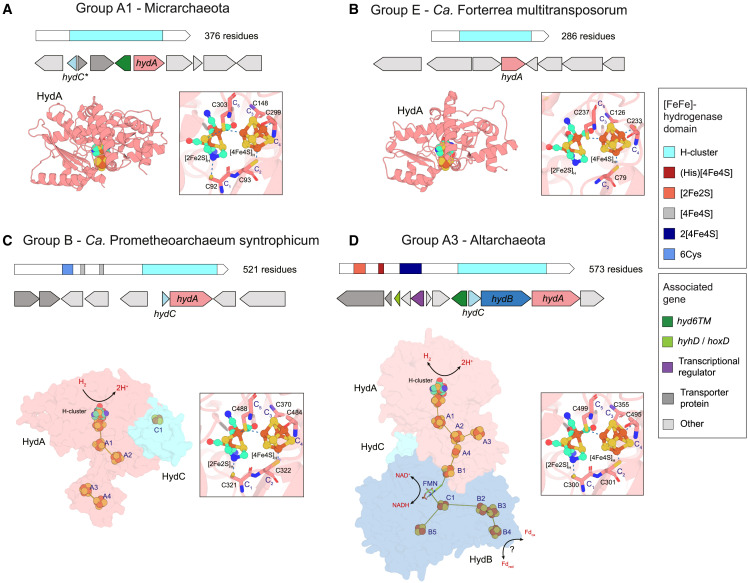
图2 古菌编码遗传上和结构上多样化的[FeFe]氢化酶
古菌的[FeFe]氢化酶具有催化活性并显示出H-簇光谱信号
我们测试了来自古菌的A1组、B组和E组[FeFe]氢化酶是否能结合催化H-簇并产生H2。为此,我们在大肠杆菌BL21(DE3)中异源表达了五种酶,使用合成模拟物[2Fe]adt([Fe2(azadithiolate)(CO)4(CN)2]2−)对其进行厌氧成熟,并按照既定协议使用气相色谱测量全细胞裂解物的H2产量(表S5;图S5A)。相对于阴性对照,所有三组成熟酶都清晰可见H2的产生(图3A;表S5)。最高的活性来自于Micrarchaeota组A1酶(表示为Mu)。含有Mu的细胞裂解物产生H2的速率是众所周知的绿藻C. reinhardtii中的[FeFe]氢化酶HydA1的一半,后者在所有实验中作为阳性对照包含在内。来自地下水古菌“Candidatus Iainarchaeum andersonii”的同源酶(表示为Ia)也产生了H2,尽管速率低了100倍。我们还观察到来自Asgard古菌的B组酶(Ps)35和来自地下水古菌“Candidatus Forterrea multitransposorum”的E组酶(表示为Fm)有大量活性。我们注意到,由于异源表达、折叠和成熟效率在酶之间可能存在差异,观察到的H2产生活性不应被视为比活性;这由Mu和Ia氢化酶之间的活动差异所说明,尽管它们具有高度的序列同源性。尽管如此,这些结果验证了DPANN和Asgard古菌均编码功能性的[FeFe]氢化酶,且最小的A1组和超微的E组酶都具有活性。
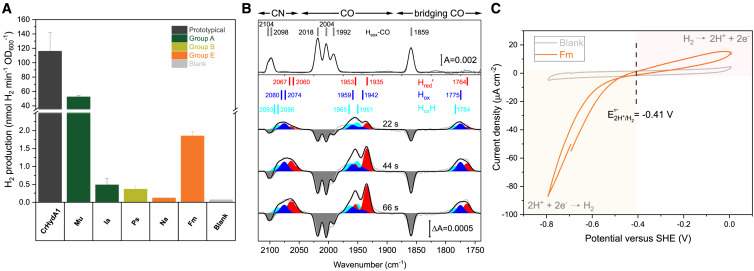
图3 古菌编码的三类[FeFe]氢化酶具有催化活性
为了更详细地了解这些超微型酶的性质,我们以E组[FeFe]氢化酶Fm为例进行了分离。在严格的厌氧条件下进行纯化并通过半酶促重构铁-硫簇后,测定该酶的铁含量为每个蛋白4.2±0.4(图S5B和S5C),与结构建模显示该酶含有单个[4Fe4S]簇一致(图2)。随后将重构的Fm氢化酶与[2Fe]adt孵育。通过衰减全反射傅里叶变换红外(ATR-FTIR)光谱学成功验证了H-簇组装,因为在预期的CO/CN配体带区域的FTIR光谱中容易观察到尖锐的辅因子带(图3B;图S5D)。我们的光谱分析表明,该酶是以抑制状态分离的。然而,光化学还原导致了向催化活性状态的转变(图3B;图S5E),让人联想到氧化活跃准备状态(Hox和HoxH)以及进一步的还原状态(Hred′)。
通过蛋白质膜电化学(PFE)研究了Fm的催化性质。在H2气氛下记录的酶的循环伏安图显示出通常与[FeFe]氢化酶相关的典型的双向催化行为(图3C)。在高驱动力(±300 mV vs. 可逆氢电极[RHE])下观察到的还原和氧化电流比较表明,该酶明显偏向于H+还原催化相对于H2气体氧化。这与它最接近的已表征同源物——来自细菌Thermotoga maritima的C组[FeFe]氢化酶(序列同一性28%)形成对比,后者明显偏好H2氧化。
总的来说,这些结果表明古菌单聚体[FeFe]氢化酶在一个非常明确的环境中结合了一个氧化还原活性的H-簇,并暗示它们介导发酵性H2产生。
在未培养的古菌中,[FeFe]和[NiFe]氢化酶形成复合体
值得注意的是,F组[FeFe]氢化酶似乎与[NiFe]氢化酶形成复合物。21个基因组通过五个基因簇编码了这些复合物,包括来自Bathyarchaeia、Brockarchaeia、Thermoplasmata、Thermoproteia和Lokiarchaeia类(图1;表S2)。它们的决定性特征是C末端[FeFe]氢化酶催化域(HydA)与N末端与3组[NiFe]氢化酶小亚基(HyhS)同源的域的融合(图4A;图S3E和S3F)。与这个融合相邻的还有四个其他基因:3组[NiFe]氢化酶的大亚基(HyhL)、电子分岔/共聚A3组[FeFe]氢化酶的二磷酸酶(HydB)和硫氧还蛋白(HydC)亚基,以及一个含有四个铁-硫簇的导管亚基(此处称为HydD)(图S2)。硫氧还蛋白、二磷酸酶和导管亚基分别与复合体I的NADH脱氢酶模块的NuoE(亚基E)、NuoF(亚基F)和NuoG(亚基G)同源。
所有这四个基因也存在于最近识别且结构上特征化的来自Acetomicrobium mobile的电子分岔[NiFe]氢化酶中;然而,古菌复合体是不同的,因为它包含一个真正的[FeFe]氢化酶催化亚基,其H-簇域与[NiFe]氢化酶小亚基融合。在古菌的[FeFe]氢化酶中,F组酶显示出最保守的遗传组织,这可能是由于它们与[NiFe]氢化酶的关联(图S2)。遗传和系统发育上独特的G组[FeFe]氢化酶,专属Brockarchaeia属JAAOZO01,也可能形成杂交复合物(图S2;表S2)。
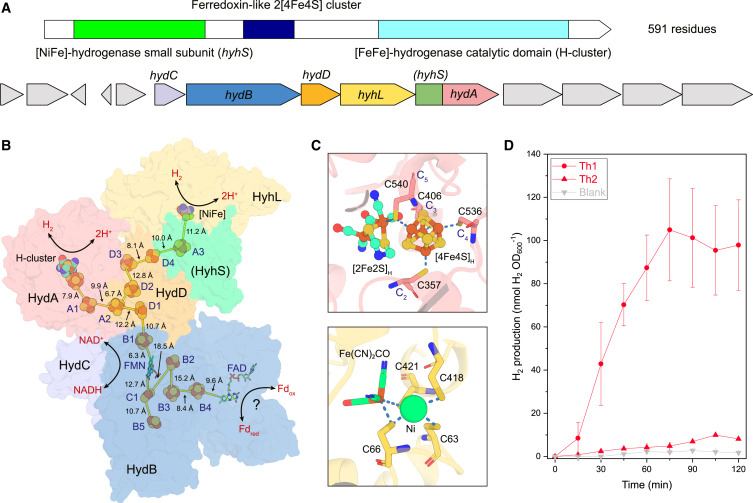
图4:古菌编码的[FeFe]和[NiFe]氢化酶预计会形成独特的复合体为了确认F组[FeFe]氢化酶的催化活性,我们从候选谱系Thermoplasmata SG8-5重组表达了两种HydA-HyhS融合蛋白,并进行了人工成熟。其中一种酶(Th1)在时间过程中迅速产生H2,与C. reinhardtii酶相比,相对活性为5%(图4D;表S5)。相比之下,另一种酶(TH2)只显示出低水平的活性(图4D)。这些蛋白质难以纯化,阻碍了详细的体外表征。很可能,如果将这些酶与其他亚基组装成复合物,它们会显示出更高的活性,尽管鉴于编码它们的古菌未经培养,且[NiFe]氢化酶难以异源生产,实现这一点将非常具有挑战性。这些测量结果仍然证实它们是真正的氢化酶,其活性可归因于H簇。最后,我们进行了AlphaFold2建模,以推断[FeFe]和[NiFe]氢化酶是否结合(图4B和4C)。单独建模时,杂交亚基被预测包含一个[FeFe]氢化酶催化域和一个[NiFe]氢化酶铁硫簇域,两者之间由一个长柔性连接子隔开(图S3F)。观察到保守的半胱氨酸配体,需要连接[FeFe]氢化酶的H簇和[NiFe]氢化酶组分的三个电子传递[4Fe4S]簇(图4C;图S4A-S4C)。然而,对所有五个遗传上连续的亚基(HydA-HyhS, HydBCD, HyhL)进行建模表明,它们通过多个氢键和盐桥形成一个稳定的电子分支/汇合复合体(ΔG = -97.06至-219.7 kJ mol−1;PISA分析)(图S3I)。结构模型表明,该复合体通过一系列铁硫簇从[FeFe]氢化酶臂或[NiFe]氢化酶接收电子,到一个可能的电子汇聚[4Fe4S]簇上,或者反之亦然。此后,电子被预测同时转移到高电位NAD+在HydC亚基上,以及一个未确定的低电位受体(可能是铁氧还蛋白)在HydB亚基的谷氨酸合成酶(GltA)域上(图4B)。这些观察结果非常引人注目,因为[NiFe]和[FeFe]氢化酶以前并不知道会结合。而且,它们暗示[NiFe]氢化酶小亚基具有惊人的模块化,因为它显然与[NiFe]和[FeFe]氢化酶都有共进化。
[FeFe]氢化酶使得在各种古菌中进行发酵和电子分叉成为可能。
我们试图了解各种[FeFe]氢化酶在古菌代谢中的作用。为此,我们对高质量古菌基因组进行了注释,以寻找与主要能量保存和碳获取过程相关的基因。所有编码[FeFe]氢化酶的古菌都被预测为专性厌氧生物,因为它们缺少末端氧化酶。这与从通常是缺氧生态系统中检索到的基因组一致,尤其是地下水、厌氧消化器以及来自温泉、热液喷口和淡水的沉积物(图1;表S1)。基于检索到的基因组,大多数DPANN古菌很可能依赖宿主衍生的有机化合物作为共生专性发酵者;一致地,它们通常编码了用于碳水化合物(主要是淀粉)和芳香族化合物的降解和发酵的基因,但一般缺乏呼吸还原酶或碳固定途径(图1)。其他古菌门被预测能够采取更广泛的代谢策略,与其较大的基因组大小相符(图S1),包括β-氧化、厌氧呼吸和不同程度的碳固定(图1)。值得注意的是,一些阿斯加德古菌编码了富马酸还原酶(Frd)、还原性脱卤酶(Rdh)和厌氧亚硫酸盐还原酶(Asr),后者也在其他四个门的某些MAGs中编码。几个谱系还被预测能够通过Wood-Ljungdahl(Lokiarchaeia, Thermoproteota)或逆向三羧酸循环(Lokiarchaeia, Heimdallarchaeia, Thermoplasmatota)途径进行自养(图1;表S1)。大多数MAGs还编码了已知在核苷挽救中起作用的核酮糖-1,5-二磷酸羧化酶/加氧酶(RuBisCO)谱系。
A组、B组和E组[FeFe]氢化酶可能在多种古菌中的有机碳发酵过程中促进辅因子再生。一半的古菌编码了2-氧代酸-铁氧还蛋白氧化还原酶,如丙酮酸-铁氧还蛋白氧化还原酶,它将糖酵解的最终产物(丙酮酸)的氧化与铁氧还蛋白的还原相结合,大多数古菌可以通过将产生的乙酰辅酶A(CoA)通过乙酰辅酶A合成酶或乙酸酯激酶反应转化为最终产物乙酸来获得ATP(图1)。结构建模支持(图2),单体A1组、B组和E组氢化酶被预测将铁氧还蛋白的再氧化与H2生产相耦合。与此角色一致,2-氧代酸-铁氧还蛋白氧化还原酶基因经常与Nanoarchaeota和Micrarchaeota MAGs中的[FeFe]氢化酶基因相邻(图S2)。相比之下,三聚体A3组[FeFe]氢化酶被预测同时再氧化铁氧还蛋白(主要由丙酮酸-铁氧还蛋白氧化还原酶还原)和NADH(例如,在糖酵解过程中还原)(图2),与其细菌对应物一致。
相应地,电子分叉/合流的A3组[FeFe]氢化酶与那些拥有相对复杂碳水化合物降解途径的DPANN古菌相关联(即Woesearchaeles、Altarchaeota、某些Iainarchaeota),通过这些途径NAD+和铁氧还蛋白将被还原(图1)。同样值得注意的是,许多DPANN古菌编码最小A1组和超小型E组[FeFe]氢化酶作为其唯一的H2代谢酶(表S1);结合[FeFe]氢化酶相比[NiFe]氢化酶的更简单成熟途径,这些基因组简化的微生物(平均完整度标准化基因组大小:1.2 Mbp)已经最小化了代谢H2的遗传和细胞成本。需要进一步研究来确定单体A1组、B组和E组[FeFe]氢化酶的哪些生化特征不同,以及它们是否具有不同的生理作用。不能排除B组酶反而消耗H2以支持阿斯加德古菌的厌氧呼吸或碳固定的可能性;然而,鉴于它们报告的H2产生活性(图3)、结构特征(图2)以及“Ca. P. syntrophicum”的产氢生活方式,这似乎不太可能。
混合型氢化酶的生理作用尚不清楚。这些酶仅由代谢功能更多样化的古菌编码,包括那些具有碳固定、β-氧化和使用第4组[NiFe]氢化酶进行能量转换的能力的古菌(图1)。预测的结构表明,这些酶通过将电子从H2转移到NAD+和可能是铁氧还蛋白,或反之亦然,从而有助于电子分叉(图3)。然而,一个单一复合体包含两个看似冗余的氢化酶模块是特殊的。一个可能的解释是,其中一个氢化酶模块可能作用于H2以外的其他底物,尤其是考虑到第3组[NiFe]氢化酶可以在体外作为硫化氢酶(即,介导元素硫还原为硫化氢)。
另一个理由是,两个氢化酶模块可能在亲和力和/或氧气耐受性方面有所不同,使得在广泛的环境条件下能够有效氧化H2。然而,最有可能的是,这些复合体充当氧化还原阀,当还原剂积累时,将电子从NADH和还原的铁氧还蛋白转移到产H2的[FeFe]氢化酶,或者在其它情况下从消耗H2的[NiFe]氢化酶转移到NAD+和氧化的铁氧还蛋白;这与之前观察到的在细菌异养生长期间响应底物可用性和氧化还原状态变化的电子分叉氢化酶作为产H2的氧化还原阀一致。
最近报道了调节相反反应的其他酶复合体,即谷氨酸合成酶(GltAB)与谷氨酸脱氢酶(GudB)之间,这些酶的GltA结构域也在混合型氢化酶复合体中共享。
[FeFe]氢化酶已被古菌多次获得,并与[NiFe]氢化酶有着古老的联系。
我们通过其催化亚基的系统发育分析(图5)和三种成熟酶(图S6)调查了[FeFe]氢化酶的进化历史。与其分类到已知组别一致,古菌的A1、A3和B组[FeFe]氢化酶与各种细菌和真核生物的同源物聚类;A组酶至少形成了六个辐射,表明它们在几个独立事件中从细菌酶横向获得,而古菌的B组和E组酶各自形成了一个单系分支(图5)。基于使用最佳支持模型构建的系统发育树(图5)及其结构的简单性(图2B),古菌的超小型发酵E组酶与细菌的多结构域感觉C组氢化酶形成了姐妹分支。
我们还重新审视了[FeFe]氢化酶在真核生物中的假定起源,鉴于我们发现[FeFe]氢化酶既存在于阿斯加德古菌中也存在于单细胞真核生物中,这符合关于真核生物起源的共生假说。我们的数据不支持所有真核生物[FeFe]氢化酶都是从阿斯加德古菌祖先垂直获得的假设。真核生物的酶至少聚类成五个不同的分支,跨越A组和B组,表明有多次横向获得(图5)。此外,与真核生物的古菌祖先最接近的阿斯加德基因组和与线粒体最相关的α-变形菌基因组中都缺少[FeFe]氢化酶,尽管这一观点可能会随着新基因组的增加而改变。尽管如此,在这些谱系中的三个里,古菌和真核生物的[FeFe]氢化酶聚类在一起(包括Lokiarchaeia与Tritrichomonas),表明一些真核生物和古菌可能在其多样化过程中后来交换了[FeFe]氢化酶基因(图5)。
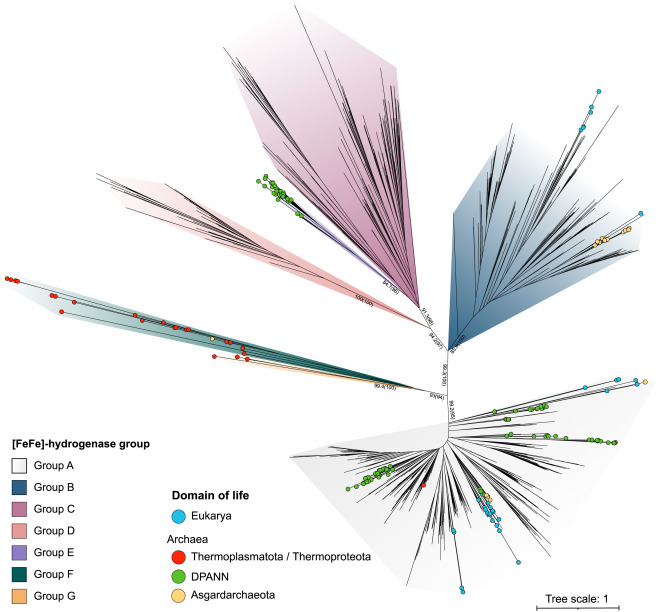
图5 古菌中的[FeFe]氢化酶多样且古老
我们还研究了合成[FeFe]氢化酶辅因子的三种成熟酶HydE、HydF和HydG的分布和系统发育。在编码[FeFe]氢化酶的130个古菌基因组中,只有五个编码全套成熟酶,另外三个编码不完整的一套(表S3)。尽管基因组不完整意味着氢化酶和成熟酶的共发生会被低估,但这并不能解释大多数基因组中缺乏成熟酶的原因。实际上,即使在培养的产氢古菌“Ca. P. syntrophicum”的基因组中,以及从中异源产生活性[FeFe]氢化酶的“Ca. F. multitransposorum”的封闭基因组中,也缺少成熟酶基因(图3)。因此,古菌可能通过替代途径合成[FeFe]氢化酶。存在替代途径与各种真核生物的报道一致,这些真核生物缺乏所有(如贾第鞭毛虫、内阿米巴)或一些(如Mastigamoeba)成熟酶,但仍然能够制造催化活性的产氢氢化酶,以及最近关于阴道毛滴虫中细胞质[FeFe]氢化酶的报道,以及在缺乏成熟酶的集胞藻细胞中异源产生活性[FeFe]氢化酶的报道。
与结构亚基的发现一致,对HydE、HydF和HydG的系统发育分析表明,古菌在多个场合获得了成熟酶(图S6)。几个古菌成熟酶与我们最近在衣原体和真核生物中鉴定的成熟酶密切相关。
系统发育分析还表明杂交氢化酶的起源很古老。在[FeFe]氢化酶催化亚基的系统发育树中,F组和G组氢化酶的序列共同形成了一个与A组和B-E超类群相距较远的单系分支,这表明这些古菌酶早期就发生了分化(图5)。这些序列形成了长分支,鉴于它们与其他氢化酶的差异(与模型氢化酶C. reinhardtii HydA1的序列同一性约为25%)。这些不同的古菌氢化酶的发现提出了一个问题,即[FeFe]氢化酶是首先在古菌还是细菌中进化;然而,由于缺乏[FeFe]氢化酶祖先的非氢化酶同源物,无法自信地确定树根以做出有力的推断。在[NiFe]氢化酶的系统发育树中,融合蛋白的催化(大)亚基形成了多个簇(图S7A)。相反,与F组[FeFe]氢化酶融合的铁硫(小)结构域和G组[FeFe]氢化酶下游的铁硫亚基各自在3组[NiFe]氢化酶中形成独特的单系亚组(图S7B)。因此,正如基因融合所指示的,杂交复合物的铁硫结构域与[FeFe]氢化酶催化亚基的共进化比与[NiFe]氢化酶催化亚基更为强烈。我们提议正式承认杂交氢化酶的组件为不同的谱系,即F组和G组[FeFe]氢化酶以及3f组和3g组[NiFe]氢化酶,鉴于它们独特的系统发育、结构和潜在的生理作用。
本研究的不足
尽管我们的组合方法显示古菌的[FeFe]氢化酶是有活性的,但这项研究并未直接观察到它们在原生宿主中的活性。几乎所有编码这些酶的古菌都是未培养的,除了两种生长速度极慢、产量低的Asgard古菌富集培养物外,因此目前没有遗传或生化系统可用于研究它们在原生宿主中的情况。虽然异源表达的古菌氢化酶结合了合成的催化H-簇并经常显示出高催化活性,但不能完全排除一些古菌通过未知途径原生产生一个独特但相关的催化簇进行H2催化的可能性,特别是考虑到大多数古菌缺乏三种常规的[FeFe]氢化酶成熟酶。鉴于其依赖于异源表达和半合成成熟,本研究无法确定这些氢化酶的原生活性率,特别是那些预测会形成多亚基复合物的酶。此外,尽管全基因组代谢分析和结构建模有助于推断[FeFe]氢化酶在宿主细胞中可能的功能,但在宿主细胞中进行大量实验是必要的,以确认它们的生理作用和相互作用。
结论
古菌已经演化出截然不同的方式使用[FeFe]氢化酶来适应厌氧环境。一方面,DPANN古菌已经演化出超微型酶,以高效处理来自碳水化合物发酵的还原剂。相反,如Brockarchaeia和Lokiarchaeia这样的谱系使用独特的[NiFe]和[FeFe]氢化酶杂交体——迄今描述的最复杂的氢化酶之一——来支持它们多样化的氧化还原生物学。这些发现将[FeFe]氢化酶从四组扩展到七组,每组都有独特的系统发育、结构和功能。除了增加我们对古菌生物学的理解外,这些发现还重新定义了我们对氢代谢和氢化酶生物化学的理解,揭示了:(1)来自生命三域的[FeFe]氢化酶都是有活性的,(2)两个主要的氢化酶类别共同进化,以及(3)氢化酶的最小尺寸。超微型氢化酶还提供了理想的模板,既可以理解酶促H2催化,例如速率、方向性、亲和力和氧敏感性的决定因素,也可以作为定向进化高效H2转化生物催化剂的灵活支架。更广泛地说,我们的方法还强调了将基因组解析宏基因组学与准确的蛋白质结构预测和异源生产研究相结合,以发现未培养微生物中新的酶和功能的潜力。
https://blog.sciencenet.cn/blog-41174-1438314.html
上一篇:太空旅行的健康影响《自然》
下一篇:灵长类动物初级视皮层视觉工作记忆的神经元表示