博文
中国科学院海洋研究所唐赢中研究员团队:海洋微藻DNA/RNA在沉积物中的稳定性及其在宏条形码分析中的意义 | IJMS
||
原文出自 IJMS 期刊
Chai, Z.; Liu, Y.; Jia, S.; Li, F.; Hu, Z.; Deng, Y.; Yue, C.; Tang, Y.-Z. DNA and RNA Stability of Marine Microalgae in Cold-Stored Sediments and Its Implications in Metabarcoding Analyses. Int. J. Mol. Sci. 2024, 25, 1724. https://www.mdpi.com/2660488
通讯作者
唐赢中 研究员
中国科学院海洋研究所
中国科学院海洋研究所,理学博士,研究员,博士生导师。主要从事甲藻有害藻华生态学研究,包括藻华藻类生活史、营养生态学、生态毒理学、化感作用等方面。主持和参与完成科研项目近20项,已在PNAS、Water Research、Molecular Ecology、Fundamental Research、Harmful Algae 等国际重要学术期刊发表论文120余篇,总引用3250余次,H指数29。担任Harmful Algae、Journal of Oceanology and Limnology 杂志副主编、中国藻类学会理事、海洋湖沼学会水环境分会委员 (理事)。
第一作者
柴召阳 助理研究员
中国科学院海洋研究所
中国科学院海洋研究所,理学博士,助理研究员。主要从事甲藻有害藻华生态学研究。主持和参与完成科研项目7项,已在Molecular Ecology、Science of the Total Environment、Harmful Algae 等国际权威杂志发表学术论文近30篇,总引用750余次,H指数13。
文章导读
环境DNA宏条形码技术是指通过对环境样品中混合的DNA进行PCR扩增、高通量测序获得大量序列,并通过与数据库中序列信息的比对,实现环境样品中物种的快速鉴定和多样性研究。该方法能够快速有效的批量检测出环境样品中的物种种类,已被广泛的应用于各种环境 (海洋、陆地、大气等) 中微型生物多样性的研究,比如水体样品中微藻的多样性研究、沉积物样品中微藻休眠体细胞的多样性研究等。但是由于该方法是基于环境中的DNA信息,且无法提供细胞形态信息,所以环境中残存的胞外DNA (即细胞破碎后残留的DNA) 可能会对该方法的准确性造成干扰。
甲藻休眠孢囊是甲藻生活史中的一个特殊阶段,绝大多数甲藻孢囊是由水体中营养细胞受到环境因素诱发或胁迫后通过有性结合形成二倍体运动合子、合子失去鞭毛、沉入水底后形成的。休眠孢囊的种类组成和多样性具有重要的生态学意义:既可以“反演”水体环境的历史变化,又可以“预见”有害藻华发生的潜在风险,同时有助于深入认识甲藻种群的地理扩散和生物入侵风险 (认识、建立种类多样性“本底”或者阐明某特殊种类的本地历史)。近年来,环境DNA宏条形码技术已被应用于甲藻休眠孢囊多样性的研究,该方法相较于传统形态学方法不仅可以获得更准确更完备的物种名录,尤其是能够检测出大量的未知种类 (即未被研究、描述的种类) 和新近发现、描述的种类,这在很大程度上弥补了传统形态学方法的不足。但因宏条形码技术无法提供细胞形态信息,沉积物中检测到的信号 (特别是此前未曾报道产孢囊的种类) 可能会被质疑是源于营养细胞或残留DNA。因此,营养细胞及其碎片中DNA/RNA在沉积物中的稳定性对于使用环境DNA宏条形码技术研究微藻休眠体细胞多样性的准确性至关重要,然而有关营养细胞及其碎片中DNA/RNA在沉积物中的稳定性研究几乎阙如。
International Journal of Molecular Sciences (IJMS) 期刊于2024年1月31日发表了文章“DNA and RNA Stability of Marine Microalgae in Cold-Stored Sediments and Its Implications in Metabarcoding Analyses”。中国科学院海洋研究所柴召阳博士为该文章第一作者,唐赢中研究员为通讯作者。本研究设计、构建了两种人工沉积物 (I和II),使用qPCR方法和宏条形码技术研究了低温厌氧环境下海洋微藻营养细胞DNA/RNA在沉积物中的稳定性及其对沉积物中甲藻孢囊检测的影响。
研究过程与结果
本研究在实验室内设计、构建了两种人工沉积物,I型沉积物中添加3种海洋微藻营养细胞,使用qPCR方法检测不同储存时间内沉积物中3种微藻DNA/RNA含量变化;针对II型沉积物 (包含2种甲藻营养细胞和3种甲藻休眠孢囊),除使用qPCR方法监测不同储存时间内2种营养细胞DNA含量的变化外,还应用宏条形码技术研究了其中甲藻 (包括营养细胞和休眠孢囊) 的种类组成和相对丰度的变化,以此判断沉积物中营养细胞DNA对甲藻休眠孢囊检测的影响。
1. I型沉积物中3种微藻营养细胞DNA/RNA稳定性
沉积物中微藻营养细胞DNA/RNA低温厌氧环境中快速降解,4 °C储存60天内,微藻营养细胞DNA降解92%以上,RNA降解98%以上 (图1)。

图1. I型人工沉积物中三种微藻DNA (A, C, E)/RNA (B, D, F) 降解率 (抑食金球藻:A, B;红色赤潮藻:C, D;尖顶斯氏藻:E, F)。
2. 甲藻营养细胞对宏条形码技术研究甲藻休眠孢囊多样性的影响
通过对宏条形码测序结果的分析发现,随着存储时间的增加,II型沉积物中两种甲藻营养细胞 (米氏凯伦藻和剧毒卡尔藻) 的相对丰度逐渐降低,4 °C储存45天后,米氏凯伦藻和剧毒卡尔藻的相对丰度分别骤降至1.99%和5.54%;三种甲藻孢囊 (尖顶斯氏藻、安德森亚历山大藻、微网裸甲藻) 的相对丰度则随着存储时间的增加逐渐升高,低温存储120天后,三种孢囊的相对丰度由起始的50%增至92% (图2)。PCoA的分析结果显示低温存储30天内的样品的群落结构与存储时间与超过45天的样品存在显著差异,该结果与相对丰度变化趋势相吻合 (图3)。另外,研究人员通过qPCR方法对II型沉积物中米氏凯伦藻和剧毒卡尔藻的DNA进行跟踪定量发现,经过4 °C储存120天后,米氏凯伦藻和剧毒卡尔藻DNA的降解率分别达到96.04%和97.02% (图4)。
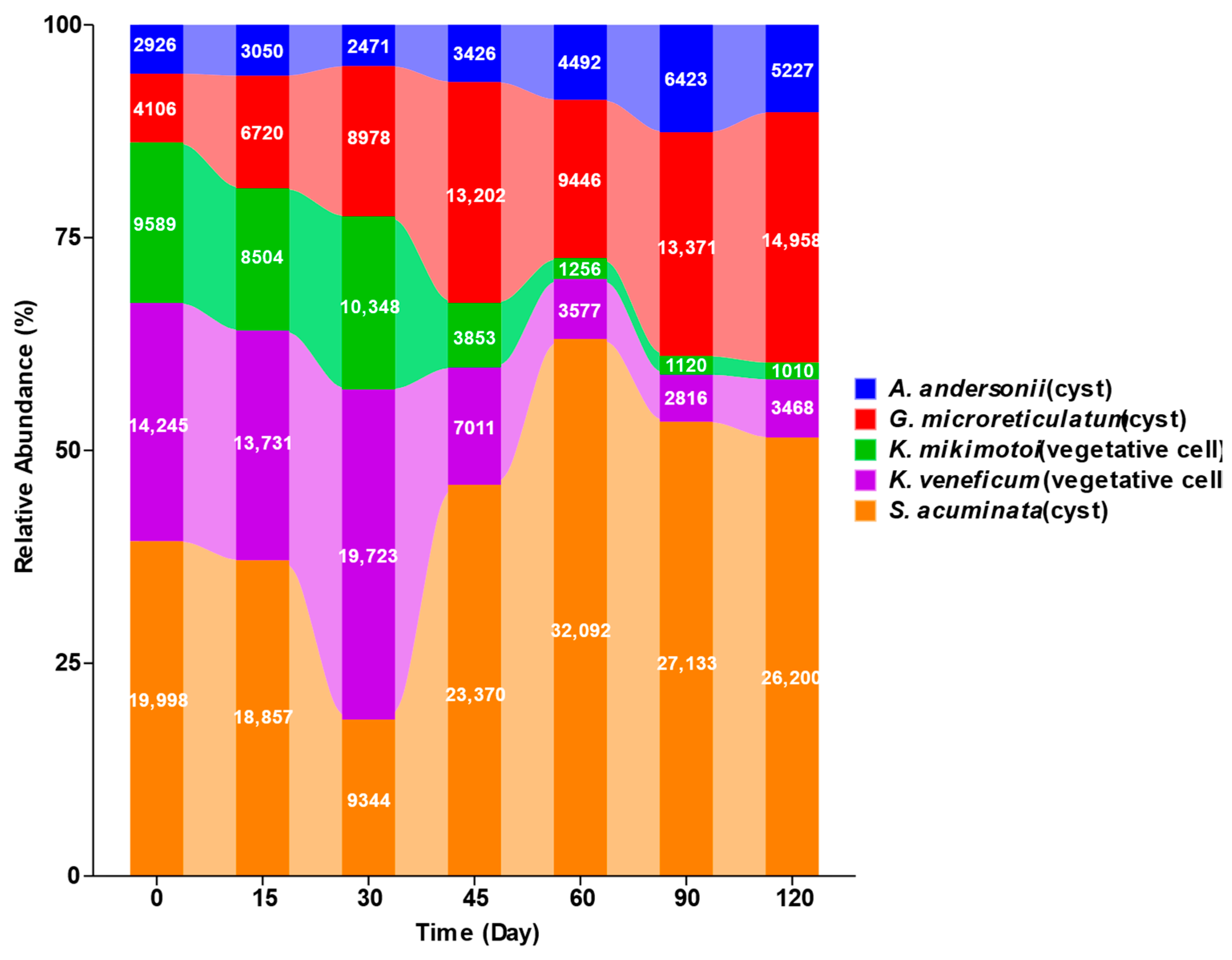
图2. II型人工沉积物中甲藻营养细胞与孢囊的相对丰度变化。
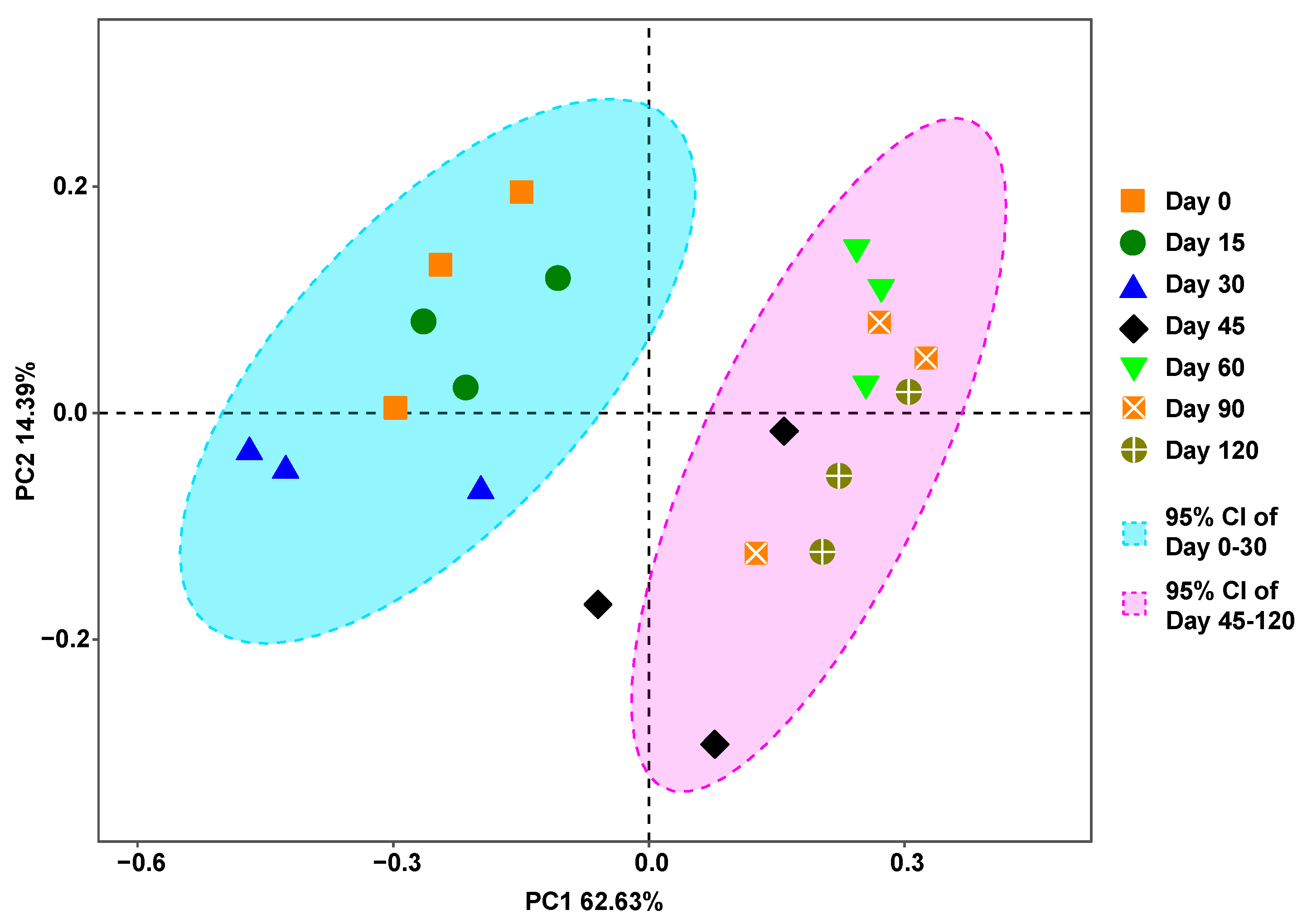
图3. II型人工沉积物样品PCoA分析。
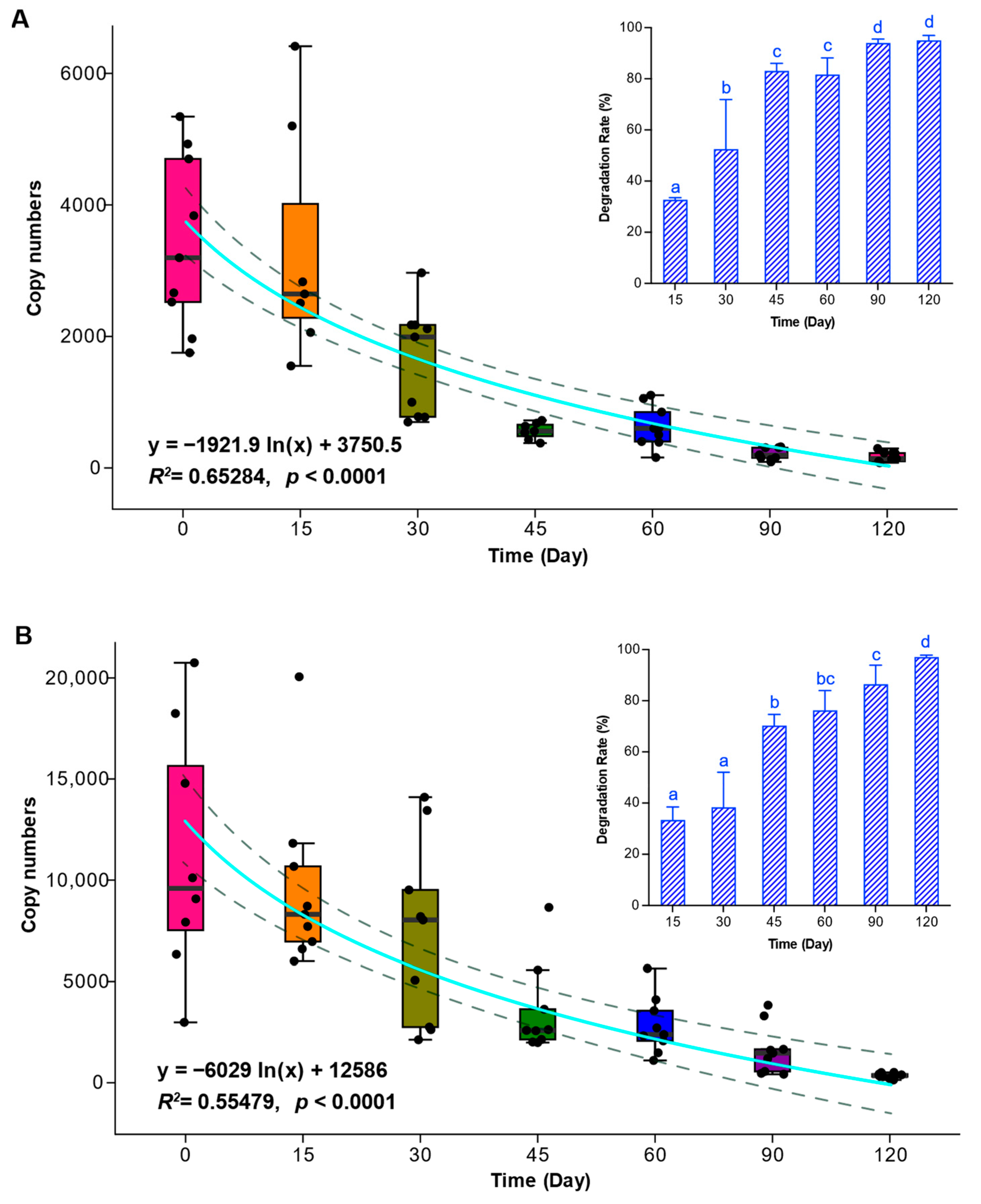
图4. II型沉积物中甲藻营养细胞 (米氏凯伦藻:A;剧毒卡尔藻:B) 28S rRNA基因拷贝数与储存时间的相关关系及DNA降解率。
研究总结
本研究发现即使是在低温的存储条件下,随着存储时间的增加,沉积物中微藻营养细胞28S rRNA基因的拷贝数发生显著的指数型下降,四个月的低温存储,营养细胞DNA和RNA的降解率分别达到95%和98%以上。研究结果表明,随着存储时间的增加,即使在比当前测序深度更深的条件下,宏条形码技术在沉积物中检测到的信号极有可能是来自沉积物中完整的细胞,而不是细胞破碎后残留的DNA。比如宏条形码技术在低温存储数月、数年的沉积物中检测到的甲藻信号最大可能是来自完整的甲藻休眠孢囊,而几乎不可能是微藻营养细胞破碎残留的DNA。本研究为应用宏条形码技术研究微型生物多样性的实验设计、样品预处理等具有重要意义。
IJMS 期刊介绍
主编:Maurizio Battino, Marche Polytechnic University, Italy
期刊发表生物化学与分子生物学、生物材料、生物物理、生物医学、化学和纳米科学等分子相关领域研究,已被 Scopus、SCIE (Web of Science)、PubMed 等数据库收录。
2022 Impact Factor:5.6
2022 CiteScore:7.8
Time to First Decision:16.3 Days
Acceptance to Publication:2.6 Days


https://blog.sciencenet.cn/blog-3516770-1436708.html
上一篇:Symmetry:聚焦对称/非对称现象研究
下一篇:Thermo期刊正式被ESCI收录 | MDPI News