博文
综述|植物泛组学:方法、应用与进展
||
随着越来越多的植物基因组的组装,人们发现一个单一的参考组装并不能反映一个物种的基因多样性。泛基因组反映了基因组中的结构变异和多态性,从而能够深入比较物种水平或更高分类水平中的变异。本综述讨论了当前的关于泛基因组整合、分析和可视化的方法。此外,也探讨了泛基因组在作物改良、进化和生物多样性研究方面的应用潜力。为了充分利用泛基因组的价值,需要整合更广泛的信息,如表型、环境和表达数据,以深入了解基因组中可变区域的作用。
物种内有广泛的基因组多样性,泛基因组就是需要捕获这种多样性,同时去掉冗余的部分,生成一个整合的单个文件。泛基因组一般包括核心部分和可有可无的部分,核心部分存在于所有个体中,而可有可无的部分只存在于某个个体中。
DNA测序方面的进展使越来越多的基因组得以组装,而且随着基因组测序的不断进展,有机会应用泛基因组来探索物种内部和跨物种的适应和进化机制,从而深入了解基因组的功能以及应用在作物育种上。
在一定的时间尺度内,植物基因组是动态变化的,通常经过多轮全基因组复制或亚全基因组的分化。基因串联复制、转座子活性、缺失、基因组重排和重组等机制在植物基因组变异中也发挥着重要作用。这些可导致基因存在/缺失变异(PAV)和结构变异(SV)。
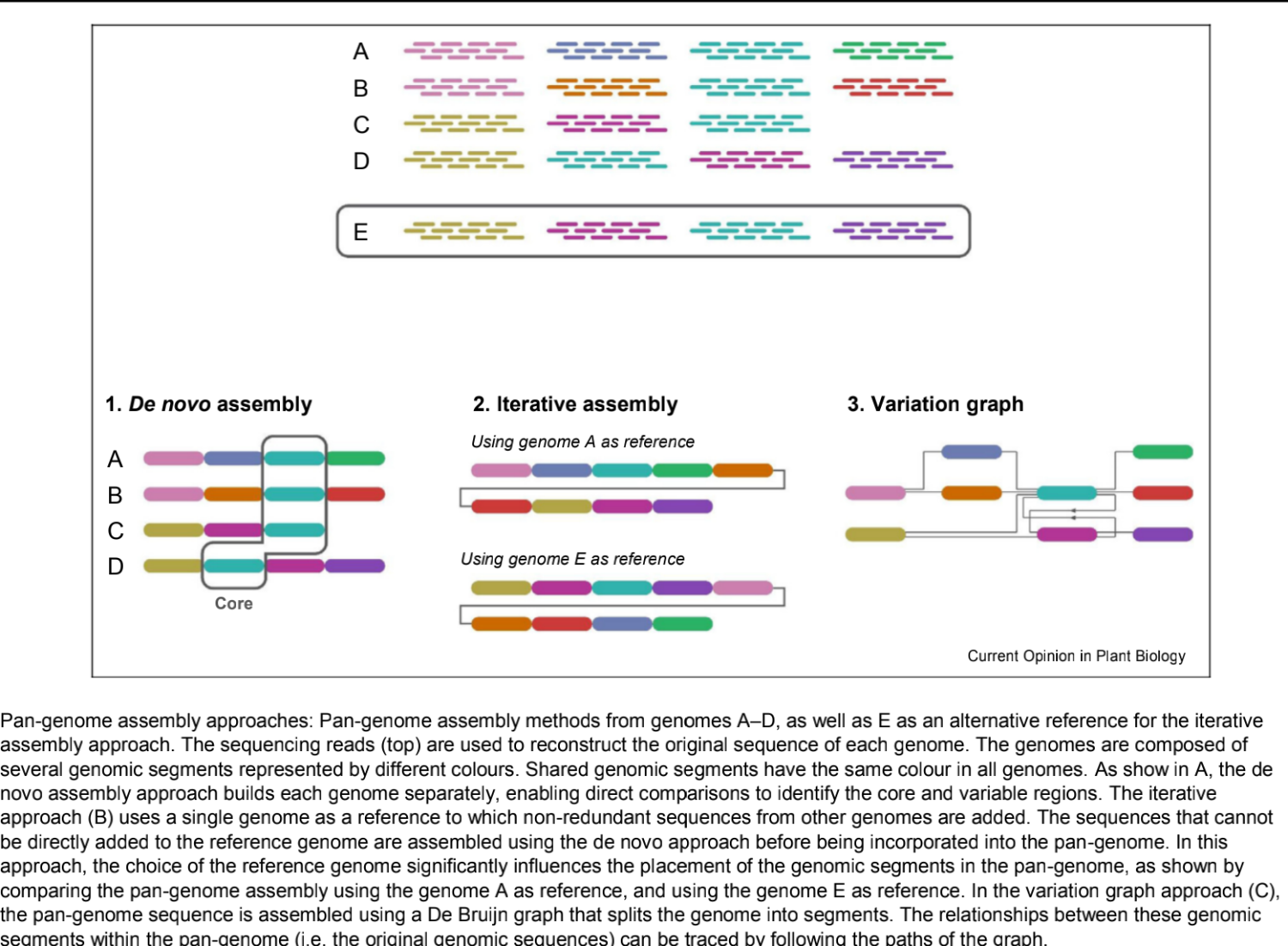
泛基因组组装方法
由于存在大量的重复序列和多倍体,植物基因组和泛基因组的组装常常具有挑战性。传统的从头组装和比较 方法证明了个体之间的基因组差异巨大。 尽管成本相对较高、且提供的结果精确性有待商榷,但它的好处是提供了基因和其他基因组元件的物理位置。相比之下, 迭代mapping和组装方法从单个参考基因组开始,然后用其他个体的非冗余序列对其进行顺序更新,最终构建成一个泛基因组。这种方法允许使用大量的测序覆盖率较低的个体,通过重新mapping至原始基因组来获得存在/缺失变异(PAV)。这两种方法的优缺点使它们具有很强的互补性。少数高质量的从头组装的基因组与 具有10 倍左右的覆盖率的大量个体的组合将允许可变捕获稀有基因,评估大群体中的存在/缺失变异(PAV)等。
迄今为止,大多数的植物泛基因组都是使用Illumina公司的短读数据产生的,这主要是因为其较低的错误率和较少的花费。然而, 在长/超长测序技术方面的快速进步可能会导致泛基因组构建方式的重大改变,因为这种长测序技术能够跨越基因组复杂的重复区域和检测大于500bp的结构变异(SV),尽管目前来看,这种长测序技术通常有较高的错误率。基于短读长和长读长的混合方法可以克服单独使用一种数据类型的缺点。
最近,双向变异图(VG)已经被用于泛基因组构造。双向变异图(VG)使用线性基因组所支持的测序读取来生成可变区域的图表,避免了装配偏好性(下图)。双向变异图(VG)可更有效地解决复杂的结构变异,因为变异的位置具有更广泛的图路径。该算法允许不断增加新的测序数据。目前,双向变异图(VG)在植物中的应用受到植物基因组结构复杂性的限制。还缺少一个用于下游分析和可视化的工具。然而,考虑到双向变异图(VG)所提供的优势,以及双向变异图(VG)工具开发的最新进展,这些限制很可能在未来得到解决。
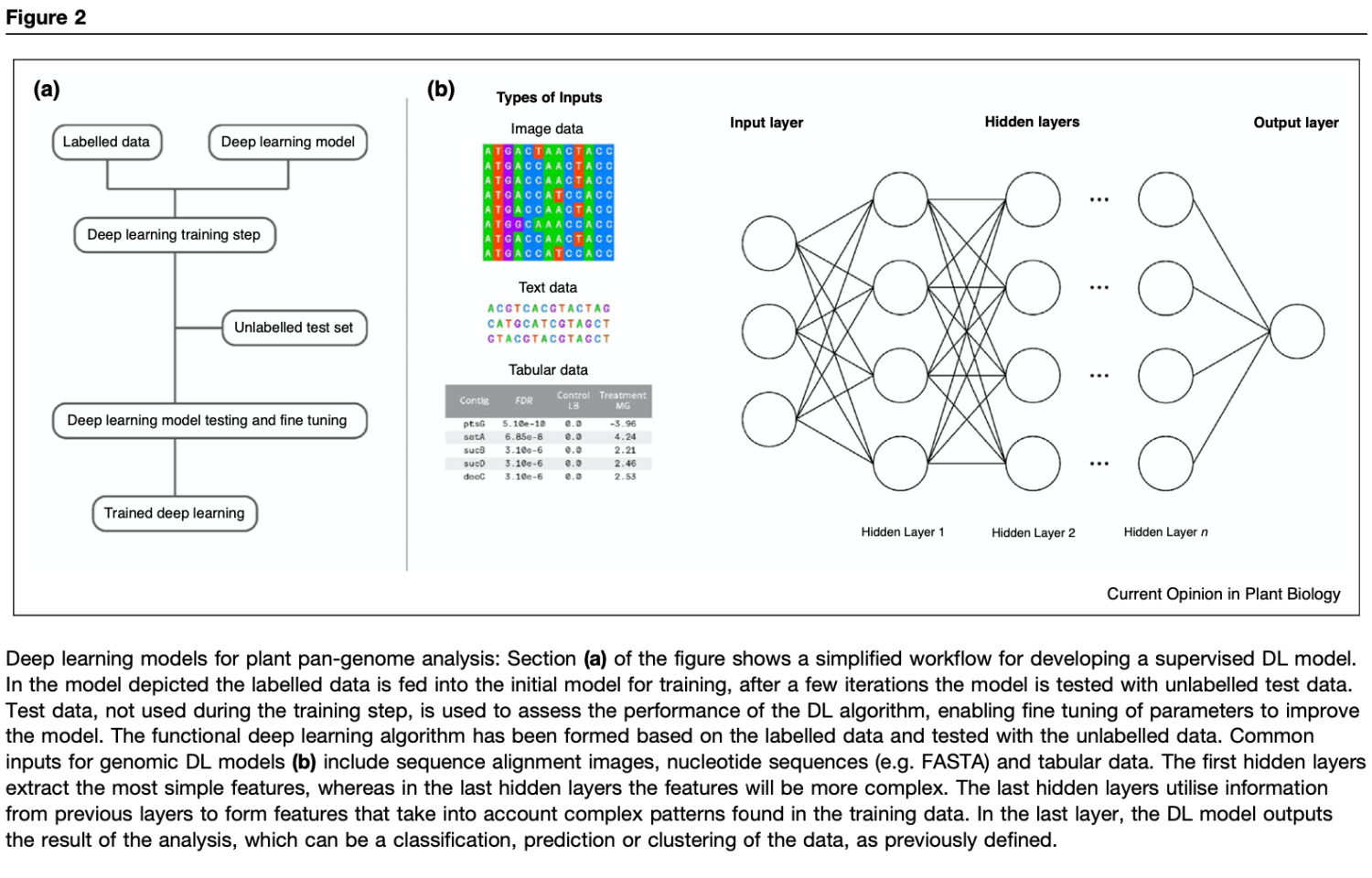
泛基因组组装的深度学习工具
机器学习正越来越多地应用于植物基因组分析。机器学习方法通常先有一个可靠的数据集,例如目标基因组区域的明确变异信息,要求研究者非常熟悉这些内容。相比之下,深度学习模型可以自主地检测和学习样本数据中的模式,需要输入的数据较少,但仍然需要数据预处理和专业知识建立一个合适的架构来解决问题。深度学习具有利用大型复杂数据集的潜力,基于广泛的统计方法生成准确的分类、预测或聚类输出。
基于深度学习变异检测的工具在生物医学的研究中已经成功实现,并且可以超越现有的工具。深度学习模型也已用于基因保守性和表达分析,CRISPR-Cas9脱靶预测和蛋白质分析。数据挖掘模型的训练需要大量经过处理和注释的数据,这限制了数据挖掘模型在植物泛基因组组装和分析中的应用,因为数据挖掘模型需要对样本进行精确的标记,以了解目标的特征。这一步骤目前还不能用于基因组数据模型,但是随着植物高质量的基因组和注释数据的不断增加,应该很快就能解决这一限制。
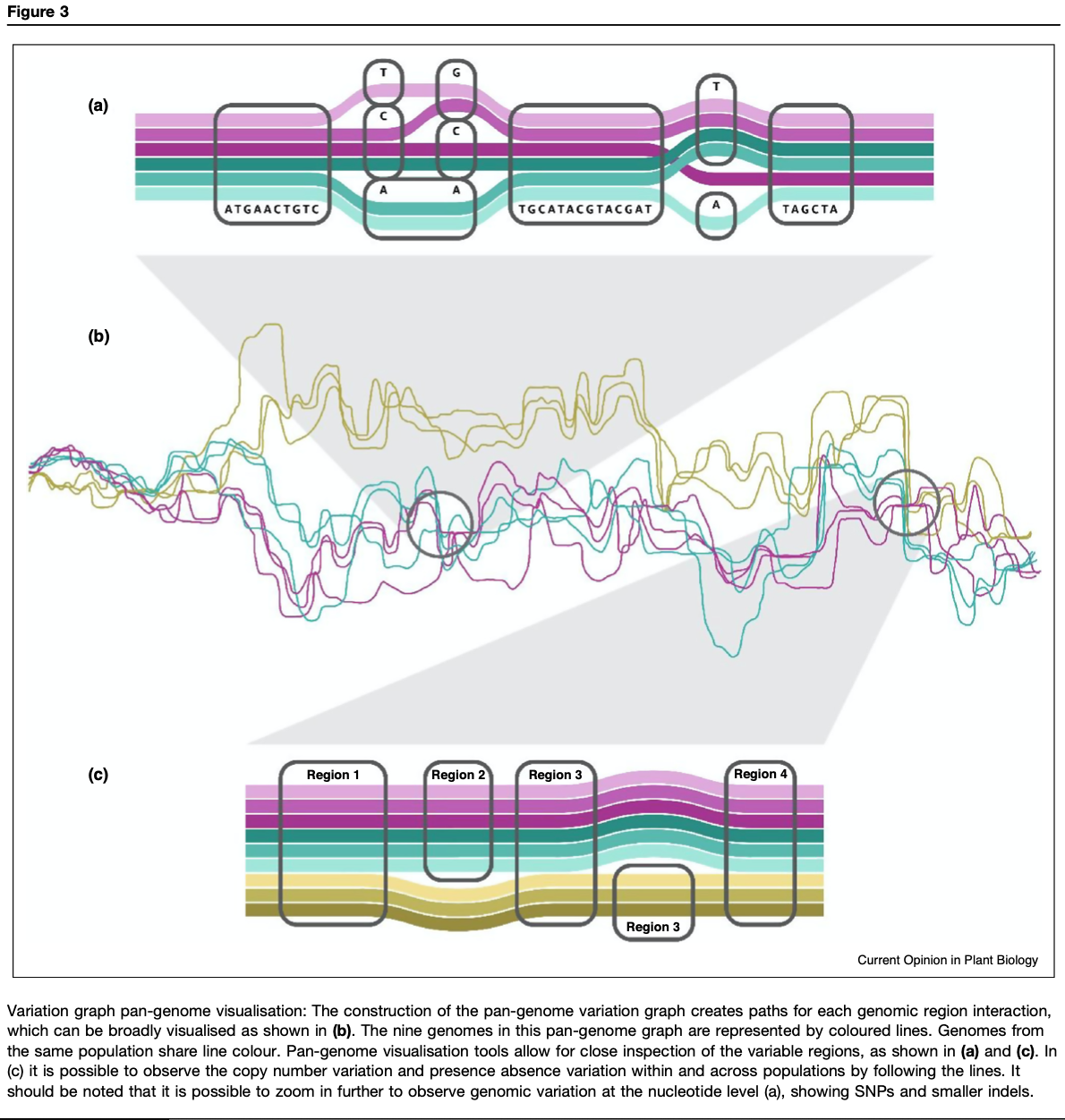
泛基因组的可视化
可视化工具已经被开发用于泛基因组,虽然其中一些可以适应于植物,但很难为大规模的样本设计可伸缩的解决方案。大多数为泛基因组开发的可视化工具要么基于参考基因组结构,要么需要用双向变异图(VG)从头组装,以便正确显示可有可无的区域和位置。例如,ppsPCP和CoGe 使用组装好的植物基因组进行比较,然后使存在/缺失变异(PAV)等可视化。一些植物泛基因组的研究已经发布了专门用于数据可视化的工具, 例如 RPAN 有 3000 个水稻基因组,GBrowse 有 16 个小麦品种,Brachypan 有 54 个二穗短柄草。这些工具可以在泛基因组数据库中搜索基因和可视化,提供基因组序列、基因注释、PAV信息和基因表达信息的访问。开发用于其他物种的可视化工具是必要的,可以促进更深入的综合分析和用于作物改良。
植物基因组的复杂性对可视化工具的开发提出了重大的计算挑战。为了克服这种局限性,一些泛基因组的研究人员采用了基于云的解决方案,以方便获得用于泛基因组操作和可视化的工具。云在这里被定义为一种通过互联网提供服务的基础设施。云数据库在高性能计算机中提供数据存储、管理以及软件维护,增加了可重复性。云“公共”环境,如为生物医学合作开发的环境,可以促进植物多组学和辅助数据的整合,如地理、表型、基因表达和功能数据。建立一个基于云计算的植物泛基因组中心也可以促进研究人员之间的数据和分析共享,通用标准方法的建立和使用使得泛基因组合作分析成为可能。
泛基因组在作物改良中的应用
基因组信息正越来越多地应用于作物育种;然而,少数的参考基因组并不能代表所有的种质,泛基因组在作物改良的应用中具有重要作用。泛基因组可以用来绘制非必须的基因组序列,突出显示哪些种质的基因提供了优异的农艺性状。作物那些非必须的基因组通常与农艺性状有关,如开花时间、病害抗性和环境胁迫反应等。作物的育种过程引起了遗传多样性的减少,特别是与重要性状连锁的基因,如抗病基因等。泛基因组可以更好地支持和研究基因存在/缺失变异 (PAV) 在作物育种中的意义。识别在育种和选择过程中丢失的优异基因,可为后续培育优异种质打下坚实的基础。对一个物种中非必须基因组的识别有助于了解哪些基因组变异可能与农艺性状有关。通过将SNP标记、泛基因组水平的 SNPs 以及存在/缺失变异 (PAV) 应用在关联分析中,可改善关联分析中优异基因的鉴定能力。在基因编辑领域的发展有可能彻底改变作物育种方法,使其从传统的杂交和选择转向基因组编辑和选择。基于基因编辑的育种应用需要对目标基因有充分的知识。
跨物种的泛基因组及其进化研究
虽然迄今为止大多数泛基因组都是在物种内构建的,但可以在更高的植物物种分类水平上构建泛基因组。这有助于鉴定单个物种特异的基因以及共有基因。这为生物学家提供了从基因组学角度探索如何定义一个物种的机会。在某些情况下,这可能导致分类学上的不一致性,但这种不一致性随后可以得到解决,就像在细菌泛基因组组学中所做的那样。
植物基因组是高度动态的,基因大小的变化导致基因家族的扩增和通过基因融合产生新的基因,所以基因组结构变异在必须基因和非必须基因中的作用可为理解基因进化提供了一个有价值的途径。泛基因组支持对基因进化历史的分析,包括同源基因的鉴定和基因家族的分析等。这些信息对于理解基因功能很重要,可以为我们将基因从一个物种转换到另一个物种提供研究基础。了解基因序列的跳跃性以及整个基因组进化的知识也可以支持对基因组更精确的自动注释,从而将基因可与表型更准确地联系起来。
挑战与未来展望
随着诸如“10KP 基因组测序计划”的开展,建造泛基因组正变得越来越具有吸引力。然而,未来的研究需要新的工具来支持泛基因组的组装、整体结构的可视化等。虽然泛基因组具有提高作物育种效率的潜力,但未来的工具和数据库必须将泛基因组与基因功能分析和进化重建结合起来。需要一套能够整合不同组学数据的泛基因组工具来解析基因组变异(包括非编码区)和复杂农艺性状之间的关系。随着作物基因编辑技术的不断进化和变得更加广泛,基因组编辑在作物改良将扮演着重要的角色。
为了能够快速有效地构建精确的泛基因组,需要与其它可用的功能数据结合起来,但这还有一些挑战有待解决。长读长序列的加入可以帮助解决重复序列和多倍体基因组难以用短序列组装的问题,随着数据准确性的提高和成本的降低,这种方法很可能主导植物泛基因组学研究。深度学习在泛基因组研究中的应用也可能有助于克服泛组装和功能研究的一些挑战。
虽然许多主要农作物现在都有泛基因组,但完整性各不相同,将其扩展到野生物种中使其在作物改良中得到更多的利用。一旦泛基因组可用于大量不同的物种,我们就可以开始了解物种是如何在基因组水平上定义的,为植物进化和多样化研究提供深入的见解。
https://blog.sciencenet.cn/blog-1094241-1224966.html
上一篇:综述|研究小麦粒重和产量的一种简化方法
下一篇:GATK流程不用再分割小麦染色体