博文
生命是机器么
 精选
精选
|
最近读到一篇不错的文章,题目叫"Is the cell really a machine?"[1],很有启发性。作者主要探讨了还原论方法解释生命问题的局限性,不过具体该怎么做,还不是很清楚。
下面这张图是13年前的图了[2],这张图我在报告里面用过,其类似的版本很多人也用过。
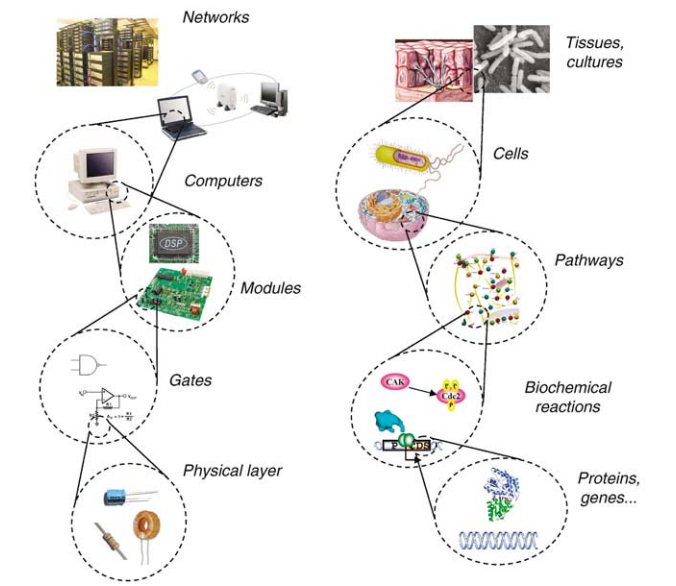
仔细推究的话它其实有很多问题,随手就可以举出,比如生化反应的随机性,生物网络的无尺度性等等。从工程的角度区别就更大了。电子工程是可以scale-up的,现在元件晶体管数目都很庞大。生物元件基本不能scale-up,现在能够实现的合成回路的复杂度仍然处于比较低的水平。信息产业花了半个多世纪从三极管做到了互联网,右边这条线能不能有类似的进度,有点让人怀疑。
反过来讲,这种类比也有一定的价值。从研究对象的复杂程度来讲,二者确有一些共通之处。我有时会思考一个问题,如果把一台工作中的计算机扔到中世纪,甚至扔给现在的小学生,假设这台计算机不会坏掉,他们会怎样研究它,能不能搞清楚计算机的工作原理(就对生命的认知而言,说我们还是小学生似乎也并不为过)。可能他们的研究方法听起来会有点奇怪,没准我们现在一些研究生命问题的方法也是这样。拿测序技术来说,有一段时间大家似乎觉得测序翻开了生命设计的大书,可以解决几乎所有问题,当然后来发现并不是这样。跟计算机类比一下,假设DNA作为存储介质就是电脑硬盘,那基因测序类似把硬盘上所有0/1读出来。虽然理论上知道所有0/1好像可以把操作系统还原出来,但若不知道编程语言,没有软件工程的知识,这些数据也无法解读。类似的我们也有RNA测序,可以粗糙的类比为把内存中的0/1读了出来。而现在对这些数据的分析还很粗糙,多数只看一下哪些基因上调了,哪些基因下调了。类似于观察到系统启动过程中内存一些区块变成了0,一些区块变成了1。这些信息又该如何利用。
还有一些相对“传统”的方法。比如说基因敲除看致死基因,类似于在windows的源码里删掉一行代码/函数,看系统还能不能启动。功能性缺陷,相当于删掉一行代码,看某个软件能不能打开。我们可以知道几百个基因是致死的,哪些基因跟功能相关。这固然是有用的信息,但进一步似乎就无能为力了。我想如果去微软面试,面试官应该不会问这种问题“windows删掉哪些源码就不能启动了,它们有什么共通之处”,而应该考一些基本的编程,数据算法。
所以我的看法是,对于生命问题,我们的方法论(methodology)其实是远远不足。即使把生命体系,比如一个细胞的复杂程度等同于计算机这种人造的,有着明确逻辑和构建方法的系统。我们都很难利用现有的方法推出其构建的原理。比较糟糕的是,有时候人们会把方法论的进步等同于获得数据/拟合数据能力的进步。似乎方法学就是用最新的,别人都没有的设备,获得最多的数据。这可能只是问题的一面。如何利用这些数据,如何用新的理解来指导新的方法,可能是生命科学需要解决的一个问题。从“观测”计算机到造出计算机,还有漫长的道路。
[1] https://www.sciencedirect.com/science/article/pii/S0022519319302292?dgcid=author
[2] http://www.ncbi.nlm.nih.gov/pubmed/16738572
https://blog.sciencenet.cn/blog-927304-1188406.html
上一篇:四大文明古国还是三大文明古国
下一篇:质粒拷贝数以及启动子活性的定量测量