博文
使用图进行多模态学习
||
使用图进行多模态学习
基于图的深度学习为生物学、化学、物理学和社会科学的突破做出了贡献。图神经网络的主要用途是学习基于神经信息传递策略的各种图组件的表示,例如节点、边、子图和整个图。学习到的表示用于下游任务,包括通过半监督学习进行标签预测、自监督学习、图设计和生成。在大多数现有的应用程序中,数据集以节点、边和表示上下文知识的附加信息形式(如节点、边和图属性)来描述图。
对复杂系统进行建模需要从不同角度、不同尺度或通过多种模式(如图像、传感器读数、语言序列和紧凑的数学陈述)描述相同对象的测量。多模态学习研究如何优化这些异构、复杂的描述,以创建具有广泛泛化、对底层数据分布的变化具有鲁棒性的学习系统,并且可以用更少的标记数据进行更多的训练。虽然多模态学习已经成功地应用于单模态方法失败的环境中,但要使其在人工智能中得到广泛应用,必须克服几个挑战。这些挑战包括为机器学习分析寻找优化的表示,以及融合来自各种模式的组合信息以创建预测模型。这些挑战已被证明是困难的。例如,多模态方法倾向于只关注在模型训练过程中最有帮助的模态子集,而忽略了可能对模型实现有用的模态——这是一个被称为模态崩溃的陷阱。此外,与每个对象必须以所有模态存在的常见假设相反,由于数据收集和测量技术的限制,几乎无法获得完整的模态集-这是一种被称为缺失模态的挑战。由于不同的模态可能导致复杂的关系依赖,简单的模态融合不能充分利用多模态数据集。图学习模型,如数据系统,通过连接不同模式的数据点作为最优定义图中的边,并为广泛的任务构建学习系统。
Ektefaie等人介绍了一个多模态图学习(MGL)的蓝图。MGL蓝图提供了一个框架,可以表达现有的算法,并帮助开发多模态学习的新方法利用图表。这个框架允许学习融合图表示,并研究前面提到的模态崩溃和缺失模态的挑战。Ektefaie等人将此范式应用于广泛的领域,从计算机视觉和语言处理到自然科学(图1)。考虑用于图像和视频推理的图像密集型图(image-intensive graphs,IIGs)(“用于图像的MGL”),用于处理自然和生物序列的语言密集型图(image-intensive graphs,LIGs)(“用于语言数据集的MGL”)和用于帮助科学发现的知识密集型图(image-intensive graphs,KIGs)(“用于自然科学的MGL”)。
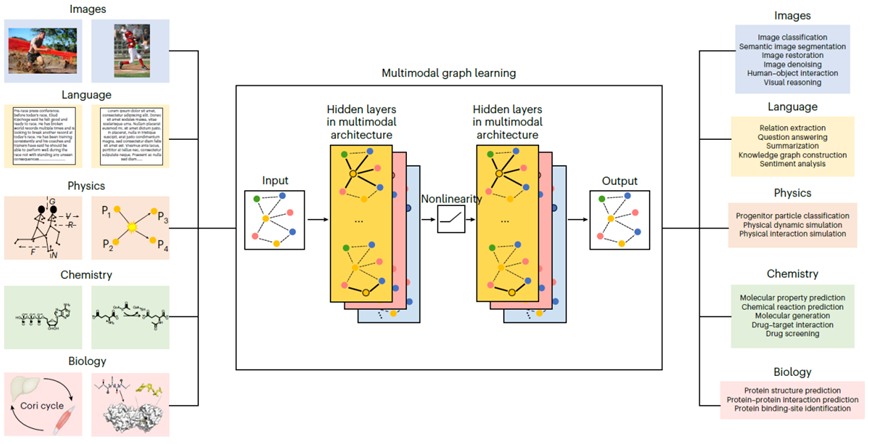
图1 以图为中心的多模态学习。左边显示的是不同的数据模式。右图显示的是MGL证明有价值的机器学习任务。MGL蓝图为计算机视觉、自然语言处理和自然科学的学习系统实现的多模态图神经架构的统一框架。
使用GNN(图神经网络)进行多模态学习是有吸引力的,因为它们可以灵活地模拟不同数据类型内部和跨数据类型的交互。然而,通过图学习进行数据融合需要构建网络拓扑,并在图上应用推理算法。给定一组多模态输入数据,MGL产生用于下游任务的输出表示。MGL可以看作是由四个学习组件组成的蓝图以端到端方式连接。在图2a、b中,强调了用于处理多模态数据的传统单模态架构组合与建议的一体化多模态架构之间的区别。MGL的前两个组成部分,识别实体和发现拓扑,可以归类为结构学习阶段(图2c)。
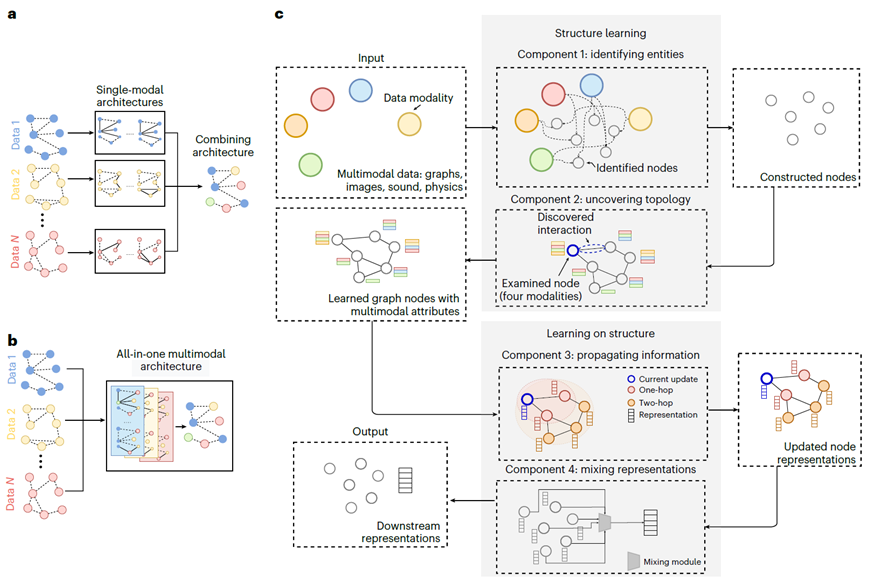
图2 MGL蓝图概述。a、多模态学习的标准方法包括组合不同的单模态架构,每个架构都针对不同的数据模态进行优化。b、相比之下,一体化多模态架构考虑了针对每种数据模态的归纳偏差,并以端到端方式优化模型参数,从而实现了表达性数据融合。c、MGL蓝图包括四个部分:识别实体、发现拓扑、传播信息和混合表示。这些组件分为两个阶段:结构学习和对结构的学习。
MGL是一个新兴领域,其应用跨越自然科学、视觉和语言领域。MGL的增长将受到完全多模态图架构以及自然科学和医学领域新应用的推动。Ektefaieden等人还概述了应用程序,以了解MGL何时有价值或没有帮助,以及需要改进以解决由多模态归纳偏差或缺乏显式图所代表的挑战。详细的情况可以参考文献[1]。
参考文献
[1] Ektefaie, Y., Dasoulas, G., Noori, A. et al. Multimodal learning with graphs. Nat Mach Intell 5, 340–350 (2023). https://doi.org/10.1038/s42256-023-00624-6
以往推荐如下:
5. EMT标记物数据库:EMTome
8. RNA与疾病关系数据库:RNADisease v4.0
9. RNA修饰关联的读出、擦除、写入蛋白靶标数据库:RM2Target
13. 利用药物转录组图谱探索中药药理活性成分平台:ITCM

https://blog.sciencenet.cn/blog-571917-1391088.html
上一篇:shinyCircos v2.0:Circos图创建和可视化工具
下一篇:肿瘤免疫治疗基因表达资源:TIGER