博文
[转载]iqtree建树命令
|||
系统发育推断(phylogenetic inference)的算法五花八门,从最简单的UPGMA法,到邻接法(neighbor joining)、最大简约法(maximum parsimony),再到复杂的的最大似然法(maximum likelihood)与贝叶斯推断法(Bayesian inference),每种方法都有不少可选择的实现工具。这些方法无一例外都遵循一个规律:越精确则速度越慢。而随着分析的数据越来越庞大,对于用户来说,需要寻找一个尽可能快速而且错误率可以容忍的工具是十分必要的。
原文链接:
mp.weixin.qq.com/s/mzAtGnai3KcI5WtA52nqtw
贝叶斯法可以准确地推断时间树,但速度最慢;而邻接法与最大简约法在处理有较大分歧的序列时容易出现长枝吸引(long branch attraction,LBA)现象,因此目前来说最大似然法还是最常用的方法。目前实现最大似然法建树的工具有MEGA、PhyML、RaxML等,然而这些软件参数设置十分复杂,尤其是核苷酸与氨基酸替代矩阵的选择往往对结果的准确性有着很大的影响,而普通用户往往难以选择。而且,这些工具的分析速度也较慢。
近几年悄然兴起一个新的系统发育推断工具Iqtree(http://www.iqtree.org/),其简化了参数设置,可以帮助用户选择最佳的进化模型,而且在速度上有很大提升。最新版的是Iqtree2,其安装方法如下所示:
tar -zxvf iqtree-2.0.6-Linux.tar.gz
cd iqtree-2.0.6-Linux
解压后iqtree执行文件就在bin/目录下,其使用方法如下所示:
iqtree [-s ALIGNMENT] [-p PARTITION] [-m MODEL] [-t TREE] ...
-s:序列比对文件(支持多个文件逗号隔开,或者包含比对文件的文件夹),可选PHYLIP、FASTA、NEXUS、CLUSTAL、MSF
--seqtype:序列类型,可选BIN、DNA、AA、NT2AA、CODON、MORPH默认为自动检测
-o:外类群列表,不同物种之间逗号隔开
--prefix:结果文件名前缀
--seed:随机数种子,主要出于调试目的
--mem:最大可使用内存,单位为G、M或百分数%
--redo:忽略检查重写输出文件,默认为off,也即从上次意外中断处开始
-T:程序运行使用的核数,可设置具体数字或者AUTO(推荐),默认为1
--threads-max:最大可使用的核数,默认为所有核
--fast:快速模式,类似FastTree
-b:非参数bootstrap次数,大于等于100
-B:超快速bootstrap次数,大于等于1000
--bnni:使用NNI优化超快速bootstrap的树,搭配-B使用
--alrt:SH近似似然比检验重复次数
-m:模型选择,设置MF自动选择最佳模型但不建树;设置MFP自动检测最佳模型并建树。此外还可以设置具体的模型,或者多个可选模型,例如-m LG,WAG
--ancestral:基于经验贝叶斯的祖先状态重建
接下来看这个工具如何使用。首先从最简单的建树开始:
iqtree -s example.phy -T AUTO
假如设置自动选择最佳模型并建树:
iqtree -s example.phy -m MFP -T AUTO
选择最佳模型并只输出模型选择结果:
iqtree -s example.phy -m MF -T AUTO
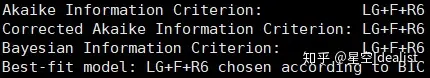
Iqtree会测试多达546个蛋白模型并给出最佳模型,结果如下所示:
使用bootstrap自助法计算节点支持率:
iqtree -s example.phy -m MFP -b 100 -T AUTO
使用SH近似似然比检验计算节点支持率:
iqtree -s example.phy -m MFP --alrt 100 -T AUTO
同时使用两种方法计算节点支持率:
iqtree -s example.phy -m MFP --alrt 100 -b 100 -T AUTO
使用超快速bootstrap自助法计算节点支持率:
iqtree -s example.phy -m MFP -B 1000 --bnni -T AUTO
使用上述设置构建500个基因组的120个串联蛋白树需要两天左右。超快自助法ultrafast bootstrap1000次比普通自助法100次要快10倍左右,是该软件的特有算法,所以一般使用Iqtree的超快自助法建树。
原文链接 https://zhuanlan.zhihu.com/p/408382758
https://blog.sciencenet.cn/blog-3434047-1389290.html
上一篇:paml正选择——codeml初学者指南
下一篇:拟合度卡方检验——摘抄《应用数理统计方法》_陶澍