博文
哈佛医学院王寿文等开发CoSpar,突破谱系追踪数据的分析瓶颈,成功预测细胞命运
||
2022年2月21日,美国哈佛医学院王寿文博士(第一作者和共同通讯)和Allon Klein教授 (共同通讯)合作在Nature Biotechnology报道开发了Coherent Sparse optimization(CoSpar)。
CoSpar通过结合单细胞转录组数据和克隆谱系信息,可以很准确地预测细胞的命运选择。这为系统地研究不同体系里体内和体外的细胞分化清除了障碍。

生物学中一个重要命题是细胞如何选择命运。这对于理解组织的修复,胚胎的发育,癌症的发生都非常关键。此外,近年来发展的细胞重编程等改造细胞状态的技术也需要对细胞分化有深刻的理解。细胞命运的研究由来已久。由于技术上的限制,以前的研究大多局限于几个特征基因的表达来刻画细胞命运的选择。由于分辨率不高,细胞被认为有不同的类型(cell type),细胞分化就是从一个细胞类型,跳到另一个类型,中间缺乏连续的过渡态。随着近几年来高通路单细胞测序技术的发展,我们已经可以轻松地从一次实验中获得成千上万个细胞的转录组。大家越来越意识到之前认为的细胞类型实际上是由很多不同的细胞状态(cell state)组成的;而细胞分化很多时候是从一个细胞状态连续变化到另一个状态。从数学的角度讲,一个高维空间上的连续曲面(细胞分化真实的状态)在低维坐标的投影(只考虑几个特征基因)可能会严重扭曲这个曲面,造成错误的印象。
如果能获得在转录组的高维空间中细胞分化的真实轨迹,那就能够对细胞分化的过程有一个更加系统性的理解。它能帮助我们更加准确地找到细胞命运选择过程中的标志基因和找到控制细胞命运的调控基因,也有助于系统地构建细胞命运的基因调控网络。知道分化轨迹可以帮助我们更加合理地设计细胞重编程等细胞改造技术的实验步骤,甚至可以帮助我们加深对于癌症的理解。如何系统地从高通量的单细胞数据中推算出细胞分化的真实轨迹是一个很有挑战性的工作。目前有很多算法试图直接从单细胞转录组数据预测细胞的命运。然而单细胞转录组数据本身不存在单个细胞的动态信息,因为测序需要把细胞杀死。因此,直接从单细胞转录组数据预测细胞命运就好像试图从一张静止的照片推测里面人物的长时间运动轨迹,这必然非常有挑战性。最新的研究暗示仅仅依赖单细胞转录组是不够的。整合新的信息,才有可能获得突破。
细胞谱系追踪历来都是研究细胞命运选择的标准手段。谱系追踪通过标记细胞去追踪单个细胞以及它的后代的分化轨迹。一个经典的例子是通过显微镜追踪秀丽隐杆线虫(C.elegans)整个细胞家谱图。这个工作发现了细胞凋亡在发育中的独特作用,被授予2002年诺贝尔奖。最新的谱系追踪技术通过往细胞的DNA中插入一小段随机的DNA,或者一段可以快速且随机突变的DNA,去追踪每个细胞分化,然后利用测序技术读取每一个细胞的谱系或者细胞克隆(clone)的身份,从而知道哪些细胞来自于共同的祖先。这样的技术可以同时追踪成千上万个细胞的谱系以及他们各自的命运选择。更重要的是,它可以和单细胞测序结合在一起,既获得每个细胞的谱系的信息,也拿到转录组的数据。这项技术已经被成功应用于研究许多生物体系,其中的发现包括造血干细胞的调控基因,和细胞重编程中的关键基因等。
然而谱系追踪数据分析起来非常有挑战性。首先,实验中我们往往只能获得某一个时刻的细胞谱系信息,而不能追踪某个细胞克隆在不同时间的情况(挑战1;图1a)。这就回到了我们一开始的问题:如何从静态的数据中推测动态的演化。对于一些体外培养的细胞分化系统,比如细胞重编程,我们可以绕过这个困难:对细胞做标记后,等它分裂产生了多个子细胞,每隔一段时间随机取一小部分细胞测序,读取克隆身份和转录组信息。这样,我们就有可能在一个克隆的层面上,获得细胞分化的近似轨迹。然而这样的实验必须精心设计,尤其是第一次采样的时间点。如果采样太早,那时一个克隆里的细胞数目很少。要么碰巧都没有被采样,从而失去了早期的信息;要么集体被拿去测序,那就无法获得后续的信息了。如果采样太晚,那克隆内部的细胞与细胞之间可能就已经产生了很大的差异,得到的不是一条轨迹,而更像是许多各不相同的轨迹的杂乱叠加(挑战2;图1b)。比起一条清晰的分化轨迹,这样的数据更像是对于细胞群体在不同时间点的采样,分析起来很有挑战性。除了以上列出的2个挑战,谱系追踪的数据还可能存在别的难题:3)不同克隆的细胞数目差异很大(图1c);4)测量时损失了克隆里的一些细胞(图1d);5)不同的克隆可能恰好拥有相同的标记DNA,因此被错误地拼接成一个克隆(图1e)。这些困难都是目前细胞谱系追踪所无法避免的,因此只能通过开发更好的计算方法去克服。这样的方法对于更好地解释谱系追踪的数据和简化实验设计等,都会有很大的帮助,从而推动谱系追踪技术被更广大可科研群体使用。
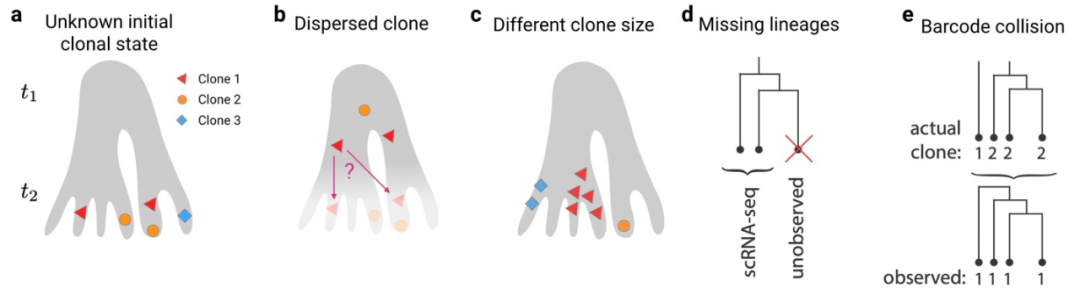
图 1. 分析细胞谱系追踪数据需要面对的5大挑战。
王寿文博士和Allon Klein教授共同开发了Coherent Sparse optimization (CoSpar )来系统地应对这五个数据分析的挑战。CoSpar考虑了三种实验设计:1), 有细胞克隆在不同时间点的谱系和转录组信息;2)虽然只有细胞克隆在末态的谱系和转录组信息,但有初始细胞群体的转录组信息;3)只有初始和末态细胞群体的转录组信息。CoSpar的目标是从这三种不同的数据类型中推算出实验体系的细胞从任何一个初始细胞状态分化到一个末态的概率。数学上,这就是一个从初态到末态的跃迁矩阵。这个跃迁矩阵可以系统地捕捉细胞分化的动力学过程,揭示早期每个细胞可能有的命运,并发现早期细胞可能潜在的命运偏好。我们的算法的核心是在满足看到的细胞克隆数据的同时,试图找到一个稀疏的跃迁矩阵,又使相似的初始细胞有类似的命运选择。本质上,就是在寻找满足观测数据,最吝啬又有局域一致性的跃迁矩阵。
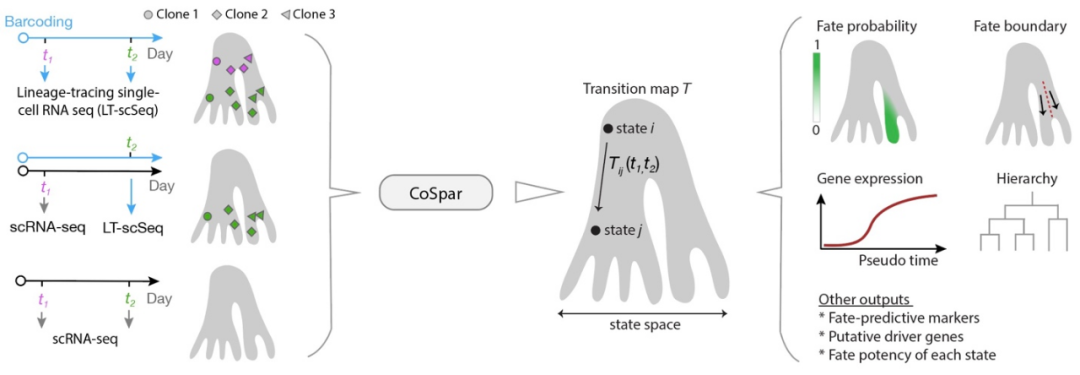
图 2. CoSpar的工作流程图。它可以接受三种不同的实验设计,从中推测从任何一个细胞初态到细胞末态的跃迁几率,并用于下游的分析。
CoSpar被应用于已经发表的三个不同体系的谱系追踪数据:造血系统,重编程,和诱导分化。利用这些数据,作者们首先深度地检验了CoSpar,发现它确实可以成功地应对以上提出的五个挑战,同时也具有很强的鲁棒性:即使只有很少的带有谱系信息的细胞,或者只有很少数目的独立克隆,CoSpar都有很优异的表现。接下来,作者们用CoSpar对每一个实验体系都做了新的预测,并且都找到了细胞早期命运的选择偏好,突破了以往分析方法的局限性。
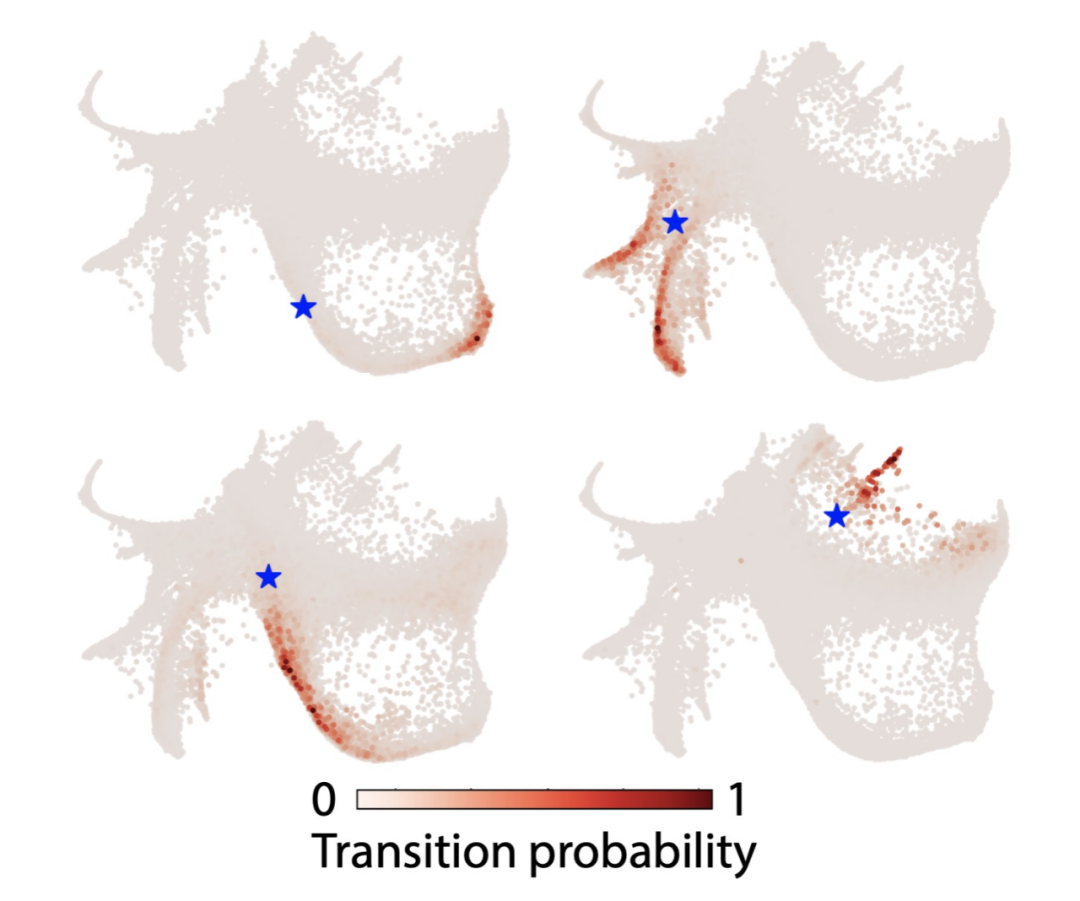
图 3. CoSpar预测造血系统里,不同初始细胞状态在2天后可能分化的位置。
在造血系统里,大家知道Mast cell,Basophil,和Eosinophil有用共同的前体细胞,然而不了解它究竟表达什么样的基因。他们比对了原始论文的分析方法(Weinreb)和CoSpar,发现CoSpar可以更好地找到这个前体细胞在转录组态空间上的位置,同时也可以发现上百个与它相关的差异化基因(Weinreb只能找到4个)。
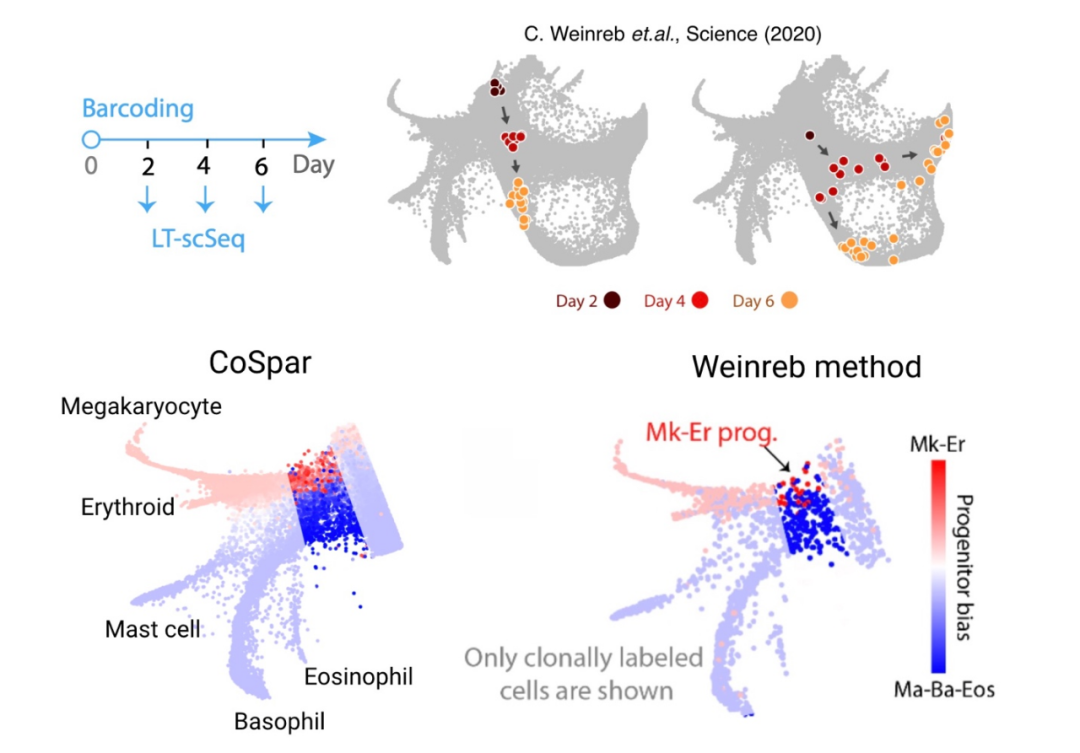
图 4. CoSpar预测造血体系中Mast cell,Basophil,和Eosinophil的前体细胞在转录组态空间上的位置。其准确性高于已有分析方法Weinreb。
在重编程系统实验中,大多数的细胞都无法完成重编程成为多能干细胞,因此一个重要的问题就是找到那些最终会实现重编程的早期细胞,理解其中的机制,从而改进实验设计,提高重编程的效果。遗憾的是,这个实验数据里第三天的细胞群体几乎没有可以检测到的细胞克隆(之后的时间节点有),因此无法直接找到那些潜在的『黑马』。而重编程的结果需要等到第28天才会揭晓。虽然如此,CoSpar惊人地发现细胞的命运偏好在第三天就已经很明显了,并且成功地预测出了有重编程潜力的群体。这个结果进一步被关键的转录因子的表达佐证。值得注意的是,CoSpar构建了一个从第3天到第28天的跃迁矩阵。要知道,时间跨度这么大,这两个细胞群体是很不一样的。这意味这以后优化重编程的实验可以考虑大大缩减需要采样的时间点,从而大大节省时间和尽力。的确,另一个类似的重编程研究需要每12小时采样,才能可靠地估计细胞的分化过程(Schiebinger et.al., Cell 2019)。
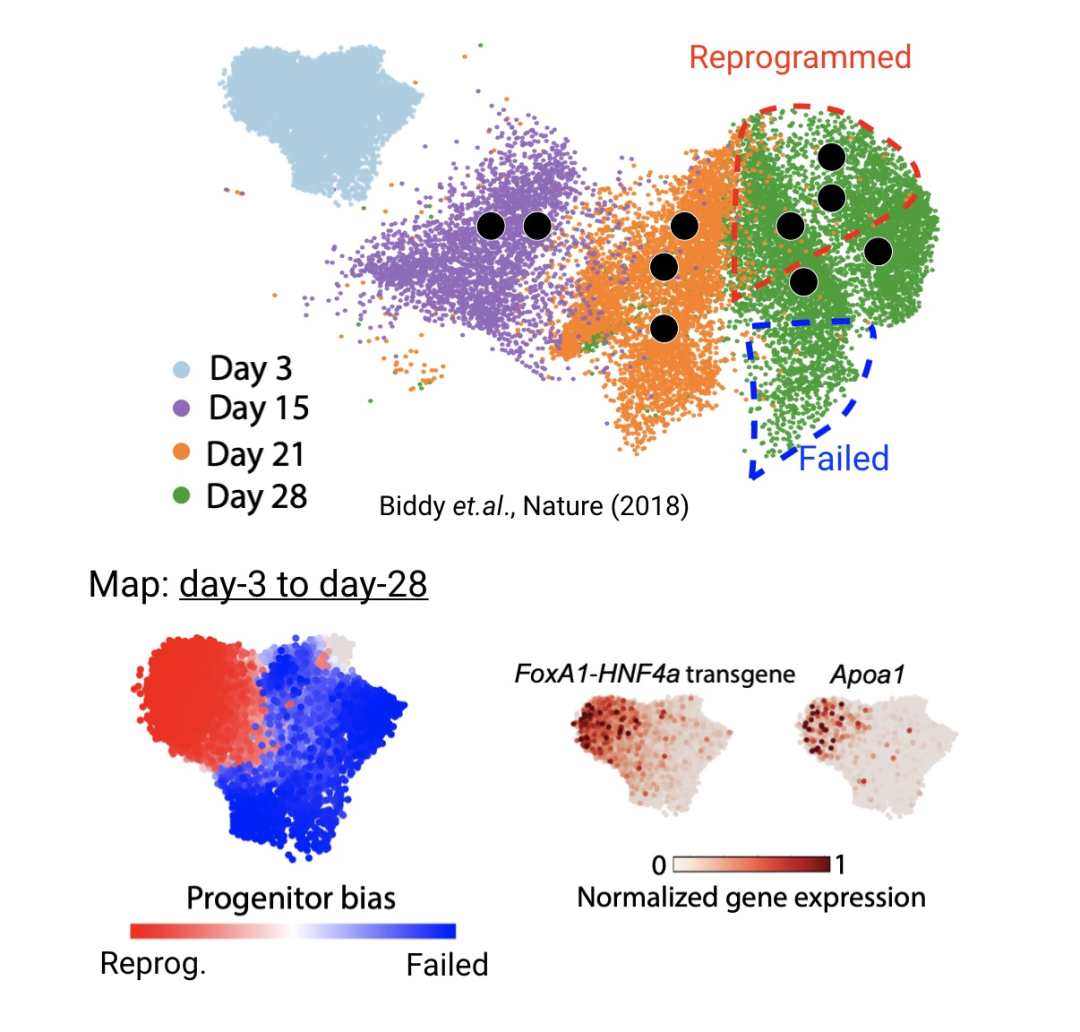
图 5. CoSpar发现在重编程实验中,细胞在第三天就已经表现出明显偏好,并预测了有重编程倾向的细胞的位置。
最后,在诱导分化的实验中,CoSpar也成功发现第17天的细胞有明显的分化偏好,其中一部分细胞会在第27天成为实验人员想要的肺细胞。同样,这里只有第27天有克隆数据。此外,第17天和第27天单细胞数据来自于不同的实验过程,因此存在系统性的实验批次效应。尽管有这些问题,CoSpar依然能很好地应对。CoSpar预测对LIF receptor的刺激可能会大大影响细胞的增值,也可能影响分化过程。王博士们进一步实验验证了这一预言。
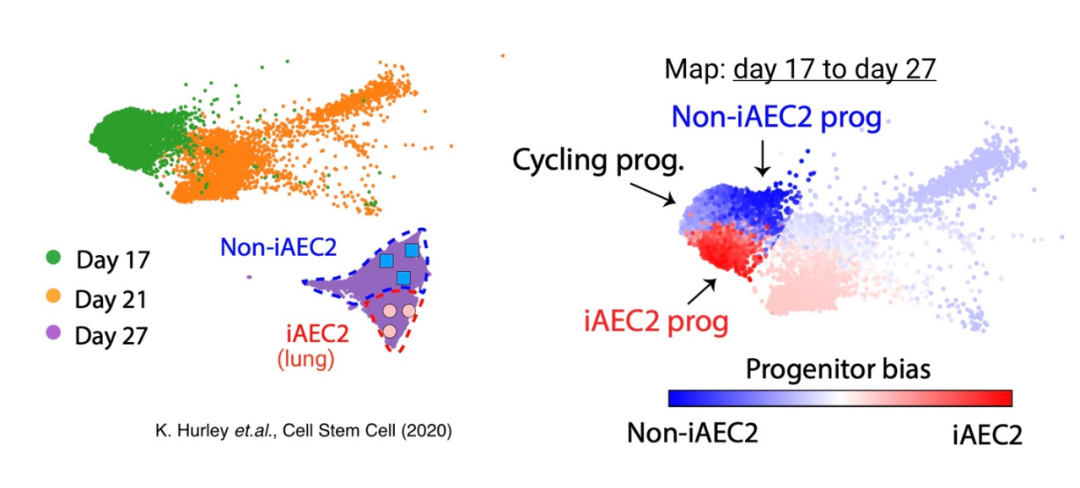
图6. CoSpar被应用于分析诱导分化体系,并揭示出第17天的细胞具有较强的分化偏好。
总结来说,谱系追踪技术具有很大的潜力去系统性地研究体内体外细胞分化的过程。然而它目前最大的瓶颈就是如何从相应的实验数据中挖掘有价值的信息。尤其是体内实验条件下,我们只能获得某一个时间节点的克隆谱系信息。如何从静态的观察获得动态的信息是一个巨大的挑战。王寿文博士开发的CoSpar通过结合单细胞转录组数据和克隆谱系信息,可以很准确地预测细胞的命运选择。这为系统地研究不同体系里体内和体外的细胞分化清除了障碍。同时,CoSpar可以有效地应对很稀疏的数据,为许多简易的实验设计带来了数据分析的方便。展望未来,一方面可以结合CoSpar和谱系追踪,深入研究更加具体的生物问题;另一方面,谱系追踪数据的分析远非完美。奠定坚实的理论基础是未来的一个努力方向!
相关论文信息:
https://doi.org/10.1038/s41587-022-01209-1
https://blog.sciencenet.cn/blog-3423233-1327020.html
上一篇:《分子细胞》:钱旭/郭宏骞/赵晓智合作揭示新型PTEN C211琥珀酸化修饰在肿瘤发生发展中的关键作用
下一篇:《细胞—干细胞》:8C样细胞可在体外捕获人类合子基因组激活程序