博文
QIIME中jackknifed_beta_diversity.py程序的运行过程
|||
1. Beta Diversity (weighted_unifrac, unweighted_unifrac)
beta_diversity.py -i pick_otus/otu_table.biom -o jackknifed/unrarefied_bdiv -t pick_otus/rep_set.tre
从所有OTU table和tree中计算beta多样性距离矩阵
运行完在jackknifed/unrarefied_bdiv/文件夹下生成两个文件:unweighted_unifrac_otu_table.txt和weighted_unifrac_otu_table.txt。即用加权unifrac和非加权unifrac计算出距离距阵
2. Rarefaction
multiple_rarefactions_even_depth.py -i pick_otus/otu_table.biom -d 5000 -o jackknifed//rarefaction/
建立稀释的OTU table,这一步与上一步无关。这里是在jackknifed/rarefaction/文件夹下生成10次jackknifed抽样
3. UPGMA on full distance matrix: weighted_unifrac
upgma_cluster.py -i jackknifed/unrarefied_bdiv/weighted_unifrac_otu_table.txt -o jackknifed/unrarefied_bdiv/otu_table_weighted_unifrac_upgma.tre
利用第1步中weighted_unifrac生成的beta多样性矩阵建立UPGMA树(在unrarefied_bdiv中生成了一个otu_table_weighted_unifrac_upgma.tre),可以理解为一个引导树。
4. Beta diversity on rarefied OTU tables (weighted_unifrac)
beta_diversity.py -i jackknifed//rarefaction/ -o jackknifed//weighted_unifrac//rare_dm/ -m weighted_unifrac -t pick_otus/rep_set.tre
利用第2步中10次抽样生成的biom文件,生成beta多样性距阵。在jackknifed/weighted_unifrac/rare_dm/文件夹下生成了10个矩阵
5. UPGMA on rarefied distance matrix (weighted_unifrac)
upgma_cluster.py -i jackknifed//weighted_unifrac//rare_dm/ -o jackknifed//weighted_unifrac//rare_upgma/
再利用上一步中的10个矩阵生成了10棵树文件。在jackknifed/weighted_unifrac/rare_upgma/下生成了10个树文件
6. consensus on rarefied distance matrices (weighted_unifrac)
consensus_tree.py -i jackknifed//weighted_unifrac//rare_upgma/ -o jackknifed//weighted_unifrac//rare_upgma_consensus.tre
再利用上一步中的10棵树生成了1棵一致树
7. Tree compare (weighted_unifrac)
tree_compare.py -s jackknifed//weighted_unifrac//rare_upgma/ -m jackknifed//weighted_unifrac//rare_upgma_consensus.tre -o jackknifed//weighted_unifrac//upgma_cmp/
生成了upgma_cmp文件夹下的文件,包括两个树文件和一个jackknife支持率的文件,其中这两个树文件是一致的,只是master_tree.tre文件的每个内节点保证有唯一的编号(来自官网上的解释)。
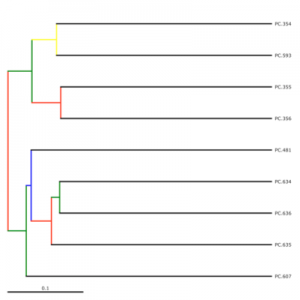
[注意] 如果要做UPGMA树图,使用的tre文件就即这个master_tree.tre文件。这在官网也有指出,另外QIIME还提供了一个make_bootstrapped_tree.py程序使用这个master_tree.tre文件以及支持率文件来绘制bootstrapped tree。
如下图:
8. Principal coordinates (weighted_unifrac)
principal_coordinates.py -i jackknifed//weighted_unifrac//rare_dm/ -o jackknifed//weighted_unifrac//pcoa/
利用第4步生成的10个距离矩阵文件生成了对应的10个PCoA分析数据,保存在jackknifed//weighted_unifrac//pcoa/文件夹下
9. emperor plots (weighted_unifrac)
make_emperor.py -i jackknifed//weighted_unifrac//pcoa/ -o jackknifed//weighted_unifrac//emperor_pcoa_plots/ -m mapfile.txt
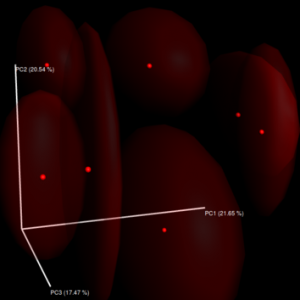
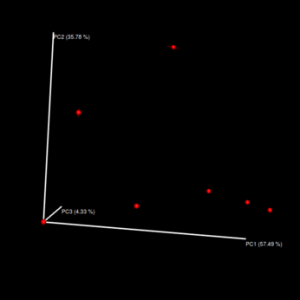
这一步生成了jackknifed//weighted_unifrac//emperor_pcoa_plots/文件夹下的PCoA 3D网页文件,里边的内容可以调整。和beta_diversity_through_plots.py程序生成的PCoA 3D图最大的差别是点周围有一片区域,这片区域在-e设置越大小模糊区域越小
例如:分别用3种不同的e值(100,1000,5000)来跑,7个样品,OTU数平均为13500。需要注意的是因为jackknifed是基于抽样的方法,所以同一条代码每次运行的结果可能都不一样。
e为100的如下:
e为1000的如下:
e为5000的如下:
===========================我是一条华丽的分隔线,请勿注释掉===============================
[注] 以下unweighted_unifrac方法分析得到的结果和上面weighted_unifrac一样,具体解析不再赘述
10. UPGMA on full distance matrix: unweighted_unifrac command
upgma_cluster.py -i jackknifed/unrarefied_bdiv/unweighted_unifrac_otu_table.txt -o jackknifed/unrarefied_bdiv/otu_table_unweighted_unifrac_upgma.tre
11. Beta diversity on rarefied OTU tables (unweighted_unifrac) command
beta_diversity.py -i jackknifed//rarefaction/ -o jackknifed//unweighted_unifrac//rare_dm/ -m unweighted_unifrac -t pick_otus/rep_set.tre
12. UPGMA on rarefied distance matrix (unweighted_unifrac) command
upgma_cluster.py -i jackknifed//unweighted_unifrac//rare_dm/ -o jackknifed//unweighted_unifrac//rare_upgma/
13. consensus on rarefied distance matrices (unweighted_unifrac) command
consensus_tree.py -i jackknifed//unweighted_unifrac//rare_upgma/ -o jackknifed//unweighted_unifrac//rare_upgma_consensus.tre
14. Tree compare (unweighted_unifrac) command
tree_compare.py -s jackknifed//unweighted_unifrac//rare_upgma/ -m jackknifed//unweighted_unifrac//rare_upgma_consensus.tre -o jackknifed//unweighted_unifrac//upgma_cmp/
15. Principal coordinates (unweighted_unifrac) command
principal_coordinates.py -i jackknifed//unweighted_unifrac//rare_dm/ -o jackknifed//unweighted_unifrac//pcoa/
16. emperor plots (unweighted_unifrac) command
make_emperor.py -i jackknifed//unweighted_unifrac//pcoa/ -o jackknifed//unweighted_unifrac//emperor_pcoa_plots/ -m mapfile.txt
https://blog.sciencenet.cn/blog-2970729-1069064.html
上一篇:rank abundance 解析
下一篇:alpha rarefaction curve 解析