博文
《细胞》:Danesh Moazed/李晴合作揭示真核DNA复制体介导的亲本组蛋白遗传与表观遗传新机制
|
北京时间2024年8月1日晚,美国哈佛医学院、霍华德休斯医学研究院(HHMI)Danesh Moazed研究组和北京大学李晴研究组合作,在Cell杂志发表了题为“A replisome-associated histone H3-H4 tetramer chaperone required for epigenetic inheritance”的研究论文。

细胞是如何实现在分裂后可以记住自己的“身份”的是一个重要的生物学问题,理解这一问题对我们认识正常发育、癌变或者衰老至关重要。由异染色质(heterochromatin)包裹的基因沉默(gene silencing)区域可以通过染色质介导的传承维持状态,这是细胞能维持一部分记忆的机制。近期研究发现,异染色质相关的亲本组蛋白修饰(H3K9me3)在裂殖酵母(Schizosaccharomyces pombe)可以通过DNA复制实现遗传且该遗传不依赖于诸如沉默子(silencer)或者RNA沉默(RNAi)等的核酸机制【1,2】。沉默相关的组蛋白修饰再通过组蛋白甲基化转移酶(Clr4/Suv39h)的“读写”(read-write)功能将其拷贝到新生成的核小体组蛋白上,以实现异染色质的表观遗传(epigenetic inheritance)。在这一科学模型中,DNA复制偶联的亲本组蛋白遗传就对异染色质的表观遗传有着重要的作用。
在本研究中,作者结合AlphaFold-Multimer结构预测、遗传、生化、生物物理和测序等手段发现DNA复制体(replisome)中介导DNA复制压力信号通路(replication checkpoint pathway)的组分Mrc1/CLASPIN存在一段和介导压力信号无关的且在真核生物中保守的组蛋白H3-H4四聚体结合区域(图一)。该区域在结构预测和物理性质上类似于核小体DNA、H2B与H2A结合H3-H4四聚体时的复合体,暗示Mrc1/CLASPIN演化出类似于核小体的性质在DNA复制期间对H3-H4四聚体进行保护以进行传承。作者通过AlphaFold-Multimer筛选(screen)发现在所有DNA复制体组分蛋白中,Mrc1/CLASPIN对H3-H4四聚体的结合获得了最高的预测分数(ipTM)。由于Mrc1/CLASPIN全长超过1000个氨基酸且基本无序(unstructured),遗传和生化手段难以研究其功能与机制,AlphaFold-Multimer作为了一种全新的手段能够帮助研究人员快速发现潜在生物大分子之间的相互作用以推进实验验证来发现新的生物学机制。
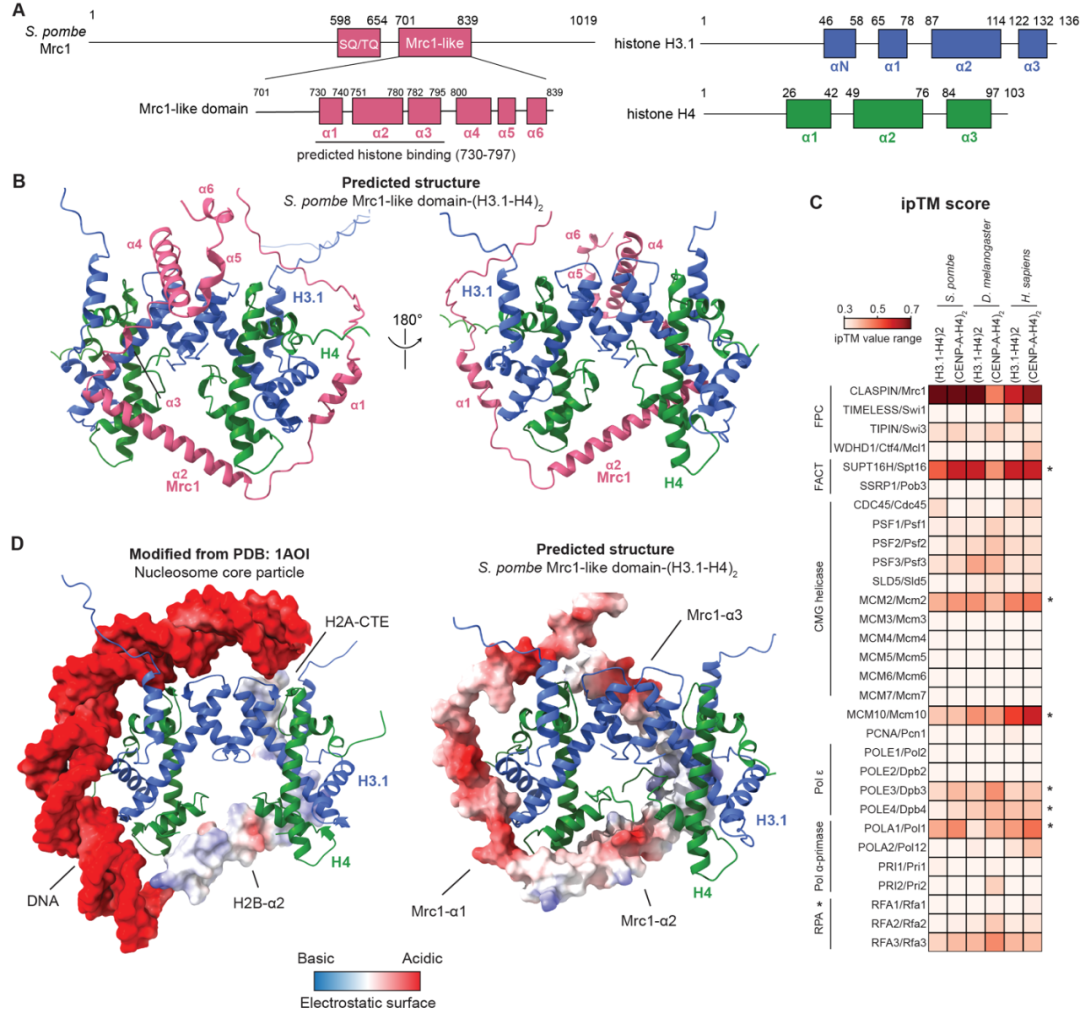
图一:AlphaFold-Multimer预测一段演化保守的Mrc1/CLASPIN区域结合组蛋白H3-H4四聚体
为验证AlphaFold的结构预测,作者首先通过生物化学手段确认了AlphaFold-Multimer预测的H3-H4四聚体结合区域的特异性,并通过生物物理手段确认在体外体系中,单个Mrc1的组蛋白结合区域结合一个H3-H4四聚体。之后作者基于AlphaFold-Multimer能量最小化的结构预测,设计了一系列在Mrc1组蛋白结合区域的突变,并确认在体外体系中一组疏水区域和一对酸性氨基酸对Mrc1结合组蛋白是必要的。作者之后又在裂殖酵母(S. pombe)与芽殖酵母(Saccharomyces cerevisiae)中分别确认了Mrc1的组蛋白结合区域的突变会导致异染色质沉默丢失。这两种酵母在4.2-3.3亿年前的演化期间已经发生分化,裂殖酵母中的异染色质是基于H3K9me3建立的,而芽殖酵母中的异染色质是由Sir2/Sir3/Sir4介导的去乙酰化H4K16染色质。Mrc1介导的亲本组蛋白传承对两种酵母截然不同的异染色质体系都是必要的,体现出DNA复制体介导的亲本组蛋白遗传并非H3K9me3特异性的遗传,而是在全基因组层面的遗传。
作者之后通过eSPAN测序手段鉴定Mrc1是主要介导亲本组蛋白遗传到先导链(leading strand)还是滞后链(lagging strand)。前期研究发现,Mcm2与Pol1的组蛋白结合区域倾向于将亲本组蛋白遗传到滞后链【3-5】,而Dpb3-Dpb4倾向于将组蛋白遗传到先导链【6】。作者首先在芽殖酵母中发现,Mrc1全敲除或大片段敲除会导致亲本组蛋白更偏向于传递到滞后链。为了仔细研究Mrc1的组蛋白结合区域是否存在内在的亲本组蛋白传承偏好,作者在芽殖酵母和裂殖酵母中分别使用小片段敲除和点突变发现Mrc1组蛋白结合区域和之前发现的组蛋白结合组分相比没有明显的先导链-滞后链偏好。作者进一步确认了Mrc1的组蛋白结合突变体使得先导链和滞后链上的亲本组蛋白密度都有显著下降。据此,作者得出结论:Mrc1是一个在DNA复制体上的组蛋白分选组分(图二)。
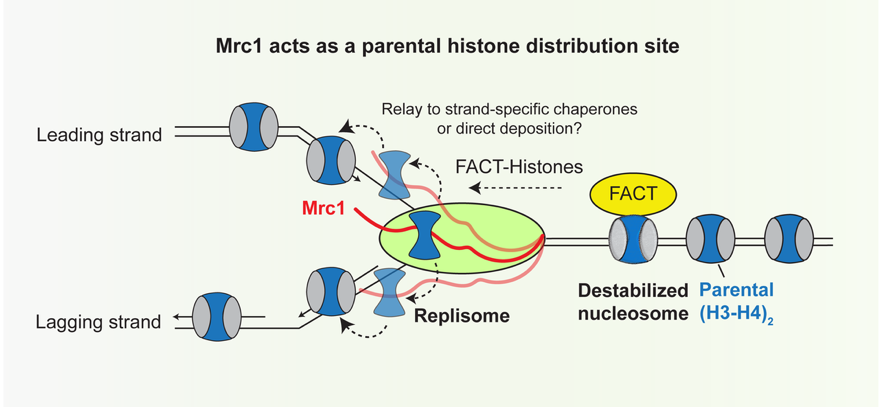
图二:Mrc1是一个在DNA复制体上的亲本组蛋白分选组分(概念图)
早期研究发现组蛋白分子伴侣蛋白(histone chaperone)FACT复合物(Spt16, Pob3/SSRP1)在染色质复制中具有重要的功能【7-10】。FACT的组分之一Spt16可以结合H3-H4四聚体【11】,暗示着Mrc1和FACT能在DNA复制期间共同或分别维持H3-H4四聚体的稳定。AlphaFold-Multimer筛选发现在DNA复制体上有多个FACT结合位点。通过遗传和生化手段,作者确认FACT与DNA复制体组分Tof1/Swi1/TIMELESS以及Pol1的结合对异染色质的表观遗传是必要的。这些研究暗示着FACT作为一个活动的组蛋白分子伴侣蛋白参与运输亲本组蛋白。
由于DNA复制体上存在多个可以结合组蛋白的组分,这些组分在复制体中占据不同区域,AlphaFold结构预测和冷冻电镜结构还发现这些组蛋白结合组分可以和FACT一起结合组蛋白。因此,作者提出DNA复制体上存在多个亲本组蛋白传承中间位点(Parental histone inheritance intermediate sites)(图三):
-P位点(P site):DNA复制体会介导核小体DNA脱离从而招募FACT复合体,此时的亲本核小体处于中间态【12】。
-D位点(Distribution site, D site):Tof1/Swi1/TIMELESS招募FACT-组蛋白至DNA复制体,Mrc1组蛋白结合区域安置于Cdc45/Mcm2并用于分配组蛋白至先导链和滞后链。
-LG1位点(Lagging strand site 1, LG1 site):Mcm2组蛋白结合区域可以和FACT安置于Tof1/Swi1/TIMELESS【13】。
-LG2位点(Lagging strand site 2, LG2 site):Pol1组蛋白结构结合区域与FACT安置于Ctf4/Mcl1/AND-1。
-LD1位点(Leading strand site 1, LD1 site):Dpb3-Dpb4安置于DNA复制酶 ε。
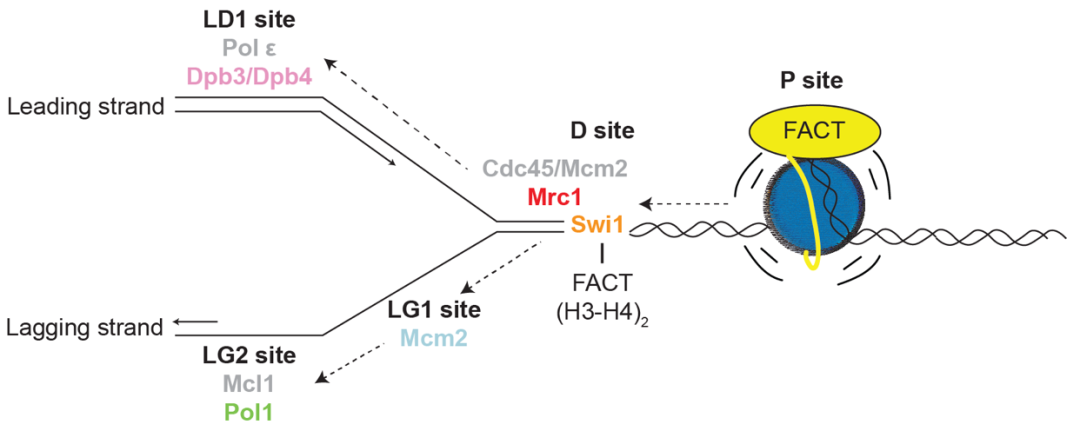
图三:DNA复制体上的多个亲本组蛋白传承中间位点模型图
综上所述,该研究为DNA复制体介导的亲本组蛋白遗传通路建立了一个研究框架。未来仍需要通过实验结构生物学手段验证所有位点的存在,并更进一步研究不同位点之间的关系以理解亲本组蛋白如何在不同位点实现动态传承。
哈佛大学博士生俞钧陶、北京大学博士生张煜婕为该论文并列第一作者,哈佛医学院、霍华德休斯医学研究院(HHMI)教授Danesh Moazed和北京大学教授李晴为共同通讯作者,美国哥伦比亚大学贾松涛(Songtao Jia)研究组,哥伦比亚大学医学院张志国(Zhiguo Zhang)研究组以及哈佛医学院Steven Gygi研究组也参与了本研究。Danesh Moazed研究组感谢美国国立卫生研究院(NIH)和霍华德休斯医学研究院(HHMI)的资助,李晴研究组感谢国家自然科学基金委员会、北京市教育委员会、北京大学-清华大学生命科学联合中心和蛋白质与植物基因研究国家重点实验室的资助。
同期,丹麦哥本哈根大学Genevieve Thon研究组与Anja Groth研究组在Cell也发表了关于Mrc1/CLASPIN的工作,哥伦比亚大学贾松涛研究组与哥伦比亚大学医学院张志国研究组在Molecular Cell杂志发表了关于Mrc1与Mcm2/Cdc45结合部分对亲本组蛋白传承与表观遗传的重要功能的研究。
相关论文信息:
https://doi.org/10.1016/j.cell.2024.07.006
参考文献
编辑 |余 荷
欲知更多内容,敬请围观小柯机器人频道:

https://blog.sciencenet.cn/blog-3423233-1444886.html
上一篇:《自然》:赵岩团队与合作者揭示去甲肾上腺素转运体转运机制及小分子和多肽类药物的结合模式
下一篇:1天2篇《细胞》,西湖大学闫浈团队揭秘叶绿体门控机制