博文
mLife | 细菌泛基因组中必需与非必需基因的朦胧边界
||

中国农业大学田长富教授和张子丁教授团队合作的文章“Cofitness network connectivity determines a fuzzy essential zone in open bacterial pangenome” 已在mLife网站正式上线。该文章主要研究了理想条件下细菌泛基因组各基因对适合度的贡献。通过对富营养条件下中华根瘤菌属菌株的泛基因组Tn-seq数据进行网络分析,发现:基因必需性类别与基因保守水平和网络连接度之间正相关,核心基因组中菌株间共有和特异的必需基因在翻译和细胞包膜生物合成等基本功能中富集,揭示了泛基因组必需与非必需基因的朦胧边界。

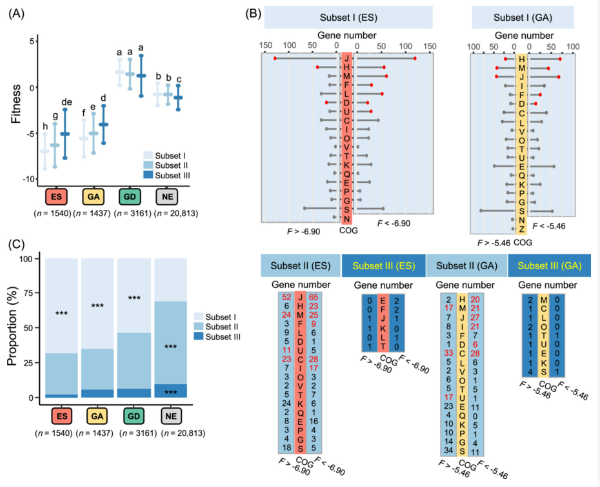
图 1 基因必需性等级与中华根瘤菌属泛基因组中的保守水平相关。(A) 不同保守水平基因之间适合度值的多重比较分析。误差线代表标准差;适合度值通过蒙特卡洛方法计算;ES (必需)、GA (生长有利)、NE (非必需) 和GD (生长不利) 由HMM方法定义;在ES和GA类别中,适合度值依次从保守程度递减的子集I、II和III逐渐增加。(B) 适合度值F高于或低于子集I平均适合度值的ES和GA基因的COG富集分析 (ES为-6.90,GA为-5.46)。图中显示了属于特定COG类别的基因数量,与五个测试菌株中具有COG注释的总基因数量相比,显著富集用红点或红色数字表示 (Fisher精确检验,p < 0.05)。(C) 子集I-III中基因比例的Fisher精确检验 (***p < 0.001)。ES、GA和GD类别富集了子集I基因。
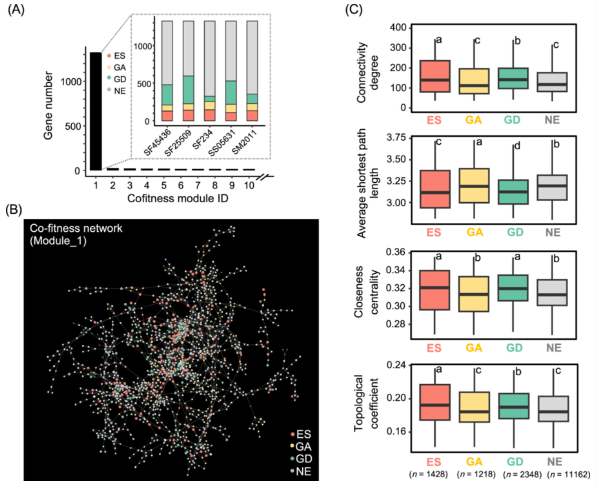
图 2 基因必需性类别与中华根瘤菌属泛基因组成员的共适合度网络连接度相关。(A)基于蒙特卡罗适合度值的共适合度网络分析 (只包括相关度最高的前1%的边)。(B) Module_1的拟合网络。圆点的大小与共适合度网络的连接度成正比。不同颜色表示 (A) 中SF45436的适合度类别。(C) 连接度、平均最短路径长度、接近中心性和拓扑系数的共适合度网络分析。图中不同小写字母表示平均值之间的显著差异 (Tukey HSD检验,校正后的p < 0.05)。
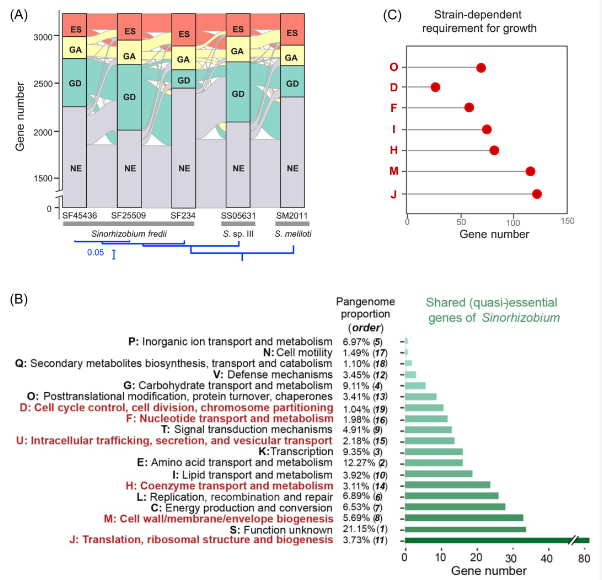
图3 中华根瘤菌属核心基因组中必需与非必需基因间的朦胧边界。(A) 核心基因的必需性类别呈现菌株依赖性。最大似然系统发育树基于五个测试菌株的3284个核心基因,所有分支具有100%的可信度检验支持。(B-C) 共有的 (B) 和菌株特异的 (C) (准)必需核心基因的COG富集分析。(B) 和 (C) 的红色表示Fisher精确检验中p < 0.05。(B) 显示了泛基因组27,724个具有COG注释的基因中每个COG类别的比例及其丰度顺序。
引用本论文:Zhang P, Zhang B, Ji Y, Jiao J, Zhang Z, Tian C‐F. Cofitness network connectivity determinesa fuzzy essential zone in open bacterial pangenome. mLife. 2024;3:277–290.
原文链接:
https://doi.org/10.1002/mlf2.12132




mLife
期刊简介
mLife是由中国科学院主管、中国科学院微生物研究所主办(中国微生物学会为合作单位)的我国微生物学领域第一本综合性高起点英文期刊。mLife瞄准全球微生物学领域高水平科研成果和前沿进展,报道内容覆盖微生物学各个学科。mLife的办刊目标是打造微生物学领域综合性国际旗舰期刊。目前,mLife已被国内外重要数据库ESCI、PubMed Central、Scopus、CSCD、DOAJ、CAS等收录。2024年6月20日,mLife首获影响因子4.5,位于Q1区。
期刊网站:
https://wileyonlinelibrary.com/journal/mLife
https://www.sciopen.com/journal/2097-1699
投稿网站:https://mc.manuscriptcentral.com/mlife
扫码关注mLife

mLife@im.ac.cn
010 - 64807055
https://blog.sciencenet.cn/blog-3563286-1445336.html
上一篇:FMH | 河南大学李昌勤教授团队:迷迭香中二萜类化合物的生物学效应及作用机制综述
下一篇:FMH | 期刊联合主编孙东晓院士:从传统药食同源到现代食药同源的转变