博文
《细胞》:张锋团队揭示真核基因编辑器Fanzor的结构多样性和DNA切割机制
|
北京时间2024年8月28日,美国Broad研究所/麻省理工学院张锋团队在Cell杂志上发表了题为“Structural Insights into the Diversity and DNA Cleavage Mechanism of Fanzor”的研究论文。
该研究展示了Fanzor蛋白在不同生物中所表现出的分子多样性,并深入解析了其通过RNA引导的DNA切割机制。

CRISPR-Cas系统凭借其RNA引导的核酸内切酶活性,已经在基因编辑领域得到了广泛应用。2023年12月,美国食品和药物管理局(FDA)批准了首个基于CRISPR技术的基因编辑疗法,这一里程碑式的进展标志着科学研究的突破已经成功转化为实际治疗,为数以万计的患者带来了新的希望。与此同时,CRISPR起源及其同源系统的研究也取得了重大突破。2023年6月,张锋团队在Nature杂志上首次报道了一种在真核生物中发现的RNA引导核酸内切酶——Fanzor (Fz) (ref.1)。Fanzor的发现源自对OMEGA系统的深入研究,这一系统由广泛存在于细菌和古细菌中的IS200/IS605和IS607转座子编码 (ref.2)。OMEGA系统包含了Cas9的祖先IsrB和IscB,以及Cas12的祖先TnpB。而Fanzor则是TnpB在真核生物中的同源物,具备可编程的RNA引导核酸内切酶活性,展示出显著的基因编辑潜力。Fanzor蛋白家族在物种分布和分子结构上展现出极大的多样性,暗示了其在生物学功能上的广泛可能性。
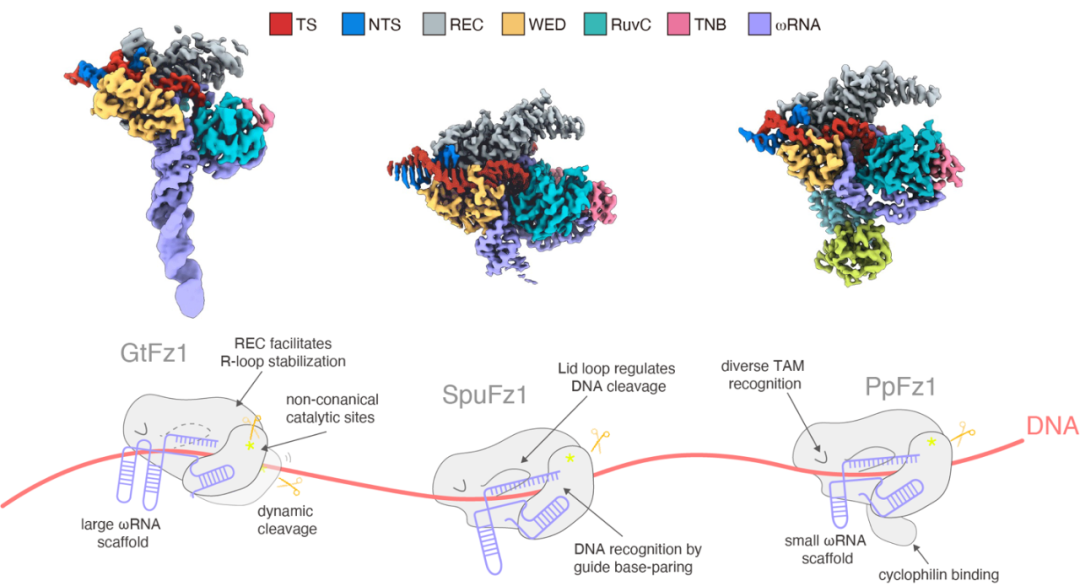
作者通过冷冻电镜解析了来自不同物种Fanzor蛋白,以及不同构象下的13个结构,包括来自藻类Guillardia theta的GtFz1、真菌Spizellomyces punctatus的SpuFz1和寄生真菌Parasitella parasitica的PpFz1。这些结构揭示了Fanzor蛋白在RNA结合、DNA识别和切割中的独特性。例如,研究发现,尽管这些蛋白在RNA结合界面上表现出保守性,但在DNA靶向临近基序(Target-adjacent Motif, TAM)的识别和负责切割的催化位点上存在差异,尤其是GtFz1的催化三联体中出现了非典型的N残基,这一变化增强了其体外DNA切割活性。此外,PpFz1中的一个独特结构域被发现能够与一种称为亲环蛋白(Cyclophilin)的蛋白质结合,提示其可能在寄主生物的生理功能中扮演重要角色。这一发现揭示了Fanzor蛋白在基因转移和寄生关系中的潜在功能。
此外,该研究通过解析不同DNA结合构象的结构,揭示了Fanzor如何调控自身以激活DNA切割活性。在GtFz1中,未结合DNA的状态以及两个结合了不同形式DNA的结构展示了REC结构域在R-loop形成和稳定中的关键构象变化。而在SpuFz1和PpFz1中,研究解析了具有不同数量Guide/DNA碱基配对的结构,揭示了RuvC结构域中的一个环状结构(通常被称为“Lid”)对蛋白激活的关键调控作用。研究发现,这个Lid能够随着Guide/DNA配对碱基的数量发生构象变化,从而调控蛋白的激活。通过对同源蛋白家族的模型分析,研究团队还发现这个Lid区域在所有Cas12、TnpB和Fanzor家族蛋白中都可能存在构象适应性,暗示了一种广泛存在的RNA引导核酸内切酶活性的激活机制。
总体而言,作者通过来自不同物种以及不同DNA结合构象的Fanzor蛋白结构分析,揭示了该家族蛋白的分子多样性,并揭示了Fanzor蛋白的激活机制。该研究不仅深化了领域对Fanzor家族分子机制的理解,也为未来基因编辑工具的设计提供了新的思路。
Broad研究所/麻省理工学院张锋课题组博后徐沛雨和Makoto Saito为该论文的共同第一作者,张锋教授为通讯作者。
相关论文信息:
https://doi.org/10.1016/j.cell.2024.07.050
参考文献
1.Makoto Saito, Peiyu Xu, Guilhem Faure, Samantha Maguire, Soumya Kannan, Han Altae-Tran, Sam Vo, AnAn Desimone, Rhiannon K. Macrae & Feng Zhang. Fanzor is a eukaryotic programmable RNA-guided endonuclease. Nature620, 660–668 (2023)
2.Han Altae-Tran, Soumya Kannan, F. Esra Demircioglu, Rachel Oshiro, Suchita P. Nety, Luke J. McKay, Mensur Dlakic, William P. Inskeep, Kira S. Makarova, Rhiannon K. Macrae, Eugene V. Koonin & Feng Zhang. Science 374,6563 (2021)
编辑 |余 荷
欲知更多内容,敬请围观小柯机器人频道:

https://blog.sciencenet.cn/blog-3423233-1448772.html
上一篇:1天2篇《细胞》,西湖大学闫浈团队揭秘叶绿体门控机制
下一篇:《自然—遗传学》:赵方庆团队开发高通量三维空间转录组新技术