博文
多肽和人工智能揭示药物设计的新途径  精选
精选
||
多肽和人工智能揭示药物设计的新途径
诸平
据《科技日报》(SciTechDaily)网站2024年9月2日刊发来自西交-利物浦大学(Xi'an Jiaotong-Liverpool University简称XJTLU)提供的消息,多肽和人工智能揭示药物设计的新途径(Peptides and AI Unveil New Pathways in Drug Design)。
中国科学家探索了多肽的聚集方式,揭示了氨基酸组成对其结构和行为的显著影响。该研究将分子模拟与人工智能相结合,预测肽的相互作用,为改进药物配方和材料,以及了解阿尔茨海默氏症等肽相关疾病提供必要的数据。
理解肽聚集:医药和材料进步的关键(Understanding Peptide Aggregation: Key to Advances in Medicine and Materials)
来自中国的研究人员研究了短肽链如何聚集在一起,以加深我们对这一过程的理解,这对药物稳定性和材料开发至关重要。相关研究结果于2024年9月2日已经在《JACS Au》杂志网站在线发表——Jiaqi Wang, Liu Zihan, Shuang Zhao, Yu Zhang, Tengyan Xu, Stan Z. Li, Wenbin Li. Aggregation Rules of Short Peptides. JACS Au, 2024. DOI: 10.1021/jacsau.4c00501. Published September 2, 2024. https://doi.org/10.1021/jacsau.4c00501
参与此项研究的有来自中国浙江温州医科大学第一附属医院(First Affiliated Hospital, Wenzhou Medical University, Wenzhou, China)、中国浙江杭州西湖大学(Westlake University, Hangzhou, Zhejiang, China)、中国江苏苏州西交-利物浦大学(Xi’an Jiaotong-Liverpool University, Suzhou, Jiangsu, China)以及中国北京清华大学(Tsinghua University, Beijing, China)的研究人员。
上述研究论文为研究被称为肽的短蛋白如何相互作用、折叠和功能提供了有价值的见解。这些发现对医学、材料科学和生物技术具有重要意义。
肽类物质是氨基酸的短链,在人体中通过构建结构、加速化学反应和支持我们的免疫系统而起着至关重要的作用。蛋白质的特定功能是由其氨基酸如何相互作用并聚集成三维结构决定的。
人工智能和分子动力学:预测肽的行为(AI and Molecular Dynamics: Predicting Peptide Behavior)
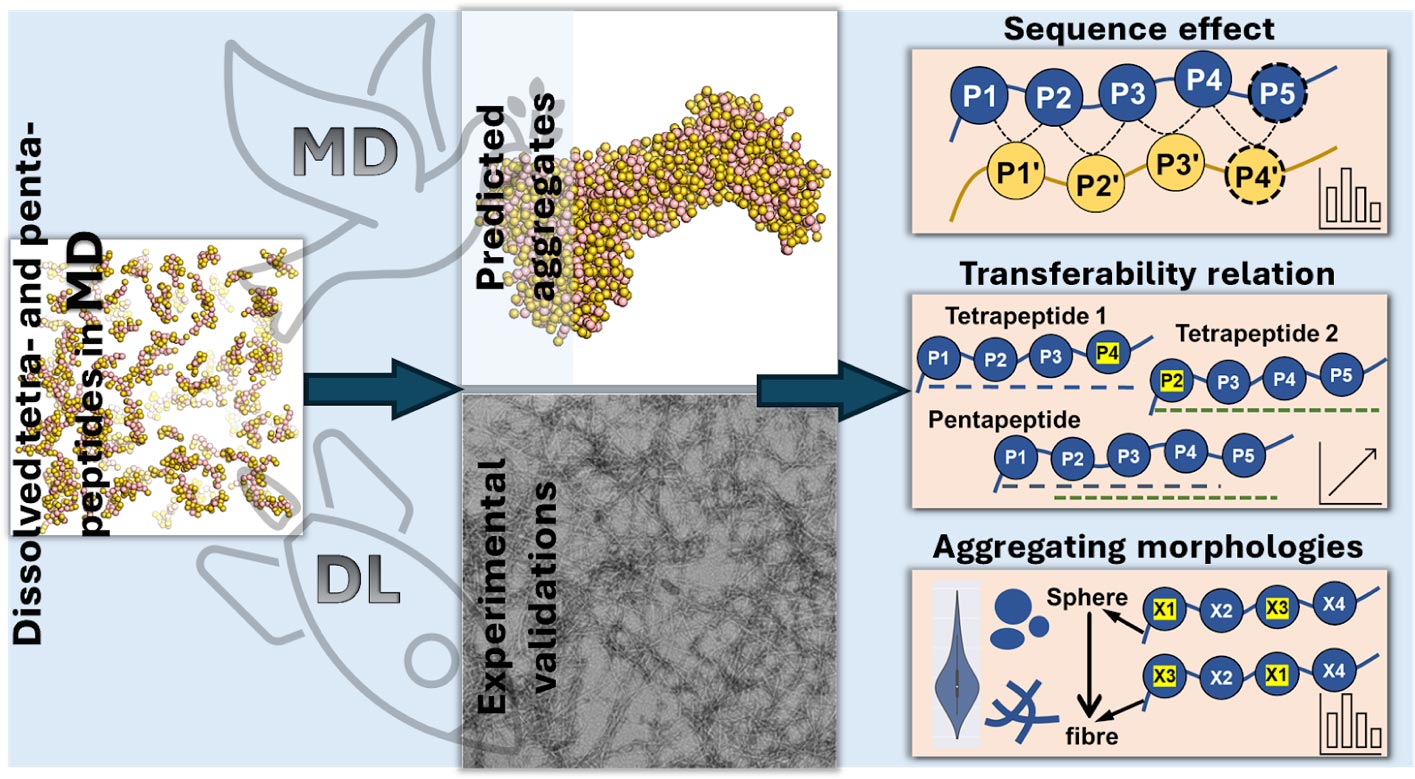
研究小组利用分子动力学模拟和变压器回归网络(Transformer Regression Networks)等包括深度学习模型的先进的人工智能技术,预测了四、五种氨基酸(分别为四肽和五肽)的不同肽如何根据氨基酸序列聚集。
通过分析16万种四肽和320万种五肽,他们发现某些氨基酸,尤其是芳香氨基酸,如色氨酸(tryptophan)、苯丙氨酸(phenylalanine)和酪氨酸(tyrosine),显著增强了聚集性,尤其是当它们位于肽链的一端——C端(C-terminus)时。这可能是因为芳香氨基酸具有环状结构,通过它们的电子云相互吸引,通常被称为π-π相互作用,这有助于它们聚集在一起。相比之下,亲水性氨基酸,如天冬氨酸(aspartic acid)和谷氨酸(glutamic acid),由于与水分子的强烈相互作用,阻止肽粘在一起,从而抑制聚集。
肽的特性及其对聚集的影响(Peptide Characteristics and Their Impact on Aggregation)
研究还表明,改变氨基酸序列会影响聚集。例如,将芳香氨基酸添加到肽链的末端会增加聚集,而将带负电荷的氨基酸放在肽链的开头则会减少聚集。研究小组还发现,肽会根据氨基酸的类型和位置聚集成不同的形状。
杭州的西湖大学助理教授、该研究的共同通讯作者之一李文彬博士(Dr. Wenbin Li)解释说:“带电荷的氨基酸通常会使肽形成长长的线状结构,而那些疏水的氨基酸往往会形成圆形的球状簇。我们还发现,通过了解四肽是如何相互聚集在一起的,我们可以预测五肽的行为,从而更容易预测更长的肽会聚集在一起。”
对生物技术和疾病治疗的启示(Implications for Biotechnology and Disease Treatment)
这些发现为预测和管理肽如何聚集提供了重要的指导。西交-利物浦大学助理教授、该研究的第一作者汪嘉琪博士(Dr. Jiaqi Wang)说:“这些知识可以帮助创造新材料,设计更稳定的药物和药物输送系统,并了解与肽聚集有关的疾病,如阿尔茨海默病,在这种疾病中,淀粉样蛋白-β肽聚集在大脑中形成破坏性斑块。
它还可以改善生物技术,如半导体、生物传感器和诊断,确保这些工具准确和一致地工作。通过提供对肽聚集的新见解,这项研究将推动生物化学、材料科学和计算生物学的发展。它还展示了人工智能与科学发现的融合。这些进步可能会在医疗、环保产品和创新技术方面取得突破。”
本研究得到了西湖大学未来产业研究中心(Research Center for Industries of the Future at Westlake University under award no. WU2022C041)、中国国家自然科学基金{National Natural Science Foundation of China (no. 52101023, no. U21A20427)}以及中华人民共和国科学技术部{Ministry of Science and Technology of the People’s Republic of China (no. 2021YFA1301603)}的资助。
上述介绍,仅供参考。欲了解更多信息,敬请注意浏览原文或者相关报道。
The elucidation of aggregation rules for short peptides (e.g., tetrapeptides and pentapeptides) is crucial for the precise manipulation of aggregation. In this study, we derive comprehensive aggregation rules for tetrapeptides and pentapeptides across the entire sequence space based on the aggregation propensity values predicted by a transformer-based deep learning model. Our analysis focuses on three quantitative aspects. First, we investigate the type and positional effects of amino acids on aggregation, considering both the first- and second-order contributions. By identifying specific amino acids and amino acid pairs that promote or attenuate aggregation, we gain insights into the underlying aggregation mechanisms. Second, we explore the transferability of aggregation propensities between tetrapeptides and pentapeptides, aiming to explore the possibility of enhancing or mitigating aggregation by concatenating or removing specific amino acids at the termini. Finally, we evaluate the aggregation morphologies of over 20,000 tetrapeptides, regarding the morphology distribution and type and positional contributions of each amino acid. This work extends the existing aggregation rules from tripeptide sequences to millions of tetrapeptide and pentapeptide sequences, offering experimentalists an explicit roadmap for fine-tuning the aggregation behavior of short peptides for diverse applications, including hydrogels, emulsions, or pharmaceuticals.
https://blog.sciencenet.cn/blog-212210-1449602.html
上一篇:可降解聚合物涂层支架与传统支架相比没有长期优势
下一篇:量子光学的突破