博文
通过高分辨率结构成像对抗多重耐药细菌
||
通过高分辨率结构成像对抗多重耐药细菌
诸平
据德国汉堡大学(University of Hamburg/ Universität Hamburg)2023年9月29日提供的消息,由汉堡大学化学系领导的欧洲研究小组提出了一项研究,展示了17种不同抗生素核糖体化合物(distinct antibiotic ribosomal compounds)的高分辨率结构。这些知识可以为开发新的抗生素来对抗多重耐药细菌铺平道路。相关研究结果于2023年8月7日已经在《自然结构与分子生物学》(Nature Structural & Molecular Biology)杂志网站发表——Helge Paternoga, Caillan Crowe-McAuliffe, Lars V Bock, Timm O Koller, Martino Morici, Bertrand Beckert, Alexander G Myasnikov, Helmut Grubmüller, Jiří Nováček, Daniel N Wilson. Structural conservation of antibiotic interaction with ribosomes. Nature Structural & Molecular Biology, 2023 Sep; 30(9): 1380-1392. DOI: 10.1038/s41594-023-01047-y. Published: 07 August 2023. https://www.nature.com/articles/s41594-023-01047-y
参与此项研究的除了来自德国汉堡大学的研究人员之外,还有来自德国哥廷根的马克斯普朗克多学科科学研究所(Max Planck Institute for Multidisciplinary Sciences, Göttingen, Germany)、瑞士洛桑联邦理工学院-洛桑大学的杜博切特成像中心(Dubochet Center for Imaging at EPFL-UNIL, Batiment Cubotron, Lausanne, Switzerland)以及捷克马萨里克大学(Masaryk University, Brno, Czech Republic)的研究人员。
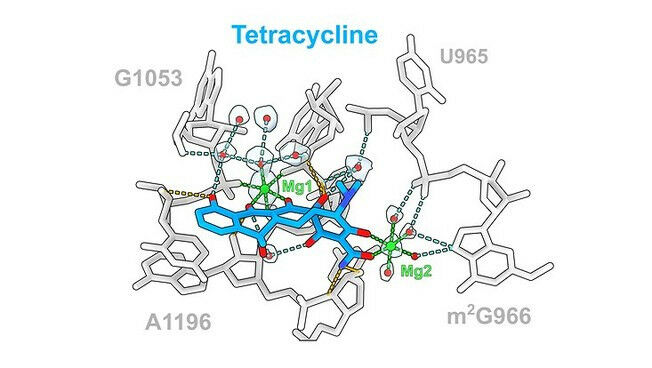
上述照片是由汉堡大学和帕特诺加(Paternoga)提供的抗生素四环素(antibiotic tetracycline,蓝色)及其与核糖体 rRNA(ribosomal rRNA,灰色)的相互作用,通过Mg1(绿色)或水分子(红色)直接(橙色)或间接(青色)相互作用介导。
核糖体(ribosome)是在植物、动物、人类和细菌细胞中发现的产生蛋白质的大分子复合物。它们是抗生素的重要靶标,在细菌中,抗生素与核糖体的亚基结合,导致读数错误甚至读数停止。这会导致形成有缺陷的蛋白质,从而失去其生物功能,然后细菌就会死亡。然而,越来越多的多重耐药细菌会阻止抗生素发挥作用。
详细了解抗生素,特别是抗生素核糖体结构,对于规避这种耐药性至关重要。在过去的20年中,已经发表了每一类主要的核糖体导向抗生素的抗生素-核糖体结构,提供了对其结合位点和作用机制的见解。
汉堡大学化学系丹尼尔·威尔逊(Daniel N. Wilson) 领导的团队使用冷冻电子显微镜以1.6 ~2.2 Å的分辨率对这些结构进行成像。“过去,抗生素核糖体结构主要以 2.5~3.5 Å的分辨率成像,分辨率的提高甚至使我们能够观察支持药物和核糖体成分之间相互作用的水分子。”丹尼尔·威尔逊说。研究人员发现,一般来说,有10~20个氢键与核糖体相互作用,并且水分子的相互作用水平不同类别的抗生素的情况各不相同。
此研究的抗生素包括6种临床相关抗生素类别,包括四环素类(呼吸道感染)、广谱氨基糖苷类(broad-spectrum aminoglycosides,结核病和脑膜炎)和截短侧耳素(pleuromutilins ,皮肤和软组织感染)。
在这项研究中,研究小组将这些抗生素应用于大肠杆菌(一种也存在于人类肠道中的大肠菌)的70S核糖体上。丹尼尔·威尔逊说:“我们期望这些信息将来可用于新型抗生素衍生物的基于结构的设计。通过识别可以改变的区域,可以选择性地置换结合水以与目标细菌相互作用。”
丹尼尔·威尔逊认为,高分辨率成像是向前迈出的一大步:“较低分辨率下的旧抗生素核糖体结构表明,每一类抗生素的结合位点是相似的;然而,在许多情况下,药物的确切位置以及药物结合位点存在深刻的差异。”丹尼尔·威尔逊指出,因此需要高分辨率的实验数据,来提供对相互作用的更准确的描述抗生素与核糖体的结合。
本研究得到了德国航空航天中心(Deutsche Zentrum für Luft-und Raumfahrt grant no. DLR01Kl1820)、德国研究基金会(Deutsche Forschungsgemeinschaft / the German Research Foundation grant no. WI3285/12-1)以及德国卓越战略(Germany’s Excellence Strategy grant no. EXC 2067/1-390729940)的资助。
上述介绍,仅供参考。欲了解更多信息,敬请注意浏览原文或者相关报道。
The ribosome is a major target for clinically used antibiotics, but multidrug resistant pathogenic bacteria are making our current arsenal of antimicrobials obsolete. Here we present cryo-electron-microscopy structures of 17 distinct compounds from six different antibiotic classes bound to the bacterial ribosome at resolutions ranging from 1.6 to 2.2 Å. The improved resolution enables a precise description of antibiotic-ribosome interactions, encompassing solvent networks that mediate multiple additional interactions between the drugs and their target. Our results reveal a high structural conservation in the binding mode between antibiotics with the same scaffold, including ordered water molecules. Water molecules are visualized within the antibiotic binding sites that are preordered, become ordered in the presence of the drug and that are physically displaced on drug binding. Insight into RNA-ligand interactions will facilitate development of new antimicrobial agents, as well as other RNA-targeting therapies.
https://blog.sciencenet.cn/blog-212210-1404474.html
上一篇:为体内细胞反应提供新的视角
下一篇:2023 年诺贝尔生理学或医学奖简介及其他