博文
QIIME2又双叒叕更新了
||
更新亮点都有啥呢?大概是几个命令和以前不一样了。以下是发布详细信息:
QIIME 2 2020.6 版本现已发布!感谢所有参与的辛勤工作!
提醒一下,我们计划发布的 QIIME 2 计划于 2020 年 8 月发布 (QIIME 2 2020.8),但请继续关注更新。
查看QIIME 2 2020.6 文档 [1]有关安装最新 QIIME 2 版本的详细信息,以及教程和其他资源。如果您遇到任何问题,请与QIIME 2论坛取得联系!
虚拟机镜像将在未来一周的某个时候更新!
重大更改
q2-diversity[2] core_metrics_phylogenetic: 参数已重命名,以适应目前正在使用的 unifrac 新的快速实现n_jobs或者n_jobs_or_threadscore_metrics&core_metrics_phylogenetic:该短语正在替换 ,因为observed features更好地描述了用户在 QIIME 2 中使用非分类功能的不同方式(OTU 是一个feature,但feature不一定是 OTU)observed_features和observed_otuscore_metrics&core_metrics_phylogenetic:不再接受零或负整数作为参数。用户应传递给这些参数以使用所有可用的物理内核n_jobs``n_jobs_or_threads``auto
发布亮点:
QIIME 2 框架
启用初始化数据的源跟踪 添加初始化数据收集的方法 进一步开发,这将允许插件开发人员展示他们的插件操作的使用实例,一个不可知的界面方式! 这包括:
Usage API改进了测试工具中的一些开发人员文档。
提高文件名的验证级别,以防止创建具有错误命名数据文件的项目。
文档
向我们展示了如何在Moving Pictures教程中的Artifact peek将一个部分添加到*"导入教程*"中,用于序列中具有条形码的multiplexed fastq文件。 阐明了 qiime tools view和 q2view 的行为为使用Windows的Linux 子系统添加了用户的本机安装说明 添加了一些粗糙的指南,用于选择最适合您的计算需求的安装方法 在 Atacama 教程中添加了子采样和样本筛选工具的示例 q2-demux更改了建议的措辞,将 vega 编辑器用于 q2 纵向变化图以反映当前实现 几个小错别字被修正 彻底改变了用户文档托管基础结构。 数据资源
添加了与 QIIME 2 一起使用的 SILVA 138 序列和分类文件格式。数据是使用 RESCRIPt 编制和格式化的(与RESCRIPt[3]共同开发)!有关这些文件如何生成以及重现此过程的流程的更多详细信息,请参阅本教程! 为 Greengenes 13_8 版本和 SILVA 138 版本添加了新的预训红尘的分类器(两者是最新版本),使用 scikit 学习 0.23.1 进行训练。这些分类器是使用 RESCRIPt 以新方式构建和测试的[4]! q2cli
修复了一条错误消息: 一个小拼写错误可能会导致令人惊讶的混乱! 合并在使用 API CLI 驱动程序的第一次迭代中。对于开发人员来说,这应该被视为"预发布",直到使用 API 和使用示例更加成熟。敬请期待在未来版本中在此领域执行更多操作! QIIME 2 工作室 2
修复了尝试导入数据时出现的错误! q2 功能分类器
说明了我们为什么我们没有blast分类器的多线程选项 在 extract-reads操作中添加了一个trim-right选项,允许在 3' 端修剪模拟读取。这对于 (例如,模拟来自 q2-dada2 的读取的修整选项)非常有用,以生成最佳修剪的参考序列以进行分类器训练,如此处所述[5],改进了 extract-reads的文档!使用 scikit -learn 0.23.1 训练新的功能分类器 Q2-phylogeny
添加了一种新方法, robinson-foulds用于比较两个进化树!q2-cutadapt[6]
添加了对混合方向的间向测序multiplex reads的支持 q2-composition [7]
修复了 ancom一个错误!可视化有 (意外) 试图除以零的倾向。删除了指向 ancom 绘图上的 vega 编辑器的链接, 等待 vega 5 的情节返工。 Q2-feature-table
向 merge方法添加了一些使用示例!添加了在表中重新命名要素或示例的新方法 修复了导致 summarize可视化效果有时显示扭曲的列宽度的错误删除了对汇总图上的 vega 编辑器的链接, 等待 vega 5 的情节返工 Q2-alignment
添加了一个新操作 , mafft-add允许将未比对的序列添加到现有对齐方式Q2-quality-filter
合并 q-score和q-score-joined到相同的方法 (q-score),并弃用q-score-joined。此方法将在 QIIME 2 的 2020 秋季版本中删除。Q2-quality-control
添加了一种新方法 filter_reads,该新方法支持通过SampleData[SequencesWithQuality]和SampleData[PairedEndSequencesWithQuality]比对到引用数据库来筛选和工件(也称为 FASTQ 序列数据)。你怎么能用这个?在去噪或重复之前筛选人类reads和其他非目标(或垃圾)序列。过渡并使用 alpha_phylogenetic和alpha_rarefaction,这是以前在alpha_phylogenetic_alt和alpha_phylogenetic_alt中提供的速度和内存优化实现。 将在下一个版本中删除。q2-diversity
添加了一个新的测试版多样性指标 jensenshannon!连接q2-diversity- lib 以执行 core-metrics和core-metrics-phylogenetic的 alpha 和 beta 多样性计算。有关相关API 更改,请参阅上面的"中断更改"。修复了允许 0 方差数值列筛选器 bioenv导致错误的错误。q2-diversity-lib
此插件计算 alpha 和 beta 多样性度量,酌情扩展可用语义类型的数量,并提供特定于度量的引文信息。 q2-types
添加了新的转换器,用于以有趣和有趣的方式处理每个样本序列 添加了更多比对序列转换器 q2-vsearch
修复了 cluster-features-closed-reference方法中的性能错误,导致某些用例具有潜在的显著加速。添加了一个很棒的新可视化工具 fastq_stats, 用于使用 vsearch 将单端或配对端序列蒸馏到 FASTQ 文件统计信息中!添加了所有 vsearch 方法接受短于 32nt 的序列的能力 设置 --fasta_width 0,在调用的 vsearch 命令上,以防止快速记录跨多行拆分q2-longitudinal[8]
从波动性图中删除了指向 vega 编辑器的链接 q2-demux
返工汇总可视化效果,以更好地通知用户缺少的reads q2-dada2
将伪池添加到所有 DADA2 工作流中,允许用户增加对样品中稀有变体(尤其是单体)的敏感性,只要在其他样本中独立观察这些变异。 更正了一个错误,其中仅包含一个功能的功能表被错误地诊断为空。 q2-sample-classifier[9]
给测试套件一些急需的 Tlc 与慷慨的重构。 q2-emperor[10]
更新到最新版本 1.0.1 1[11]
修复了双图箭头在可视化并行绘图时会错误地显示的问题。
修复了在分类数据中部分使用连续和发散颜色图的问题。
修复了具有排序数值和非数值的 Bug。
修复了轴设置无法从 Python API 正确加载的问题。
更新了各种依赖项。
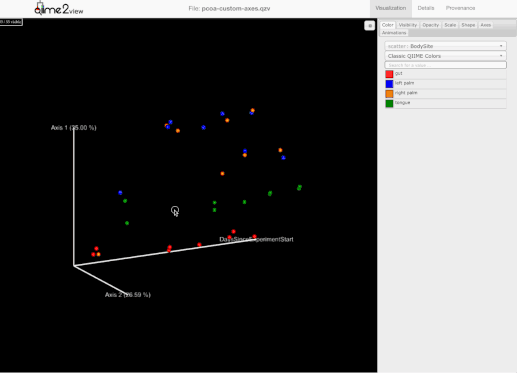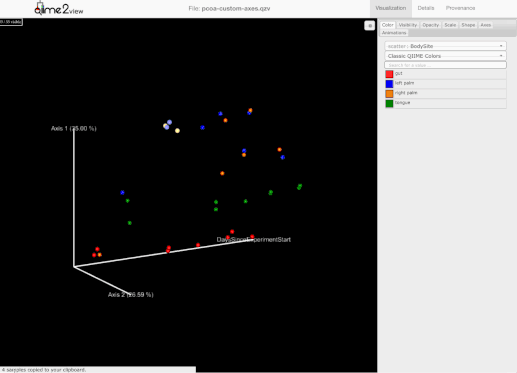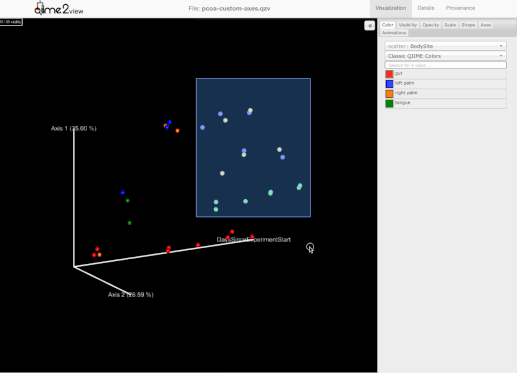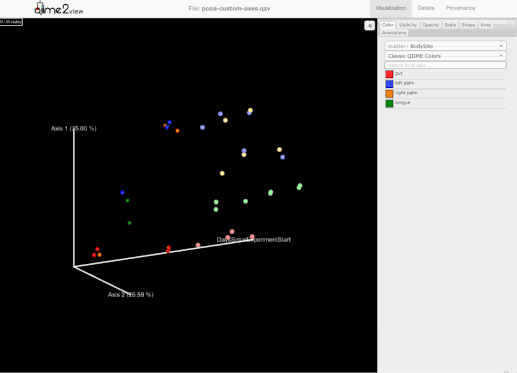
添加了选择一组示例并保存示例名称到用户剪贴板的能力。为此,用户需要按住Shift键并选择他们感兴趣的示例:
[
添加了对非混合混合方向单索引读取的支持。
快乐QIIMING!
QIIME 2 2020.6 文档 : https://docs.qiime2.org/2020.6/ q2-diversity: https://docs.qiime2.org/2020.6/plugins/available/diversity/ RESCRIPt: https://forum.qiime2.org/t/rescript-sequence-reference-database-management-tutorial/15494 构建和测试的: https://forum.qiime2.org/t/rescript-sequence-reference-database-management-tutorial/15494 此处所述: https://forum.qiime2.org/t/right-trim-option-in-extract-reads-for-paired-end-data/14191 q2-cutadapt: https://docs.qiime2.org/2020.6/plugins/available/cutadapt/ composition : https://docs.qiime2.org/2020.6/plugins/available/composition/ q2-longitudinal: https://docs.qiime2.org/2020.6/plugins/available/longitudinal q2-sample-classifier: https://github.com/qiime2/q2-sample-classifier q2-emperor: https://github.com/qiime2/q2-emperor 1.0.1 1: https://github.com/biocore/emperor
https://blog.sciencenet.cn/blog-623545-1240472.html
上一篇:[转载]GWAS和群体遗传学笔记
下一篇:宏转录组学习笔记(三)