博文
以史为鉴:新型冠状病毒2019-nCov感染可能的紧急预防与治疗策略
|
摘要
在目前新型冠状病毒传播途径不明晰的背景下,为控制病毒的传播和改善病人的预后状况,发展公共卫生和医药防治方法迫在眉睫。全基因组测序显示新型冠状病毒(2019-nCoV)与其近亲SARS冠状病毒(SARS-CoV)有非常强的序列相似性。2019-nCoV感染宿主靶细胞的spike蛋白相对于SARS-CoV显现一些关键的非同义突变,这可能导致已有针对SARS冠状病毒spike 蛋白为靶点的治疗方法和药物对2019-nCoV的有效性下降,但生物制剂和环状多肽类药物提供了潜在靶点。此外,RdRp蛋白和3CLpro蛋白在内的关键药物靶点与SARS-CoV共有极高的大于95%的序列相似度。因此,本文提出四种潜在药物(ACE2多肽,雷姆昔韦Remdesivir,3CLpro-1和一种新型乙烯基砜蛋白酶抑制剂)可能用于针对2019-nCoV感染的治疗。同时,本文也总结了前人对于这些靶点的药物研究工作,希望对今后相关的广谱抗新型冠状病毒药物的研究提供指导信息。
背景
2019新型冠状病毒(2019-nCoV)是一种源起于武汉华南海鲜市场的可感染人的冠状病毒,现已在中国广泛传播。1截至2020年1月26日,已经报道确诊病例超过2000例,死亡56例(新华社消息)。因目前对致病病原体知之甚少,供奋斗在疫情前线的医护人员和专家的治疗方案非常少。得益于中国研究人员极其迅速地完成了该病毒的分离和测序研究,可获知的该病毒全基因组序列(GenBank ID:MN908947.3)为治疗方法的研究奠定了基础。虽然研制针对新型冠状病毒特定靶点的疫苗,小分子药物和生物学疗法非常必要,但这些研究周期非常长,难以对目前奋战一线的医护人员和患者提供实际的帮助。新型冠状病毒与SARS冠状病毒(SARS-CoV, GenBank ID: NC_004718.3)在基因组上有82%同源性,几种关键酶更是有超过90%的相似性(图2-3, 5-6)。此前针对SARS冠状病毒和中东呼吸道感染综合征(MERS-CoV)的药物研究成果可能可直接应用到针对新型冠状病毒的治疗中。冠状病毒依赖其表面表达的spike蛋白实现对其宿主细胞表面受体的结合,进而进入受体细胞(图1)。2对于新型冠状病毒,有研究表明其细胞表面受体为血管紧缩素转化酶2(ACE2)。3在病毒进入受体细胞之后,病毒正义RNA直接与宿主的核糖体结合,翻译出两条大体量的共同终端的蛋白,经蛋白酶解过程后直接用于新病毒颗粒的组装。4两种参与蛋白酶解过程的关键蛋白酶分别为冠状病毒主蛋白酶(3CLpro)和木瓜蛋白酶样蛋白酶(PLpro)。5为复制其RNA基因组,冠状病毒编码一基于RNA的RNA聚合酶(RdRp)作为其复制酶,6以上四种蛋白对该病原体不可或缺。目前已有针对spike, RdRp, 3CLpro和 PLpro四种蛋白为靶点的治疗方法可能可应用于针对新型冠状病毒的治疗。本综述将具体分析上述四种蛋白在新型冠状病毒和SARS冠状病毒的相似性,并提出预防和治疗的策略。因目前对新型冠状病毒的毒性知之甚少, 本文还将讨论新型冠状病毒spike蛋白与ACE2受体的相互作用, 这可能挑战目前的观点,即2019-nCoV比SARS-CoV更不易归因于spike 与ACE2之间较弱的相互作用。
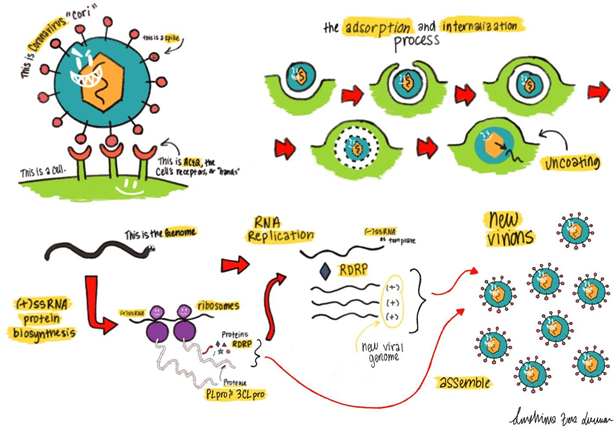
图 1.冠状病毒进入并在宿主细胞内复制的生命周期。冠状病毒依赖其表面表达的spike蛋白实现对其宿主细胞表面受体的结合,进而进入受体细胞。在病毒进入时RNA(+)链被释放,开始产生病毒外壳和复制RNA基因组。
Spike蛋白
新型冠状病毒与SARS冠状病毒均编码一大分子量spike蛋白(2019-nCoV: 1253 个氨基酸; SARS-CoV: 1273 个氨基酸),两种不同病毒来源的spike蛋白序列相似度为76%,但蛋白的N端存在大量变异(图2A)。Spike蛋白含有三个亚基,S1, S2和S3。SARS病毒的S1亚基包含一个与ACE2蛋白有着高亲和性的受体结合区域(RBD),负责识别细胞的受体。现有的研究表明新型冠状病毒亦含有该受体结合区域以用于与ACE2蛋白的结合及后续的细胞膜融合。我们对RBD受体结合区域进行序列比对,发现两种病毒有73.5%的序列相似性(图2A)。然而,新型冠状病毒与ACE2蛋白受体直接作用的区域(图2B: 1和2)内存在大量非保守性突变(图 2B)。2SARS冠状病毒spike蛋白和ACE2受体络合物的晶体和冷冻电镜结构(PDB entries: 2AJF and 6ACD)都表明只有区域1和区域2通过氢键相互作用和疏水相互作用与ACE2结合。因新型冠状病毒的这两个关键区域的某些氨基酸残基被替代,这可能导致氢键相互作用和疏水相互作用减弱。因此,目前人们预测新型冠状病毒的受体结合区域与ACE2受体的相互作用将弱于SARS冠状病毒。3然而这两个区域都是高度环状结构,这两个环状结构域内大量的变异会导致蛋白结构重排,这可能会引入新的甚至可能更强的与ACE2受体间的相互作用。这两种病毒在区域2几乎不存在序列上的相似性;但是得出此蛋白会以同样方式折叠与ACE2受体相互作用的结论为时尚早。假定新型冠状病毒的区域2以SARS冠状病毒冠状病毒中存在的方式折叠,F486将向右偏移,与ACE2受体的L79和M82两个残基产生强疏水相互作用。这些相互作用因在SARS RBD-ACE2络合物中相同位置显著更小的残基L472的存在而不会发生。另外,冠状病毒spike蛋白残基Q474也可能与ACE2受体中的Q24残基在空间位置重排后产生强氢键相互作用。在区域1中,SARS冠状病毒中重要的残基Y484在新型冠状病毒中被替换成了Q498,然而,残基P499作为一个已知的二级结构干扰因素,在新型冠状病毒中有可能导致受体作用区域中区域1的结构重排。再加上Q498和N501,这些残基可能与ACE2中K353和其他残基形成新的氢键。此外,另外一个值得注意的在两种病毒RBD之间的差别是K417(2019-nCoV)/V404(SARS-CoV)。这是一个存在于凹陷的RBD区域之中的残基,在SARS病毒与ACE2的相互作用中,这一残基不产生相互作用。但在新型冠状病毒中,带有正电荷的长链并赖氨酸残基有可能分别与ACE2中的H34和D30两个残基产生强氢键相互作用和盐桥相互作用。虽然分子模拟实验可以用于上述相互作用的研究,在相关的蛋白络合物晶体衍射和冷冻电镜结构被解析之前,其不确定性还不能被完全排除。基于形势的紧迫性,多个研究小组应已在进行这方面的研究。此外,另一研究方向是表达出新型冠状病毒spike蛋白的RBD区域,用生物化学方法解析其与ACE2蛋白的相互作用强度,并与SARS冠状病毒做比较。总之,在相关实验研究结果之前,关于新型冠状病毒spike蛋白与ACE2受体相互作用弱于SARS冠状病毒的断言为时尚早。
SARS病毒、灭活SARS病毒、DNA疫苗和病毒载体疫苗,并已经被成功应用于针对动物SARS冠状病毒的疫苗防护。7,12,13相似的策略也可能可应用于针对新型冠状病毒的疫苗开发。SARS冠状病毒spike蛋白在受体结合和膜融合上的作用使得它成为疫苗研发和抗病毒药物的良好靶点。基于spike蛋白的针对SARS的疫苗研究已有几篇综述总结。7-11研究SARS病毒疫苗的策略有减毒此外,直接将新型冠状病毒2019-nCoV的RBD区域与免疫增强佐剂相结合作为疫苗,直接触发人体针对其RBD区域的抗体合成,以中和新型冠状病毒。14
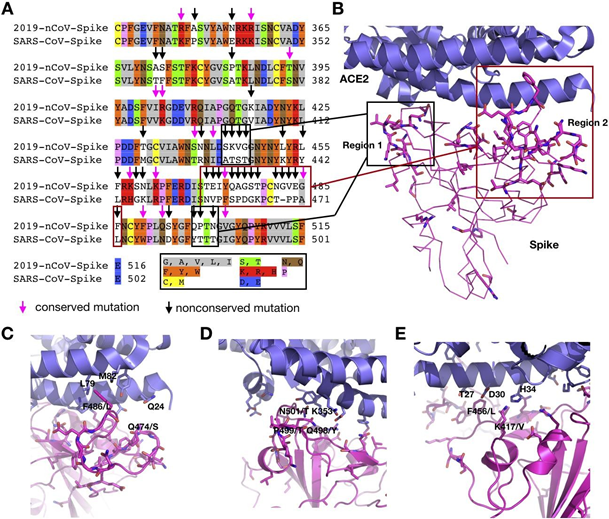
图2. A) 2019-nCoV与SARS-CoV spike蛋白 RBD结构域氨基酸序列比对, 强调了保守和非保守突变。B-E) 2019-nCov spike蛋白(基于PDB entry 2AJF,使用Modeller建立同源模型)与区域1和区域2 ACE2之间的各种结合相互作用。
虽然用于中和SARS冠状病毒spike蛋白的抗体和多肽见于报道,这些抗体和多肽可能并不能很好地用于新冠状病毒的治疗。鉴于前述的讨论,两种病毒spike蛋白RBD区域中直接参与与ACE2蛋白相互作用的两个区域非常不同。针对SARS病毒RBD开发的多肽和抗体应当只能与新型冠状病毒的RBD区域产生弱的相互作用。因此,研究开发能够与新型冠状病毒RBD区域产生强相互作用的新抗体和多肽用于阻断病毒与细胞受体的相互作用尤为关键。包括我们在内的许多研究小组已经发展了构建大环多肽文库的方法,并将它们应用于针对药物靶点的快速筛选方法。15-20将这些多肽库应用于针对新型冠状病毒RBD区域或者两个与ACE2蛋白相互作用的区域可能可快速筛选出针对新型冠状病毒的大环多肽药物。虽然我们已经开始了相关的研究,但是漫长的药物研发过程使得这些成果将很难被应用于现今疫情中的感染者。结合抗击SARS冠状病毒的经验,另外一种替代方法则是直接应用与新型冠状病毒RBD或者ACE2序列相同的多肽。从SARS病毒RBD和ACE2受体中产生的多肽已被发展针对SARS病毒感染的新型治疗方法,这些多肽可以阻断病毒与受体之间的相互作用。例如,与SARS冠状病毒RBD有重合的多肽(氨基酸471-503)可有效阻断ACE2与SARS病毒的结合,抑制SARS病毒进入Vero细胞,EC50 高达41.6 μM。21此外,一结合了两段ACE2序列(氨基酸22-44和351-357),中间以甘氨酸相连的多肽,表现出极强的抗SARS活性,IC50 达0.1 μM。22在任何阻断病毒-受体相互作用的强力药物出现之前,可以尝试使用基于新冠状病毒RBD区域的多肽或者其组合鸡尾酒疗法来进行治疗。
RdRp蛋白
虽然新型冠状病毒与SARS冠状病毒在基因组上只有82%相似性,但两者的RpRd蛋白却有极高的96%相似性(图3A)。RpRd蛋白有一个大而深的凹槽结构区作为RNA合成的活性中心。两种病毒的RpRd蛋白残基上的差异都发生在远离这一活性中心的区域(图3B)。23这两种酶的高序列保守型使得针对SARS病毒RdRp蛋白为靶点发展的高活性抑制剂很可能也能有效抑制新型冠状病毒RdRp酶。

图3 A) 2019-nCoV RdRp与SARS-CoV RdRp之间氨基酸序列比对,强调了保守和非保守突变。B) SARS-CoV RdRp活性位点的晶体结构(PDB词条:6NUS)
虽然没有深入研究,但有几种化合物可以抑制SARS病毒RdRp酶或其RNA复制过程。其中一种有抗病毒活性的是金三羧酸(aurintricarboxylic acid, ATA in图4)。ATA是一种阴离子聚合物,可以与多种蛋白质靶点相互作用,包括HIV-1,HIV-2相关的gp120,且被报道可以阻止SARS病毒复制(EC50 = 0.2 mg / mL)。24-26虽没有直接实验证据,计算模型表明ATA可能与RdRp相互作用。27其他RdRp抑制剂均为核酸类似物,这为阻止病毒RNA复制提供了最有有希望的方法。核酸类似物利巴韦林(ribavirin,RBV)已被测试针对于SARS病毒,以及SARS病毒及MERS病毒感染病人。28-31RBV的有效性不能确定,有些研究甚至报道它加重病人的病情。32nsp14酶的核酸外切酶活性已被证实可以移除错配和嵌入的核酸类似物,使nsp14的核酸外切酶活性灭活被证实可以提高核酸类似物如RBV的有效性。31,33要使核酸类似物有效抑制病毒RNA复制,则该物质必须能逃过核酸内切酶的监测,或其活性超过核酸内切酶。雷姆昔韦(Remdesivir, GS-5734)是第二种情形的绝佳例子。这一带有1’-氰基的苷酸类似物前药表现出针对人呼吸道上皮细胞(HAE)模型和老鼠的非常强抗SARS (IC50 = 0.069 μM) 及抗MERS((IC50 = 0.074 μM)活性。34雷姆昔韦针对多种蝙蝠冠状病毒的有效性也被研究,被认为具有广谱的抗冠状病毒活性。在减弱核酸外切酶活性的细胞系中,冠状病毒对雷姆昔韦的易感性增加。35冠状病毒针对雷姆昔韦的抗性研究则在β-冠状病毒模型和鼠肝炎病毒(murine hepatitis virus, MHV)中进行研究。MHV通过了前体化合物GS-441524(含有6’-羟基而不是磷酰胺)的筛选,在RdRp产生了两个突变,F476L和V553L。这些突变赋予了MHV针对雷姆昔韦5.6倍的抗性,当在SARS病毒中引入一致的突变后,SARS病毒对雷姆昔韦的抗性增加了六倍(0.01 μM vs 0.06 μM)。被有抗性的SARS病毒感染的老鼠在四天后表现显著更低的病毒滴度。35综上,雷姆昔韦具有可以超越nsp12的校正能力,而针对其的耐药突变会减弱病毒的毒力。此策略针对冠状病毒RdRp的药物开发不仅为新型冠状病毒2019-nCoV的治疗提供了基础,也可作为广谱抗病毒方法用于应对将来的冠状病毒爆发。
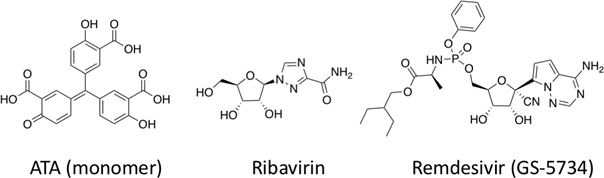
图4. 通过RdRp机制抑制SARS-CoV病毒复制的药物结构
3CLpro和 PLpro蛋白
3CLpro 和PLpro 是两种在复制和包装新一代病毒过程中起作用的蛋白酶,二者可处理从基因组RNA到结构或非结构蛋白的多肽翻译。PLpro 也可作为去泛素酶,其功能是去泛素化宿主细胞蛋白如干扰素因子3(IRF3),以及灭活核因子活化B细胞k轻链增强子vated B细胞(NF- kB)的途径,36而这会使感染病毒的宿主细胞受到免疫抑制。正是因为这两种蛋白酶关乎病毒的复制和控制宿主细胞,对病毒至关重要,使它们成为可行的抗病毒药物的靶点。与RdRp 蛋白类似,2019 - nCoV 和SARS-CoV在3CLpro编码上有96%的显着的序列同一性(图5A)。3CLpro会自动形成二聚体,它的每个单体包含两个区域,即N 末端催化区域和C 末端区域(图5B)。36,37在催化区域上大多数显示两种病毒之间差异的氨基酸残基是在蛋白表面上。虽然S46(2019-nCoV)/ A(SARS- CoV)可能与结合到活性位点的底物或抑制剂有相互作用,但其从A(丙氨酸)到S(丝氨酸)的小的结构变化应当不会显著改变小分子抑制剂与活性位点的结合。能有效抑制SARS-nCoV 3CLpro的小分子抑制剂预计对2019-nCoV 3CLpro具有相似的作用。
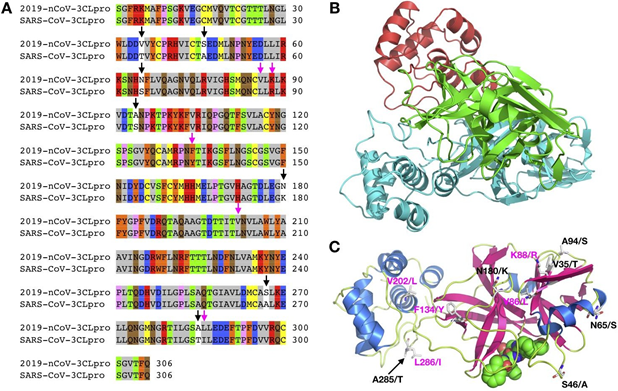
图 5. A) 2019-nCoV 3CLpro与SARS-CoV 3CLpro之间氨基酸序列比对, 强调了保守和非保守突变。B-C)使用基于SARS-CoV 3CLpro结构建模2019-nCoV 3CLpro结构(PDB条目:2A5I)
与3CLpro 不同,来自两种病毒的PLpro 仅共享83%的序列同一性(图6A)。两种病毒之间不同的氨基酸残基几乎覆盖PLpro的所有表面。氨基酸组成的显著性变化可影响两种PLpro 酶与其配体的相互作用。但是,形成活性位点的三个二级结构成分在两个PLpro蛋白中没有变化(图6B)。38因此,针对SARS - CoV PLpro 开发的抑制剂可能也适用于2019-nCoV PLpro 。
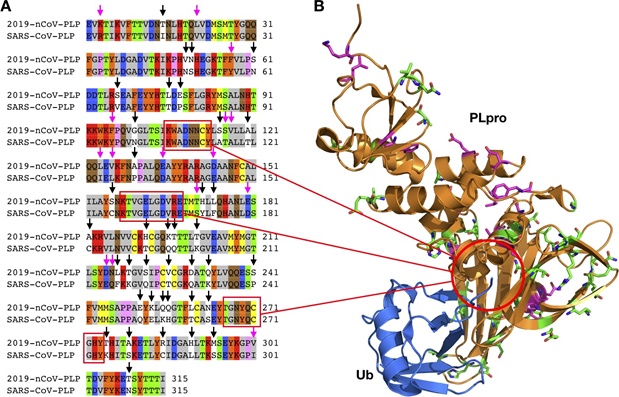
图 6. A) 2019-nCoV PLpro与SARS-CoV PLpro之间氨基酸序列比对, 强调了保守和非保守突变。B) SARS-CoV PLpro的晶体结构(PDB项:4MM3)
在过去的二十年中,对SARS-CoV进行药物治疗的许多研究集中在3CLpro和PLpro的小分子,肽和拟肽抑制剂的开发。就结合和抑制这两种蛋白酶而言,许多抑制剂都在μM水平。39- 44但是,目前已有一些高效的低纳摩范围的抑制剂,它们可以与其他蛋白酶抑制剂疗法结合使用,以帮助对抗病毒。45我们将基于被抑制以阻止病毒控制宿主细胞的蛋白酶的不同将抑制剂分为几类。每种化合物均通过SARS-CoV,MERS-CoV或去泛素化细胞模型进行了测试。目前已经开发了数百种抑制3CLpro和PLpro的小分子化合物,我们选取了几个自2000年代初以来最有效的抑制剂。化合物结构和抑制浓度总结于图7。这些化合物在抑制浓度处于低微摩范围,还有进一步开发的空间。但是,已有广泛的构效关系研究可用于指导靶向2019-nCoV抑制剂研究时需要修饰哪些取代基。此外,该总结还可以指导研究人员和卫生专业人员使用两种或多种这类化合物的联合疗法,在治疗冠状病毒感染的人方面已有联合疗法的先例。其中一个化合物3CLpro-1(图7)对SARS-CoV的IC50值是200 nM。41这种强的抑制作用可能足以对抗2019-nCoV病毒。
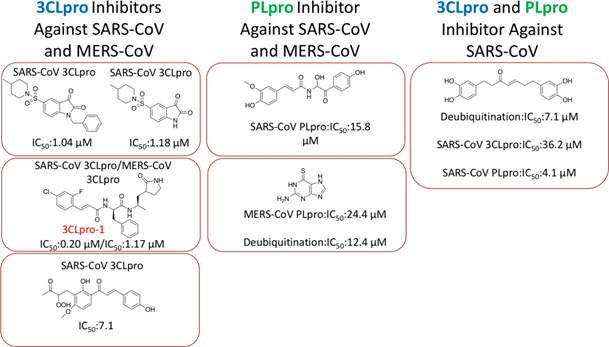
图7. 这是CoV蛋白酶抑制剂的代表,为设计2019- nCoV新型小分子蛋白酶抑制剂提供了执行SAR的支架。3CLpro-1被认为是最有效的抑制剂。
3CLpro和PLpro均为半胱氨酸蛋白酶。可能为其开发出具有高效价的共价抑制剂。最近 Zhou 及其同事开发了一类特异性靶向冠状病毒进入的潜在共价半胱氨酸蛋白酶抑制剂。45虽然与3CLpro和PLpro没有直接关系,但是这类乙烯基砜小分子能够在nM范围内抑制病毒的复制。该小组发现,丝氨酸蛋白酶抑制camostat卡莫司他与其乙烯基砜半胱氨酸蛋白酶抑制剂的组合使用能够抵抗SARS-CoV。与对照组相比,使用联合疗法治疗的患有SARS-CoV小鼠的存活率显著提高。他们研究了乙烯基砜小分子的几种变体,其对应的IC50值如图8所示(化合物A到C)。这类乙烯基砜小分子为药物构效关系研究提供了新的结构骨架。此外,还可以对这类小分子针对3CLpro和PLpro特异性抑制作用进行测试,以进一步阐明其作用机理。考虑到它们对SARS-CoV的高效抑制作用,它们可能对2019-nCoV同样有效。

图8. 乙烯基砜蛋白酶抑制剂,防止CoV进入,并与camostat卡莫司他联合增加小鼠模型的存活率与非典-CoV感染。
了解正冠状病毒亚科的更远的成员也可以为新的治疗方案提供灵感,例如对猫冠状病毒(FCoV)及其突变形式猫传染性腹膜炎病毒(FIPV)的研究。研究发现三肽亚硫酸氢盐加合物GC376和NPI64(图9)均在0.04 μM浓度下对 FIPV复制具有显著抑制作用。46FIPV和SARS-CoV的3CLpro共有大约50%的序列同一性,但总体结构保守。 在基于FRT的活性分析中,发现GC376对SARS-CoV中3CLpro的IC50值是FIPV的4.9倍。结合较高的化合物细胞毒性(在CRFK细胞中GC376和NPI64分别是 CC50> 150 μM和CC50 = 61.91 μM),应尽快研究这些掩蔽醛类弹头(masked-aldehyde warheads)抑制剂对2019-nCoV的3CLpro的作用。

图 9. 三肽亚硫酸氢盐加合物已被证明可以防止猫冠状病毒FIPV的病毒复制。在基于frede的活性试验中,GC376(左)对SARS-CoV 3CLpro产生了类似程度的抑制。
结论
2019-nCoV和SARS-CoV在其RdRp和3CLpro蛋白上具有非常高的序列同源性。先前的研究已经发现了一些基SARS-CoV中这两种蛋白为靶点的小分子抑制剂。我们设想雷姆昔韦和3CLpro-1可直接用于治疗2019-nCoV。由于雷姆昔韦是一种正在进行临床试验的药物,因此中国相关部门可能会与吉利德进行谈判,以决定是否可能将该药物用于2019-nCoV患者。 其他潜在针对新型冠状病毒的小分子抑制剂如图8和9所示。2019-nCoV spike蛋白的RBD与ACE2的结合区域与SARS-CoV spike 蛋白有显著差异,尤其是在与ACE2蛋白受体直接作用的两个区域(图2B:1和2)。这种差异排除了将先前开发作用于SARS-CoV spike蛋白RBD的抗体和治疗性肽用于治疗2019-nCoV的可能。但是,开发RBD和ACE2的同源多肽及其鸡尾酒疗法用于抑制RBD-ACE2相互作用以防止感染可能是一快速治疗方案。
致谢
德克萨斯州 A&M 药物研发中心的研究资金由国家卫生研究院(基金 R01GM121584 和 R01GM127575)、德克萨斯州癌症预防与研究所(基金 RP170797)和 Welch 基金会(基金 A-1715)提供。我们感谢Sunshine Zea Leeuwon在设计所有图形时提供的帮助。
References
1. Zhou, P.; Yang, X.-L.; Wang, X.-G.; Hu, B.; Zhang, L.; Zhang, W.; Si, H.-R.; Zhu, Y.; Li, B.; Huang, C.-L.; Chen, H.-D.; Chen, J.; Luo, Y.; Guo, H.; Jiang, R.-D.; Liu, M.-Q.; Chen, Y.; Shen, X.- R.; Wang, X.; Zheng, X.-S.; Zhao, K.; Chen, Q.-J.; Deng, F.; Liu, L.-L.; Yan, B.; Zhan, F.-X.; Wang, Y.-Y.; Xiao, G.; Shi, Z.-L., Discovery of a novel coronavirus associated with the recent pneumonia outbreak in humans and its potential bat origin. bioRxiv 2020, 2020.01.22.914952.
2. Li, F.; Li, W.; Farzan, M.; Harrison, S. C., Structure of SARS coronavirus spike receptor- binding domain complexed with receptor. Science 2005, 309 (5742), 1864-8.
3. Dong, N.; Yang, X.; Ye, L.; Chen, K.; Chan, E. W.-C.; Yang, M.; Chen, S., Genomic and protein structure modelling analysis depicts the origin and infectivity of 2019-nCoV, a new coronavirus which caused a pneumonia outbreak in Wuhan, China. bioRxiv 2020, 2020.01.20.913368.
4. Baranov, P. V.; Henderson, C. M.; Anderson, C. B.; Gesteland, R. F.; Atkins, J. F.; Howard,
M. T., Programmed ribosomal frameshifting in decoding the SARS-CoV genome. Virology 2005,
332 (2), 498-510.
5. Ziebuhr, J.; Snijder, E. J.; Gorbalenya, A. E., Virus-encoded proteinases and proteolytic processing in the Nidovirales. Journal of General Virology 2000, 81 (4), 853-879.
6. Xu, X.; Liu, Y.; Weiss, S.; Arnold, E.; Sarafianos, S. G.; Ding, J., Molecular model of SARS coronavirus polymerase: implications for biochemical functions and drug design. Nucleic Acids Research 2003, 31 (24), 7117-7130.
7. Cavanagh, D., Severe acute respiratory syndrome vaccine development: experiences of vaccination against avian infectious bronchitis coronavirus. Avian Pathol 2003, 32 (6), 567-82.
8. Du, L.; He, Y.; Zhou, Y.; Liu, S.; Zheng, B. J.; Jiang, S., The spike protein of SARS-CoV--a target for vaccine and therapeutic development. Nat Rev Microbiol 2009, 7 (3), 226-36.
9. Jiang, S.; He, Y.; Liu, S., SARS vaccine development. Emerg Infect Dis 2005, 11 (7), 1016- 1020.
10. Taylor, D. R., Obstacles and advances in SARS vaccine development. Vaccine 2006, 24 (7), 863-871.
11. Roper, R. L.; Rehm, K. E., SARS vaccines: where are we? Expert Rev Vaccines 2009, 8 (7), 887-898.
12. Holmes, K. V., SARS coronavirus: a new challenge for prevention and therapy. J Clin Invest 2003, 111 (11), 1605-9.
13. Navas-Martin, S. R.; Weiss, S., Coronavirus replication and pathogenesis: Implications for the recent outbreak of severe acute respiratory syndrome (SARS), and the challenge for vaccine development. J Neurovirol 2004, 10 (2), 75-85.
14. He, Y.; Zhou, Y.; Liu, S.; Kou, Z.; Li, W.; Farzan, M.; Jiang, S., Receptor-binding domain of SARS-CoV spike protein induces highly potent neutralizing antibodies: implication for developing subunit vaccine. Biochem Biophys Res Commun 2004, 324 (2), 773-81.
15. O'Neil, K. T.; Hoess, R. H.; Jackson, S. A.; Ramachandran, N. S.; Mousa, S. A.; DeGrado,
W. F., Identification of novel peptide antagonists for GPIIb/IIIa from a conformationally constrained phage peptide library. Proteins: Structure, Function, and Bioinformatics 1992, 14 (4), 509-515.
16. McLafferty, M. A.; Kent, R. B.; Ladner, R. C.; Markland, W., M13 bacteriophage displaying disulfide-constrained microproteins. Gene 1993, 128 (1), 29-36.
17. Hipolito, C. J.; Suga, H., Ribosomal production and in vitro selection of natural product- like peptidomimetics: The FIT and RaPID systems. Current Opinion in Chemical Biology 2012, 16 (1), 196-203.
18. Palei, S.; Becher, K. S.; Nienberg, C.; Jose, J.; Mootz, H. D., Bacterial Cell-Surface Display of Semisynthetic Cyclic Peptides. Chembiochem 2019, 20 (1), 72-77.
19. Xiao, W.; Wang, Y.; Lau, E. Y.; Luo, J.; Yao, N.; Shi, C.; Meza, L.; Tseng, H.; Maeda, Y.; Kumaresan, P.; Liu, R.; Lightstone, F. C.; Takada, Y.; Lam, K. S., The Use of One-Bead One- Compound Combinatorial Library Technology to Discover High-Affinity αvβ3 Integrin and Cancer Targeting Arginine-Glycine-Aspartic Acid Ligands with a Built-in Handle. Molecular Cancer Therapeutics 2010, 9 (10), 2714-2723.
20. Wang, X. S.; Chen, P.-H. C.; Hampton, J. T.; Tharp, J. M.; Reed, C. A.; Das, S. K.; Wang, D.-S.; Hayatshahi, H. S.; Shen, Y.; Liu, J.; Liu, W. R., A Genetically Encoded, Phage-Displayed Cyclic-Peptide Library. Angewandte Chemie International Edition 2019, 58 (44), 15904-15909.
21. Hu, H.; Li, L.; Kao, R. Y.; Kou, B.; Wang, Z.; Zhang, L.; Zhang, H.; Hao, Z.; Tsui, W. H.; Ni, A.; Cui, L.; Fan, B.; Guo, F.; Rao, S.; Jiang, C.; Li, Q.; Sun, M.; He, W.; Liu, G., Screening and Identification of Linear B-Cell Epitopes and Entry-Blocking Peptide of Severe Acute Respiratory Syndrome (SARS)-Associated Coronavirus Using Synthetic Overlapping Peptide Library. Journal of Combinatorial Chemistry 2005, 7 (5), 648-656.
22. Han, D. P.; Penn-Nicholson, A.; Cho, M. W., Identification of critical determinants on ACE2 for SARS-CoV entry and development of a potent entry inhibitor. Virology 2006, 350 (1), 15-25.
23. Kirchdoerfer, R. N.; Ward, A. B., Structure of the SARS-CoV nsp12 polymerase bound to nsp7 and nsp8 co-factors. Nat Commun 2019, 10 (1), 2342.
24. González, R. G.; Blackburn, B. J.; Schleich, T., Fractionation and structural elucidation of the active components of aurintricarboxylic acid, a potent inhibitor of protein nucleic acid interactions. Biochimica et Biophysica Acta (BBA) - Nucleic Acids and Protein Synthesis 1979, 562 (3), 534-545.
25. Cushman, M.; Wang, P.; Chang, S. H.; Wild, C.; De Clercq, E.; Schols, D.; Goldman, M. E.; Bowen, J. A., Preparation and anti-HIV activities of aurintricarboxylic acid fractions and analogs: direct correlation of antiviral potency with molecular weight. Journal of Medicinal Chemistry 1991, 34 (1), 329-337.
26. He, R.; Adonov, A.; Traykova-Adonova, M.; Cao, J.; Cutts, T.; Grudesky, E.; Deschambaul, Y.; Berry, J.; Drebot, M.; Li, X., Potent and selective inhibition of SARS coronavirus replication by aurintricarboxylic acid. Biochemical and Biophysical Research Communications 2004, 320 (4), 1199-1203.
27. Yap, Y.; Zhang, X.; Andonov, A.; He, R., Structural analysis of inhibition mechanisms of Aurintricarboxylic Acid on SARS-CoV polymerase and other proteins. Computational Biology and Chemistry 2005, 29 (3), 212-219.
28. Chiou, H. E.; Liu, C. L.; Buttrey, M. J.; Kuo, H. P.; Liu, H. W.; Kuo, H. T.; Lu, Y. T., Adverse effects of ribavirin and outcome in severe acute respiratory syndrome: experience in two medical centers. Chest 2005, 128 (1), 263-72.
29. Muller, M. P.; Dresser, L.; Raboud, J.; McGeer, A.; Rea, E.; Richardson, S. E.; Mazzulli, T.; Loeb, M.; Louie, M.; Canadian, S. R. N., Adverse events associated with high-dose ribavirin: evidence from the Toronto outbreak of severe acute respiratory syndrome. Pharmacotherapy 2007, 27 (4), 494-503.
30. Al-Tawfiq, J. A.; Momattin, H.; Dib, J.; Memish, Z. A., Ribavirin and interferon therapy in patients infected with the Middle East respiratory syndrome coronavirus: an observational study. International Journal of Infectious Diseases 2014, 20, 42-46.
31. Smith, E. C.; Blanc, H.; Vignuzzi, M.; Denison, M. R., Coronaviruses Lacking Exoribonuclease Activity Are Susceptible to Lethal Mutagenesis: Evidence for Proofreading and Potential Therapeutics. Plos Pathog 2013, 9 (8).
32. Stockman, L. J.; Bellamy, R.; Garner, P., SARS: systematic review of treatment effects.
PLoS Med 2006, 3 (9), e343.
33. Ferron, F.; Subissi, L.; De Morais, A. T. S.; Le, N. T. T.; Sevajol, M.; Gluais, L.; Decroly, E.; Vonrhein, C.; Bricogne, G.; Canard, B.; Imbert, I., Structural and molecular basis of mismatch correction and ribavirin excision from coronavirus RNA. P Natl Acad Sci USA 2018, 115 (2), E162- E171.
34. Sheahan, T. P.; Sims, A. C.; Graham, R. L.; Menachery, V. D.; Gralinski, L. E.; Case, J. B.; Leist, S. R.; Pyrc, K.; Feng, J. Y.; Trantcheva, I.; Bannister, R.; Park, Y.; Babusis, D.; Clarke, M. O.; Mackman, R. L.; Spahn, J. E.; Palmiotti, C. A.; Siegel, D.; Ray, A. S.; Cihlar, T.; Jordan, R.; Denison, M. R.; Baric, R. S., Broad-spectrum antiviral GS-5734 inhibits both epidemic and zoonotic coronaviruses. Science Translational Medicine 2017, 9 (396), eaal3653.
35. Agostini, M. L.; Andres, E. L.; Sims, A. C.; Graham, R. L.; Sheahan, T. P.; Lu, X.; Smith, E. C.; Case, J. B.; Feng, J. Y.; Jordan, R.; Ray, A. S.; Cihlar, T.; Siegel, D.; Mackman, R. L.; Clarke, M. O.; Baric, R. S.; Denison, M. R., Coronavirus Susceptibility to the Antiviral Remdesivir (GS-5734) Is Mediated by the Viral Polymerase and the Proofreading Exoribonuclease. mBio 2018, 9 (2), e00221-18.
36. Báez-Santos, Y. M.; St. John, S. E.; Mesecar, A. D., The SARS-coronavirus papain-like protease: Structure, function and inhibition by designed antiviral compounds. Antiviral Research 2015, 115, 21-38.
37. Lee, T. W.; Cherney, M. M.; Huitema, C.; Liu, J.; James, K. E.; Powers, J. C.; Eltis, L. D.; James, M. N., Crystal structures of the main peptidase from the SARS coronavirus inhibited by a substrate-like aza-peptide epoxide. J Mol Biol 2005, 353 (5), 1137-51.
38. Ratia, K.; Kilianski, A.; Baez-Santos, Y. M.; Baker, S. C.; Mesecar, A., Structural Basis for the Ubiquitin-Linkage Specificity and deISGylating activity of SARS-CoV papain-like protease. Plos Pathog 2014, 10 (5), e1004113.
39. Chen, X.; Chou, C.-Y.; Chang, G.-G., Thiopurine Analogue Inhibitors of Severe Acute Respiratory Syndrome-Coronavirus Papain-Like Protease, a Deubiquitinating and deISGylating Enzyme. Antiviral Chemistry and Chemotherapy 2009, 19 (4), 151-156.
40. Cheng, K.-W.; Cheng, S.-C.; Chen, W.-Y.; Lin, M.-H.; Chuang, S.-J.; Cheng, I. H.; Sun, C.- Y.; Chou, C.-Y., Thiopurine analogs and mycophenolic acid synergistically inhibit the papain-like protease of Middle East respiratory syndrome coronavirus. Antiviral Research 2015, 115, 9-16.
41. Kumar, V.; Shin, J. S.; Shie, J.-J.; Ku, K. B.; Kim, C.; Go, Y. Y.; Huang, K.-F.; Kim, M.; Liang, P.-H., Identification and evaluation of potent Middle East respiratory syndrome coronavirus (MERS-CoV) 3CLPro inhibitors. Antiviral Research 2017, 141, 101-106.
42. Liu, W.; Zhu, H.-M.; Niu, G.-J.; Shi, E.-Z.; Chen, J.; Sun, B.; Chen, W.-Q.; Zhou, H.-G.; Yang, C., Synthesis, modification and docking studies of 5-sulfonyl isatin derivatives as SARS-CoV 3C-like protease inhibitors. Bioorganic & Medicinal Chemistry 2014, 22 (1), 292-302.
43. Park, J.-Y.; Jeong, H. J.; Kim, J. H.; Kim, Y. M.; Park, S.-J.; Kim, D.; Park, K. H.; Lee, W. S.; Ryu, Y. B., Diarylheptanoids from Alnus japonica Inhibit Papain-Like Protease of Severe Acute Respiratory Syndrome Coronavirus. Biological and Pharmaceutical Bulletin 2012, 35 (11), 2036-2042.
44. Park, J.-Y.; Ko, J.-A.; Kim, D. W.; Kim, Y. M.; Kwon, H.-J.; Jeong, H. J.; Kim, C. Y.; Park, K. H.; Lee, W. S.; Ryu, Y. B., Chalcones isolated from Angelica keiskei inhibit cysteine proteases of SARS-CoV. J Enzym Inhib Med Ch 2016, 31 (1), 23-30.
45. Zhou, Y.; Vedantham, P.; Lu, K.; Agudelo, J.; Carrion, R., Jr.; Nunneley, J. W.; Barnard, D.; Pohlmann, S.; McKerrow, J. H.; Renslo, A. R.; Simmons, G., Protease inhibitors targeting coronavirus and filovirus entry. Antiviral Res 2015, 116, 76-84.
46. Kim, Y.; Shivanna, V.; Narayanan, S.; Prior, A. M.; Weerasekara, S.; Hua, D. H.; Kankanamalage, A. C. G.; Groutas, W. C.; Chang, K.-O., Broad-Spectrum Inhibitors against 3C-Like Proteases of Feline Coronaviruses and Feline Caliciviruses. Journal of Virology 2015, 89 (9), 4942- 4950.
47. Song, Y. H.; Kim, D. W.; Curtis-Long, M. J.; Yuk, H. J.; Wang, Y.; Zhuang, N.; Lee, K. H.; Jeon, K. S.; Park, K. H., Papain-Like Protease (PLpro) Inhibitory Effects of Cinnamic Amides from Tribulus terrestris Fruits. Biological and Pharmaceutical Bulletin 2014, 37 (6), 1021-1028.
本文作者:Jared S. Morse, Tyler Lalonde, 许世卿, 刘文设 美国德州A&M大学化学系药物研发实验室
Translated by Daniel, Edit by Xu
如需转载请联系美捷登www.medajden.com
描二维码关注“美捷登”微信

https://blog.sciencenet.cn/blog-475824-1216268.html
上一篇:中医药方向的研究,可以投稿这些杂志(一)
下一篇:关于新型冠状病毒已经发了多少篇论文?Nature告诉你