博文
使用SnapGene viewer绘制比较基因簇结构图
|
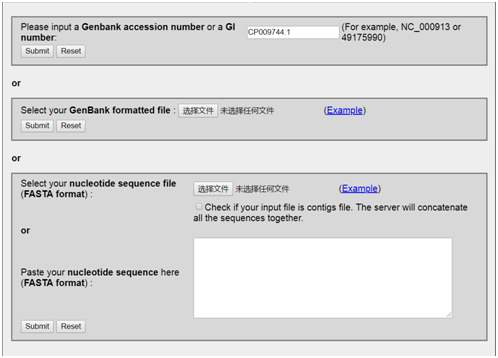
前期介绍了如何使用SnapGene viewer寻找酶切位点和设计引物,SnapGene viewer是一款专业的质粒图谱绘制软件,也可以绘制比较基因簇结构图。今天以基因组里的原噬菌体为例,在这里教大家使用SnapGene viewer绘制比较基因簇结构图,想要学习的小伙伴们,可以跟着学习哦!
1 寻找原噬菌体
1.2 选择prophage
选取PHAGE_Shigel_SfII

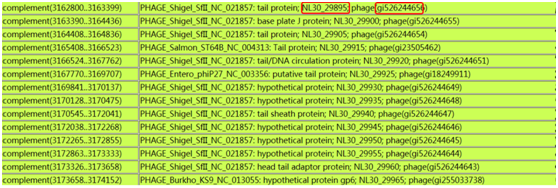
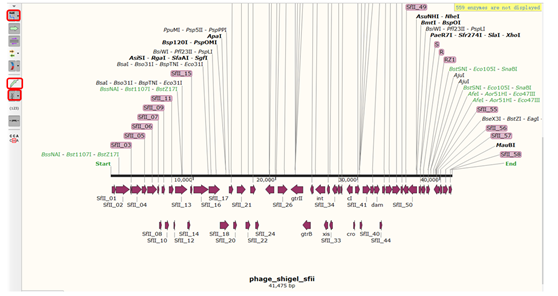
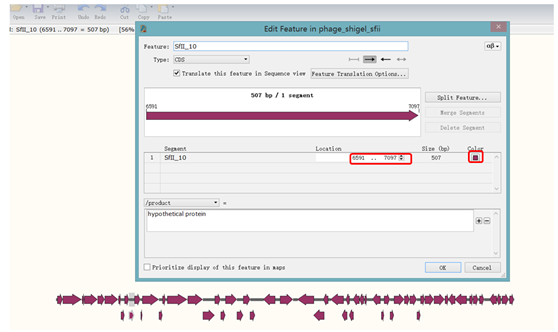
打开Adobe Illustrator CS6,选中矢量图,放置到一个模板里,小编这里就只加了标尺与名称,根据需要你们可以自行添加标注与注释等信息。
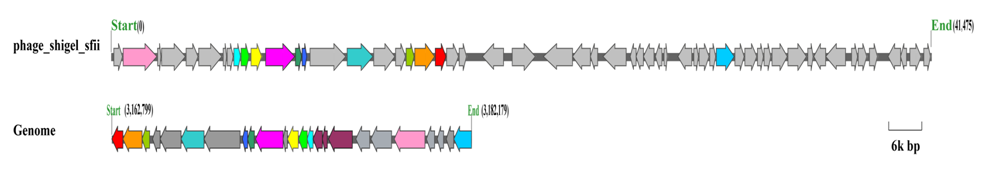
这项绘图的小技巧学会了吗?
更多信息请关注下方公众号

https://blog.sciencenet.cn/blog-3445347-1249949.html
上一篇:Fastaq——fasta文件转化为fastq文件
下一篇:一文学会质粒图谱绘制软件——SnapGene viewer
扫一扫,分享此博文