博文
14篇Nature论文合集!“DNA元件百科全书”公布第三阶段成果
||

“DNA元件百科全书”(ENCyclopedia of DNA Elements,ENCODE)国际合作计划于2003年启动,合作团队由美国国立人类基因组研究所(NHGRI)组织成立,致力于绘制人类与小鼠基因组功能性元件的综合图谱。 北京时间2020年7月29日晚23时,ENCODE计划发布第三阶段成果,公布了超过120万个人类与小鼠体内调控基因的候选功能性元件。 《自然》、《自然—方法》和《自然—通讯》联合发表14篇论文描述了这一结果,为基因组组织和功能带来了新的认知。
在《自然》发表的一篇概述性文章中,美国麻省大学医学院教授翁志萍和同事描述了在ENCODE前两个阶段上扩展增加的近6000项新实验(4834项涉及人类样本,1158项涉及小鼠样本)。
之前几个阶段的大部分研究使用的都是模型细胞系,而第三阶段包含了超过1369个生物样本来源的503种细胞或组织类型。
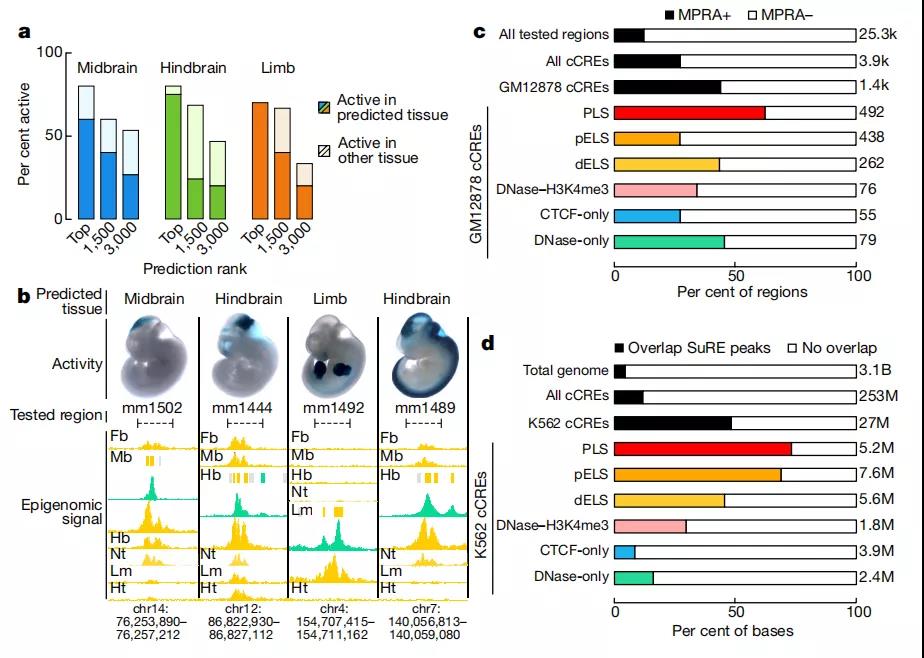
根据ENCODE已经绘制出的数百万个元件,研究人员还建立了一个在线注册库,里面包含了926535个人类的和339815个小鼠的候选顺式调节元件(调节基因转录的非编码DNA区域),覆盖到各自基因组的7.9%和3.4%。
合集中的其他论文利用ENCODE的数据集揭示了决定部分功能性元件作用方式的原理。
例如,美国斯坦福大学教授Michael Snyder和同事绘制了24种人类细胞类型内染色质(DNA与蛋白的复合物)的相互作用,发现不同细胞间的染色质环状结构差异会影响基因表达。
研究人员还通过小鼠研究了顺式调节元件在产前哺乳动物发育中的调节作用。《自然》发表的三篇论文报道了胎鼠在八大发育阶段的具体信息。
其中两篇论文里,美国加州大学圣地亚哥分校教授任兵和同事以及美国索尔克生物研究所教授Joseph Ecker和同事表明,与小鼠发育期间特定组织中有活性的部分增强子元件(能增强基因转录的调节DNA序列)同源的人类元件,存在疾病相关性遗传突变的富集。
这些结果或为探索与人类发育障碍相关的调节元件打下了基础。
《自然》同时发表的一篇的“观点”文章中,Michael Snyder和同事指出,控制基因组调控和功能的元件密集编码在人类基因组中。
不过,虽然发现了大量这类元件,许多影响特定细胞类型或状态的元件依然有待鉴定。
在ENCODE的第四阶段,大量工作将用来拓展所分析的细胞类型和组织。
附:14篇论文标题及链接 1,Expanded encyclopaedias of DNA elements in the human and mouse genomes https://www.nature.com/articles/s41586-020-2493-4 2,Perspectives on ENCODE https://www.nature.com/articles/s41586-020-2449-8 3,A large-scale binding and functional map of human RNA-binding proteins https://www.nature.com/articles/s41586-020-2077-3 4,Landscape of cohesin-mediated chromatin loops in the human genome https://www.nature.com/articles/s41586-020-2151-x 5,Occupancy maps of 208 chromatinassociated proteins in one human cell type https://www.nature.com/articles/s41586-020-2023-4 6,An atlas of dynamic chromatin landscapes in mouse fetal development https://www.nature.com/articles/s41586-020-2093-3 7,Spatiotemporal DNA methylome dynamics of the developing mouse fetus https://www.nature.com/articles/s41586-020-2119-x 8,The changing mouse embryo transcriptome at whole tissue and single-cell resolution https://www.nature.com/articles/s41586-020-2536-x 9,Global reference mapping of human transcription factor footprints https://www.nature.com/articles/s41586-020-2528-x 10,Index and biological spectrum of human DNase I hypersensitive sites https://www.nature.com/articles/s41586-020-2559-3 11,An integrative ENCODE resource for cancer genomics https://www.nature.com/articles/s41467-020-14743-w 12,Transcriptional activity and strain-specific history of mouse pseudogenes https://www.nature.com/articles/s41467-020-17157-w 13,Detecting sample swaps in diverse NGS data types using linkage disequilibrium https://www.nature.com/articles/s41467-020-17453-5 14,Supervised enhancer prediction with epigenetic-pattern recognition and targeted validation https://www.nature.com/articles/s41592-020-0907-8
https://blog.sciencenet.cn/blog-3423233-1244195.html
上一篇:钱书兵课题组阐明5’UTR在mRNA翻译以及降解中的作用
下一篇:佘群新/Guillermo Montoya团队合作解析冰岛硫化叶菌III-B CRISPR-Cas复合物的三维结构