博文
【工具篇之四】Scaffold 软件中Protein View功能崭露头角
||

蛋白视图
此视图窗一共3个不同的相关联窗格构成,包括选取感兴趣蛋白窗口、人工截取谱图窗口和鉴定肽段、以及查看蛋白覆盖率和其他的质谱实验相关参数等。如图1所示
不同的窗口大致如下:
蛋白窗口,它提供了两个相互不同的定位目标蛋白和显示所选择蛋白特性的窗口。
肽段窗口,显示与所选目的蛋白的特征肽段相关参数和信息
蛋白质序列窗口,通过不同的选项卡提供有关的详细信息,所选的肽段二级谱图等。
过滤蛋白视窗中蛋白质和肽段的功能,对蛋白和肽段可信阈值的设置。
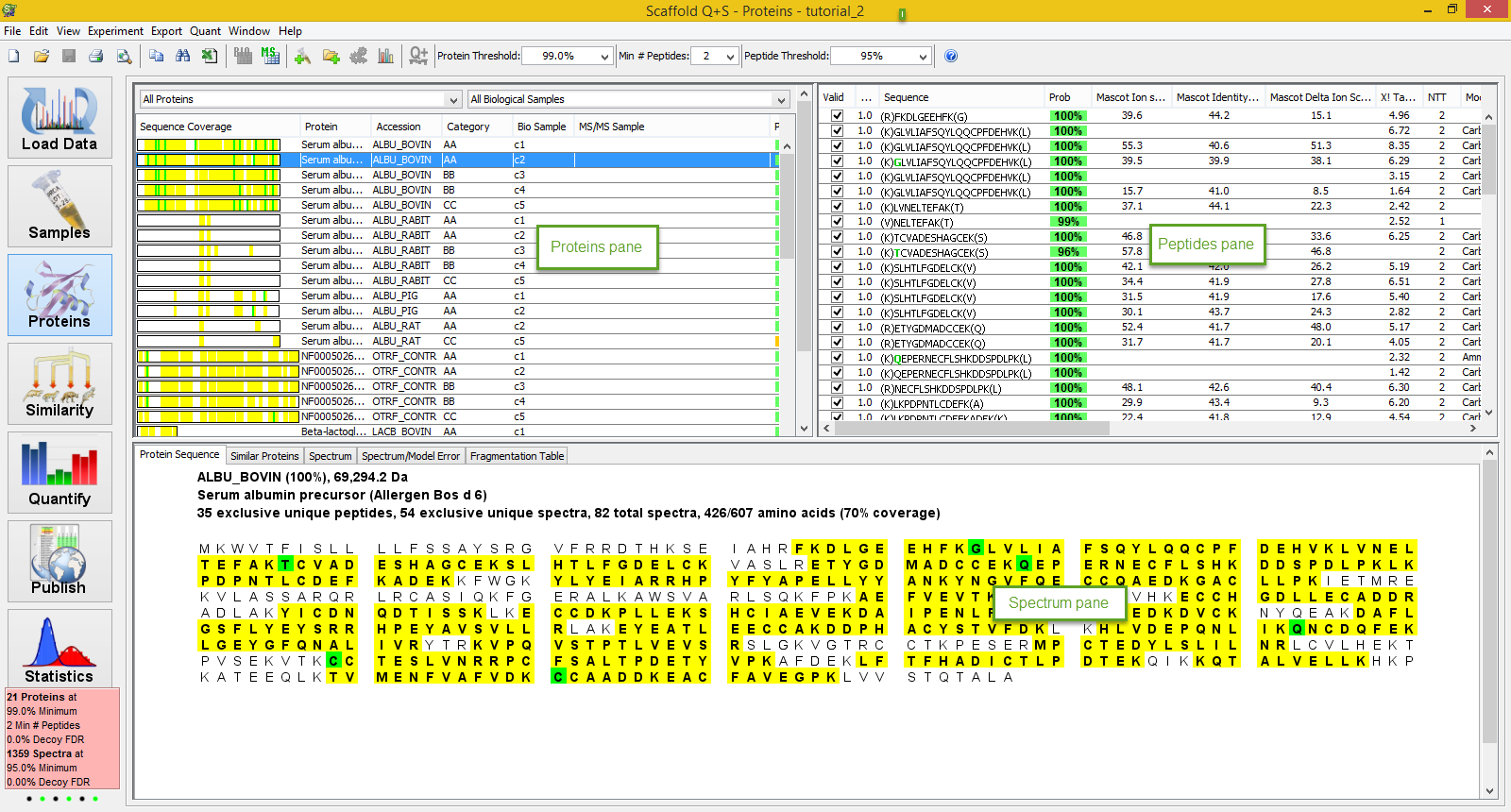
图1
蛋白窗口
蛋白质窗口位于蛋白视窗的左上角,它包括两个下拉列表和表格。如图2所示
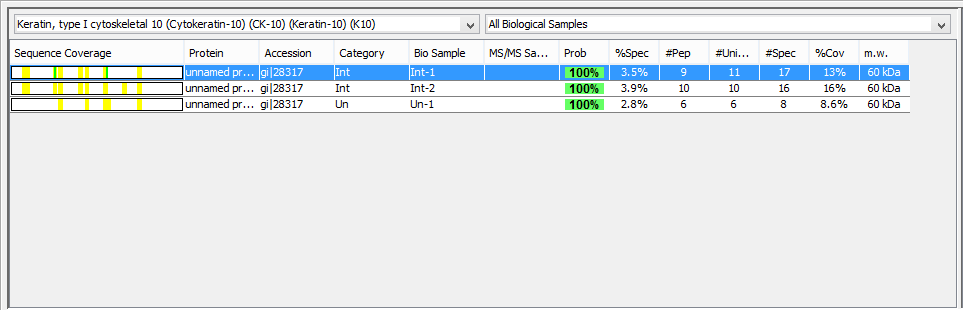
图2
1.序列覆盖率
对于选定的蛋白质,每一行都展示其蛋白的相对应信息都是根据表格中所选的参数而定,一些栏中提供蛋白的信息描述,主要包括下面信息:
2.蛋白描述信息
登录号—蛋白登录号
生物样本选项—来源于生物样本分类时候出现
质谱样本选项—来源于质谱样本分类时候出现
分子量—蛋白质分子量
Pro数值—表示蛋白可信度
%Spec—所有谱图的蛋白百分数
#Pep—独有肽段的数目
#Unique—独有的唯一谱图计数
#Spec—此变量是显示肽段的行数
%Cov—所有识别到的氨基酸或序列的覆盖率表分数
3.左上方下拉列表
它可以过滤表格,使每一行显示的信息中每个生物组合质谱组样本中只有一个选定的蛋白,或者展示所有的蛋白质,下拉选项中包括“所有蛋白质”和出现在样本表汇总的蛋白质组合蛋白簇列表。这意味着在列表中发现的蛋白质可以根据过滤器和阈值发生变化,请详见【工具篇】中的筛选窗口。
4.右上角下拉列表
根据当前选择的摘要级别,它根据生物实验组或者质谱实验组进行筛选。
多肽窗口
多肽窗口定位在蛋白视图的右上方,包括谱图表格。这个表格是显示一系列所鉴定到的肽段的谱图。如图3所示是蛋白视图中的多肽窗口。
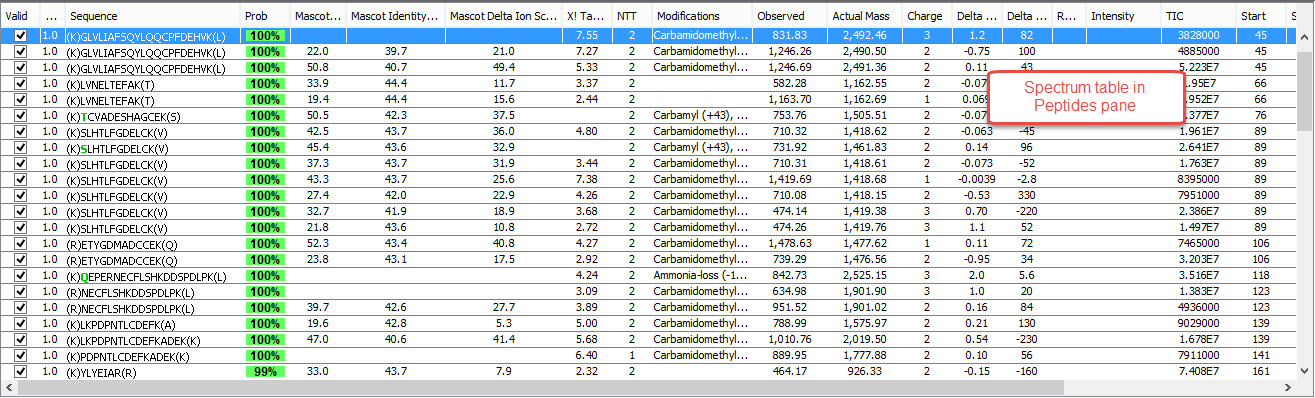
图3
1.谱图表格
表格的每一行都表示一个图谱,而每一列提供类似于它所识别的肽段序列,其检索分数信息等参数均旨在帮助用户手动验证每一个图谱。
谱图表格栏:
有效性——第一列是肽段有消息的选项,此选项是为了检查肽段谱图是否被算入蛋白的可信肽段,当导入数据时,被鉴定的肽段已经按照指定的算法进行打分,Scaffold软件自动定义谱图是否有效,如果人工去检索一个图谱,用户能够根据自己的判断去进行肽段有效性评价,通过对不同的肽段可信度对肽段进行可信度的筛选。
肽段序列——肽序列经过质谱鉴定,肽段中的氨基酸序列中前缀和后面括号表示肽段的前后氨基酸残基,在整个蛋白质序列中,这给用户提供了额外的信息,比如确认肽是色氨酸还是半色氨酸,基于它是在K或R之后开始的。
可能性——Scaffold软件提供的可能性判断是在上传数据的时候就已经被赋予了。肽段的可信度是带有不同着色的,具体参考可信度图例。
搜索引擎打分—会在蛋白列表中有一系列的打分数值,Scaffold软件将不同搜索引擎分析的打分汇总到了一起,下面是常见的不同的打分方式:
SQUEST—Xcorr 和 DeltaCn
Mascot—Ion score、Identity score和Delta Ion Score
X!Tandem—log of expectation score
NTT—胰酶酶切后的末端数目
修饰—所鉴定肽段发生的修饰类型和位点
质荷测量值:
Observed-母离子的质荷比
Actual Mass-质荷比乘以电荷数是实际相对质量
Charge—肽段带点数
Delta Da—偏移的相对分子质量
Delta PPM—母离子误差值
保留时间—以秒为单位,在原始上传数据中得到。
丰度—肽段母离子丰度
TIC—总二级质谱离子电流
Start—肽段开始索引
Stop—肽段结束索引
2.肽段质量的计算方法
谱图中提供四种不同的质量数值,分别是:
•Observed m/z
• ActualPeptide Mass (AMU)
•Calculated +1H Peptide Mass (AMU)
• Actualminus Calculated peptide mass (AMU)
• Actualminus calculated peptide mass (PPM)
AMU表示质谱仪器所检测的肽段理论值,通过Observed(M/Z)计算得到如下公式:
Actual Peptide Mass (Da)=Observed (M/Z) × Spectrum Charge -1proton Spectrum Charge
理论多肽质量是通过将所有氨基酸残基加和起来所得再加一分子的水:
Theoretical Peptide Mass(Da)= ΣAminoAcid Residue Mass + 1water
图谱中提供理论的肽段质量+1个质子的质量在“Calculated+1H Peptide Mass”这一栏下面。
Delta Mass (Da)=Actual Peptide Mass - Theoretical Peptide Mass(Da)=(Actual Peptide Mass)– ( Calculated + 1H Peptide Mass - 1Proton)
Delta Mass in PPM的计算方法如下:
Delta Mass(PPM)= Delta Mass(Da) 106/ Actual PeptideMass(Da)
蛋白序列窗口
蛋白序列窗口提供
蛋白序列选项卡
相似蛋白选项卡
二级谱图选项卡
误差选项卡
碎裂表格选项卡
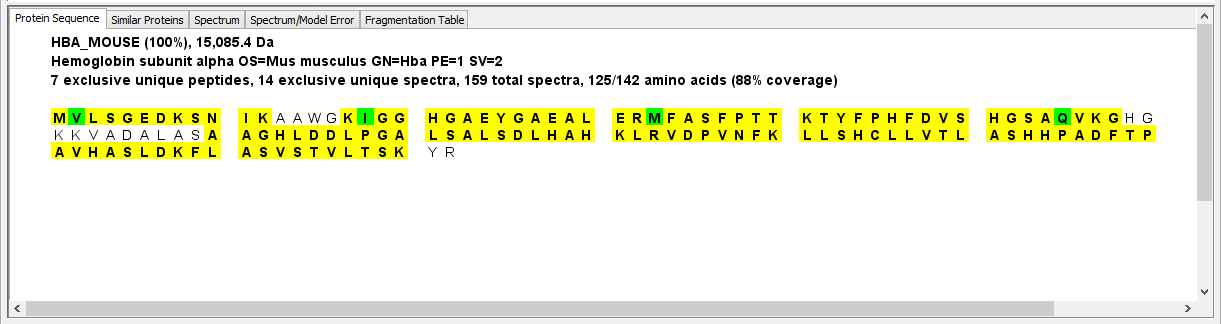
图4
1.蛋白序列选项卡:
如图4所示,选项卡中显示的整个蛋白的氨基酸序列,然后黄色表示所鉴定到的氨基酸序列,然而绿色是发生修饰的氨基酸。
2.谱图选项卡:
如下图所示,详细可参考scaffold1介绍: 详细介绍
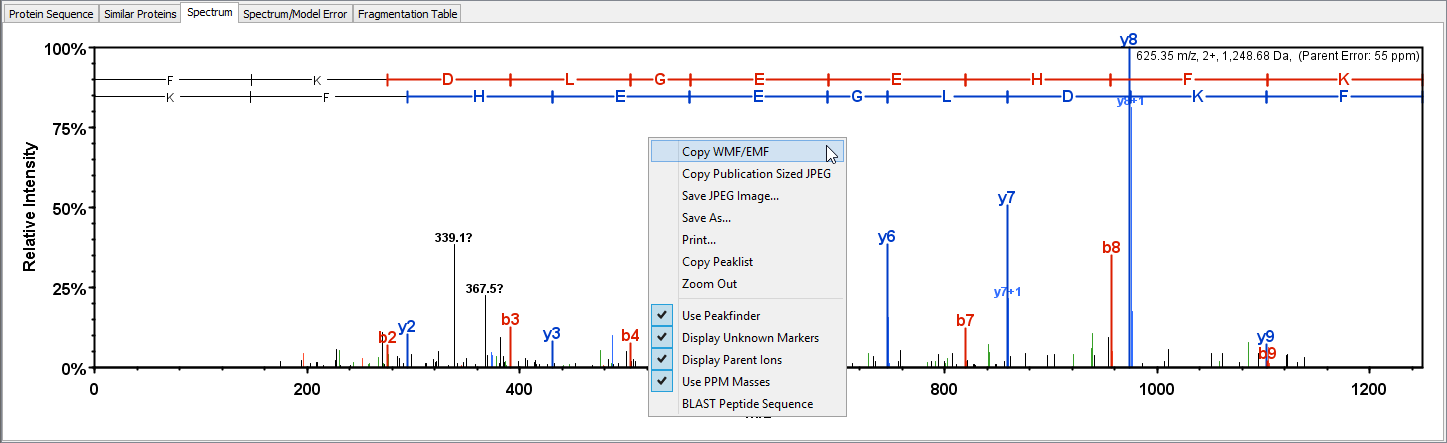
3.误差选项卡
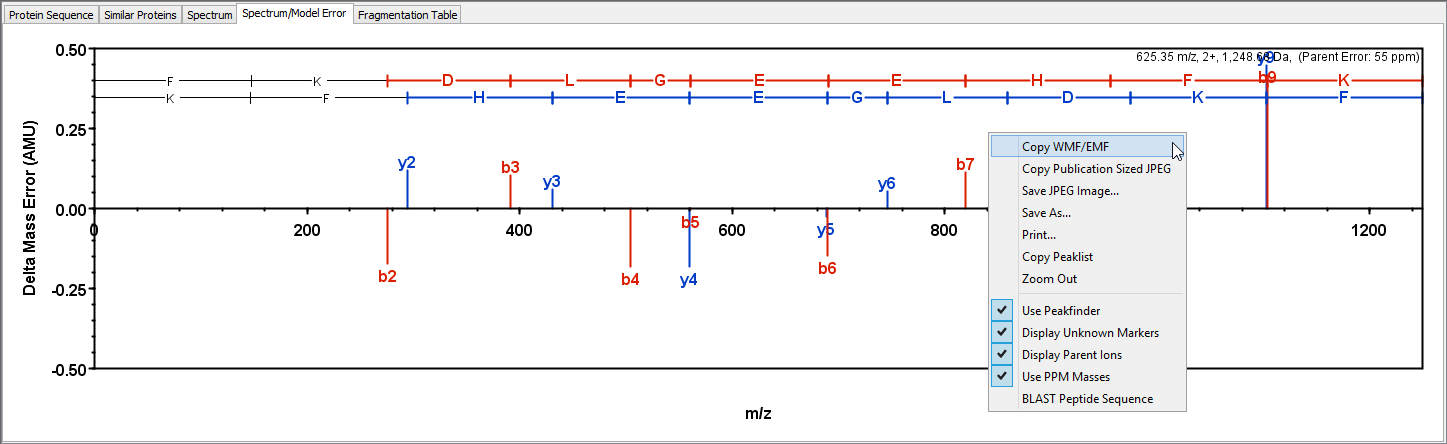
此谱图误差选项卡显示的是误差图表,其定位为所选的肽段的峰-测量片段峰。
纵坐标是AMU数值,所显示在样本加载时候设置为质量精确下的最大值。
4.碎裂信息选项卡
此选项卡所显示的信息是与所选肽段谱图匹配的多肽碎片离子的碎裂信息,表上和峰上都能判断此峰图哪个对应的离子以及其离子信息,肽段碎裂表格与谱图是互补的,表格展示的是碎裂离子的阶梯信息,而谱图能够显示其对应的强度,未匹配的峰以及标记的固有离子信息。

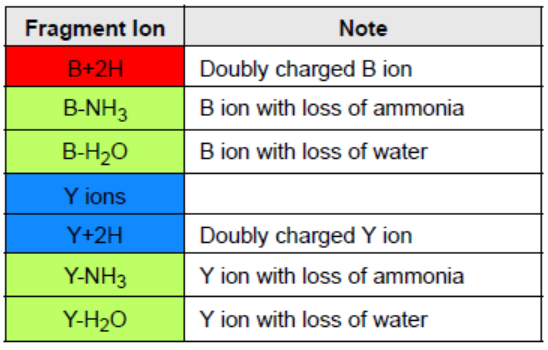
带颜色的表格是对应谱图上的多肽碎裂离子
白色的表格则是预测的多肽片段,但是谱图中并未检测到
黑色的表格则是非化学产生的多肽离子。
AIMS---蛋白质谱服务商,提供快速便捷的 LC-MS/MS 蛋白定性/定量检测。
https://blog.sciencenet.cn/blog-3402731-1175118.html
上一篇:【工具篇之三】Scaffold 软件中Sample View功能小试牛刀
下一篇:【方法篇】不同处理方法对质谱分析互作蛋白的影响